一、 MultiQC介绍
FastQC、Qualimap 和RSeQC等不少生信工具都可以给样品生成一个评估结果,但是几乎所有的质控工具都是针对单个样本生成一个报告
MultiQC,基于Python的小工具能把多个质控结果整合,其强大的功能主要体现在以下三个方面:
1)能将测序数据的多个QC结果整合成一个HTLM网页交互式报告,同时也能导出pdf文件;
2)支持多种分析类型的质控结果查看,如:RNAseq、Whole-Genome Seq、Bisulfite Seq、Hi-C和MultiQC_NGI;
二、安装MultiQC
## 安装conda
## 安装python2环境
##conda create --name python2 python=2.7 -c https://mirrors.ustc.edu.cn/anaconda/cloud/bioconda/ -y
conda activate python2 ##激活python2环境(conda)
##conda install multiqc
conda install -c bioconda -c conda-forge multiqc
multiqc . ##运行测试


三、MultiQC使用
1、将需要测评的数据用FastQC测评(详细操作见 “Fastqc安装运行(jdk安装)”)
2、以下为三份测评结果:
prefetch SRR15971001
prefetch SRR15971005
prefetch SRR15971013
##下载三份测评SRA数据
fastq-dump --gzip --split-files SRR15971001
fastq-dump --gzip --split-files SRR15971005
fastq-dump --gzip --split-files SRR15971013
##--gzip参数是为了生成压缩为gz格式的fastq文件,以节省磁盘空间
fastqc SRR15971001_1.fastq.gz
fastqc SRR15971001_2.fastq.gz
fastqc SRR15971005_1.fastq.gz
fastqc SRR15971005_2.fastq.gz
fastqc SRR15971013_1.fastq.gz
fastqc SRR15971013_2.fastq.gz
##双端测序,所以生成的是正向和反向文件,所以都需要测评
multiqc .
**中途退出的话,要重新 conda activate python2 ##激活python2环境(conda)
**再者需要将以上解压分析后的文件转移到一个文件夹下进行评估

html报告可以直接网页打开就可以查看
multiqc_data文件夹,包含一些数据基本的统计信息和日志文档

四、结果分析:
- General Statistics

所有样本数据基本情况统计
%Dups——重复reads的比例,该六个样本重复reads的比例都很高,说明有用的reads很少
%GC——GC含量占总碱基的比例,比例越小越好(主要看这个GC含量)测序中的GC偏好指的是基因组上GC含量在50%左右的区域更容易被测到,产生的reads更多,这些区域的覆盖度更高,在高GC或者低GC区域,不容易被测到,产生较少的reads,这些区域的覆盖度更少。
M Seqs——总测序量(单位:millions)
-
Sequence Counts

序列计数
这里可以看到重复reads,六个重复reads比例都偏高,说明这些样本的序列中有用的reads数目较少 -
Sequence Quality Histograms

每个read各位置碱基的平均测序质量
绿色区间——质量很好
橙色区间——质量合理
红色区间——质量不好
此处可以看出6个样本均落在绿色区间,测序质量良好
- Per Sequence Quality Scores

具有平均质量分数的reads的数量
绿色区间——质量很好
橙色区间——质量合理
红色区间——质量不好
可以看出这六个序列的整体测序质量还是很不错的,所有的都在绿色区域
- Per Base Sequence Content
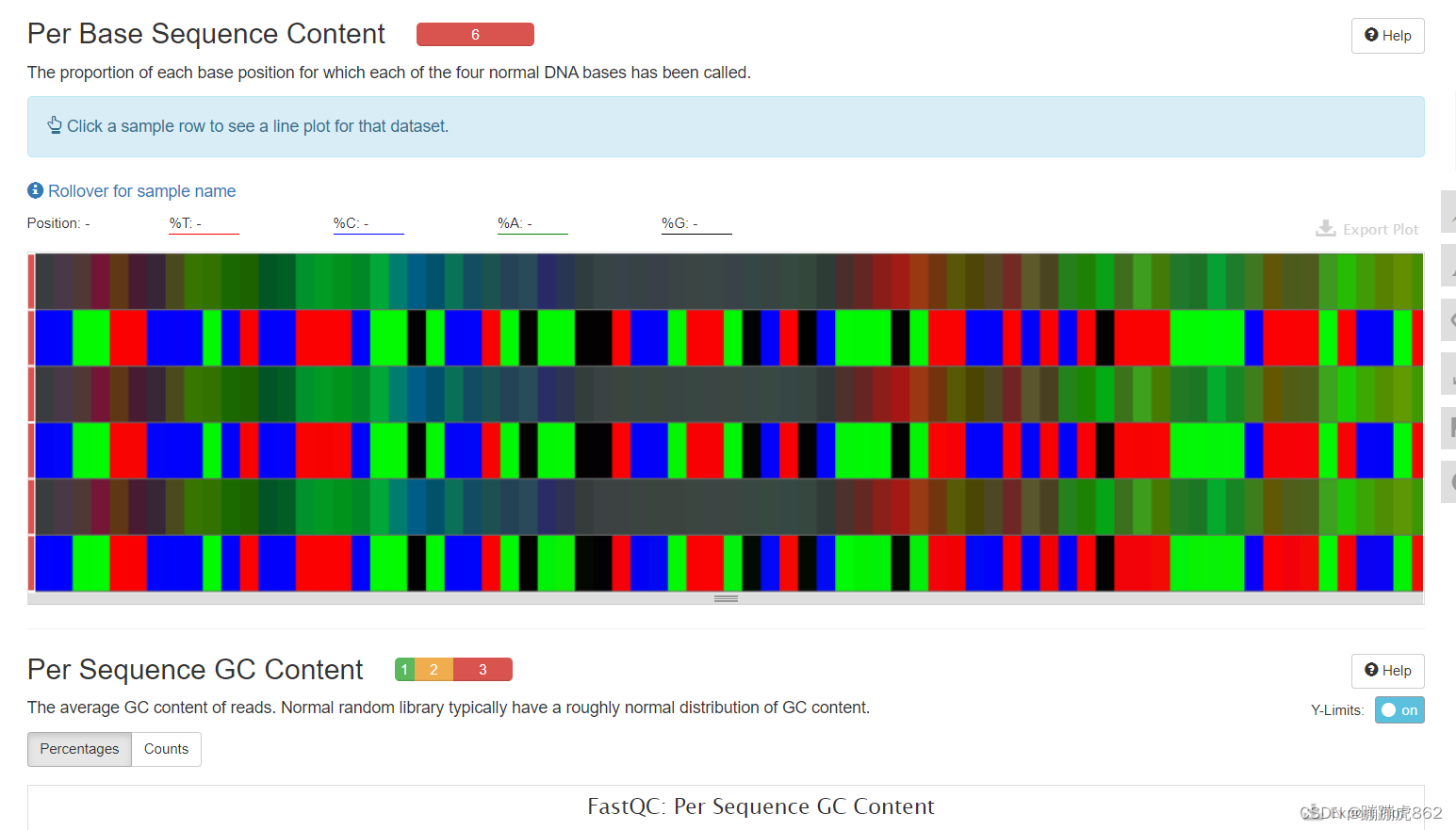
每个read各位置碱基ATCG的比例
结果显示六个序列都报错,说明每个位置每种碱基出现的概率差别很大,可能有过表达序列的污染
-
Per Sequence GC Content

reads的平均GC含量
正常的样本的GC含量曲线会趋近于正态分布曲线,曲线形状的偏差往往是由于文库的污染或是部分reads构成的子集有偏差(overrepresented reads)。形状接近正态但偏离理论分布的情况提示我们可能有系统偏差
这里结果显示部分序列被报错,从形状上来看曲线和正态曲线相差甚远,可能是由于文库的污染或是部分reads构成的子集有偏差造成的 -
Per Base N Content
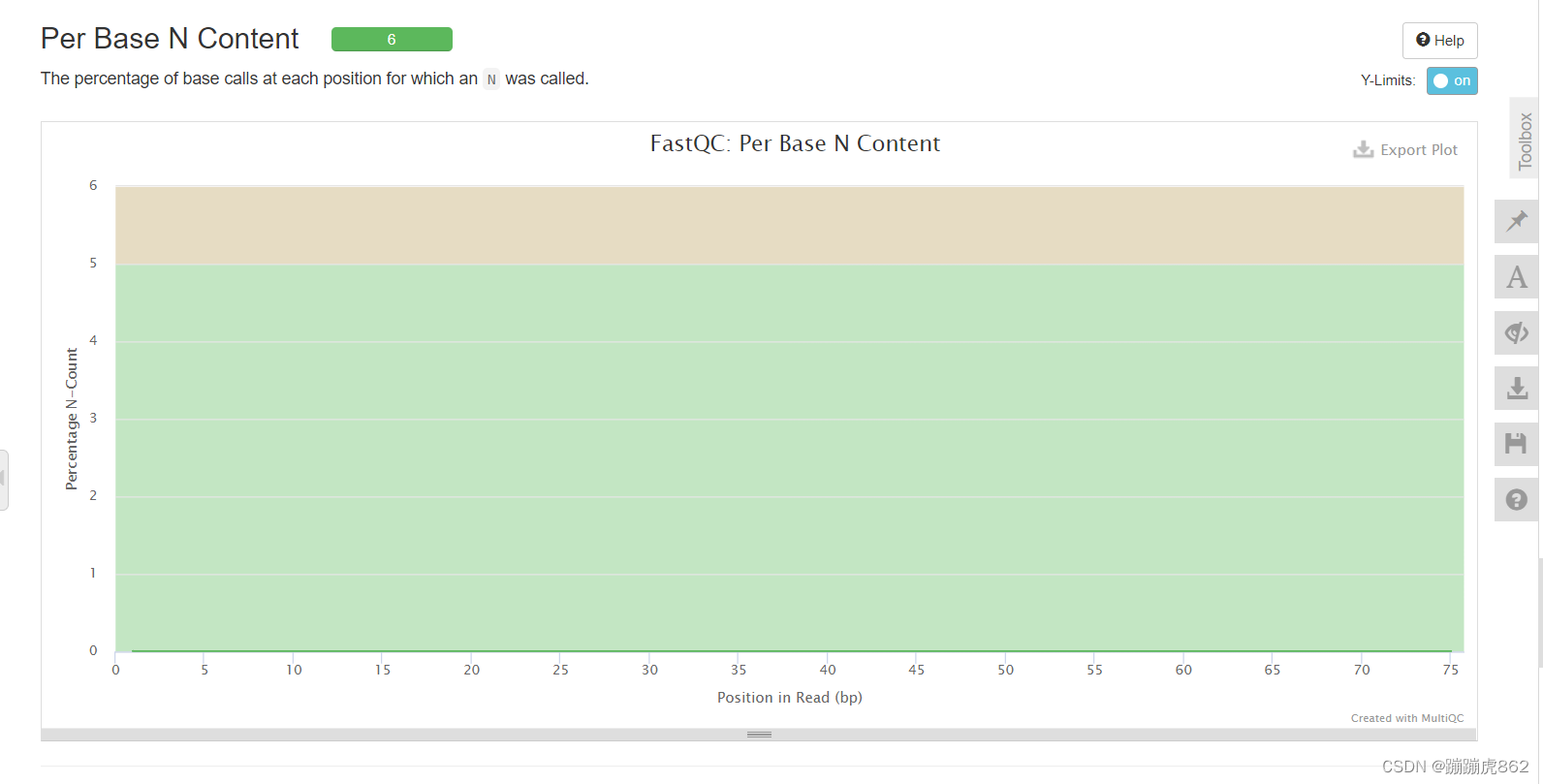
每条reads各位置N碱基含量比例
当测序仪器不能辨别某条reads的某个位置到底是什么碱基时,就会产生“N”,统计N的比率,正常情况下,N值非常小
- Sequence Length Distribution

序列长度分布
对于这六个序列,每次测序仪测出来的长度主要都在75bp
- Sequence Duplication Levels

每个序列的相对重复水平
- Overrepresented sequences
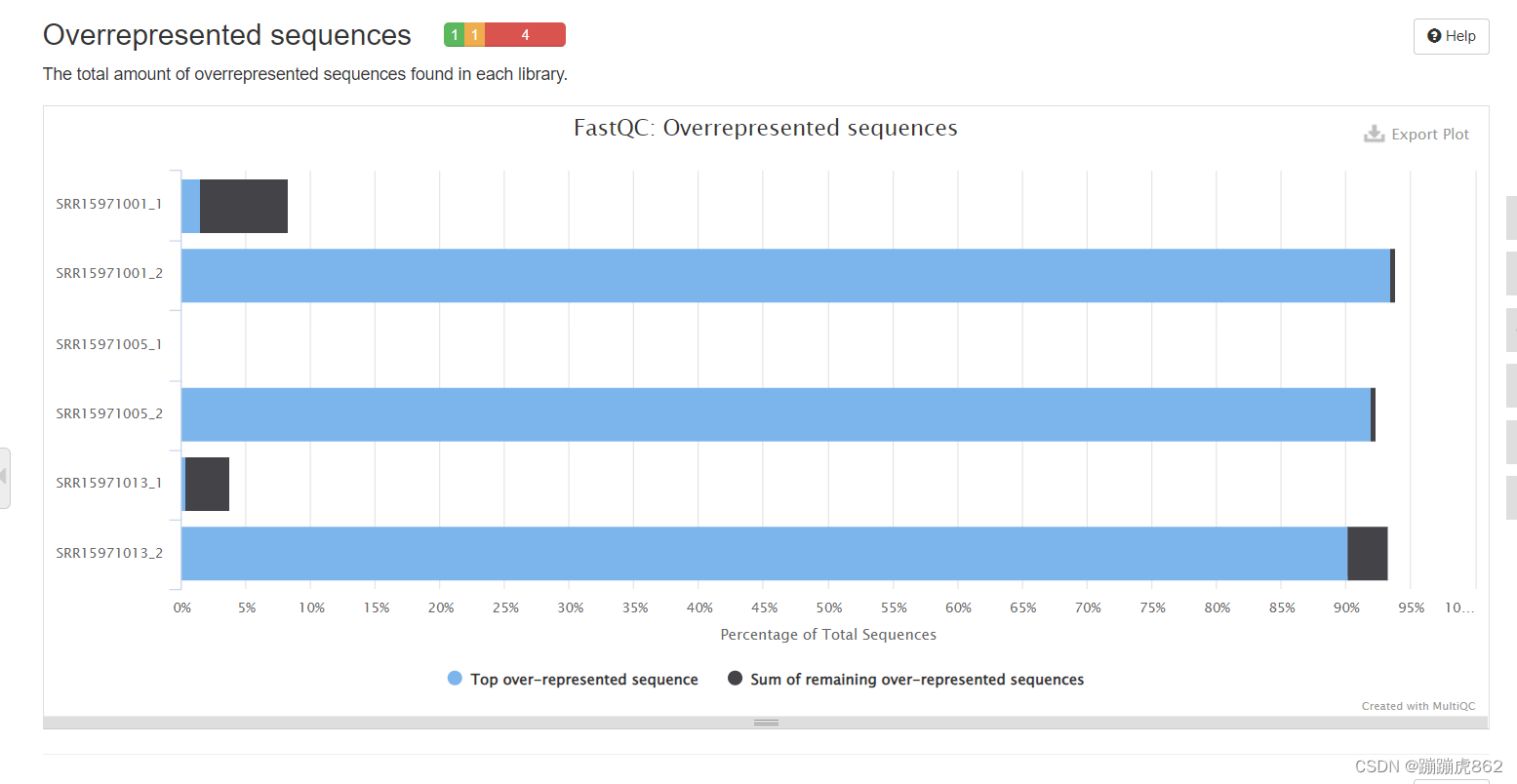
文库中过表达序列的比例
- Adapter Content

接头含量























 2597
2597

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








