
在上次的GSEA教程(“便携式”GSEA分析 - Do GSEA without "GSEA software" )中,我们给大家演示了如何跳过官方的GSEA软件,直接用R语言进行GSEA分析,非常方便快捷。在上次教程的最后,我们给大家提了一个问题:如何对某些特定的基因进行GSEA富集分析?比如,免疫相关的基因,肿瘤相关的基因等等。换句话说,我们在用GSEA做富集分析时,如何选择自己感兴趣的特定基因集合,这是一个非常实用的问题。比如我们研究的是肿瘤,那我们自然不想对不相关的通路进行富集,否则可能对我们的最终结果产生混淆。如果我们恰好对某一些通路感触颇深,或者就只想看看某几条通路是否在两个组别中有变化,那么接下来的操作会对大家有所帮助。本章内容,我们就来跟大家讲解,如何利用特定基因集进行GSEA分析。
首先,让我们再简单回顾下GSEA的操作过程,(1)我们需要按顺序排列好的gene list用于分析,(2)需要参考基因集pre-defined gene set,那么这个从哪里来呢?这么跟大家说吧,在GSEA中富集出来的基因功能类或者基因集合都是提前定义好的,谁定义的呢?当然是GSEA官方或者一些权威数据库(比如KEGG通路数据库,Gene Ontology数据库等)。举个例子,有哪些基因隶属于p53 signaling pathway或者MAPK singaling pathway是不需要我们操心的,有专家已经帮我们定义好了。那么,如何去查看或者下载这些预定义好的基因集合呢?打开如下链接:
https://www.gsea-msigdb.org/gsea/index.jsp
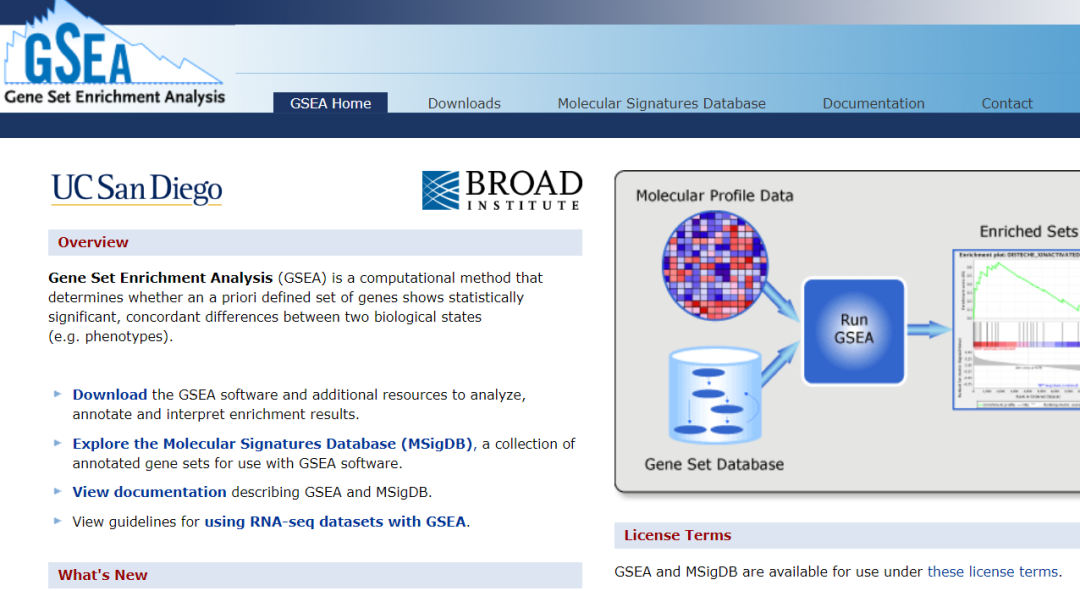
上面就是GSEA的官方网站的主页,如何找到我们感兴趣的基因集呢?如下图操作,找到MsigDB(Molecular Signatures Databases),这里面包含了GSEA定义的所有基因集,分为八个大类,分别由“H"和“C1”-“C7”开头,每个大类中都包含了哪些基因集在网页中有详细解释。比如C6是肿瘤相关,C7是免疫相关。
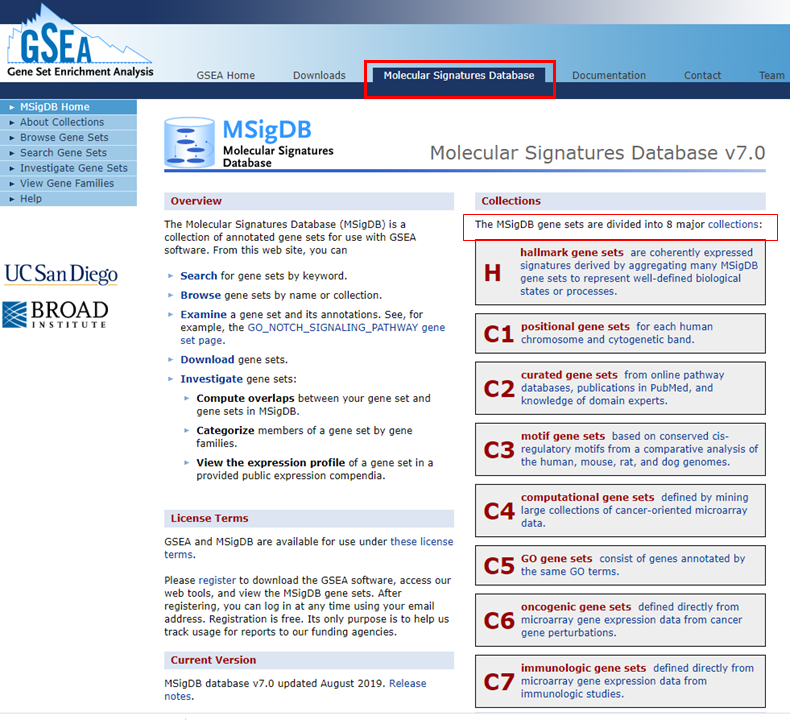
点击进去之后,有每个部分的详细描述,我们可以根据自己的需求选择特定的基因集。
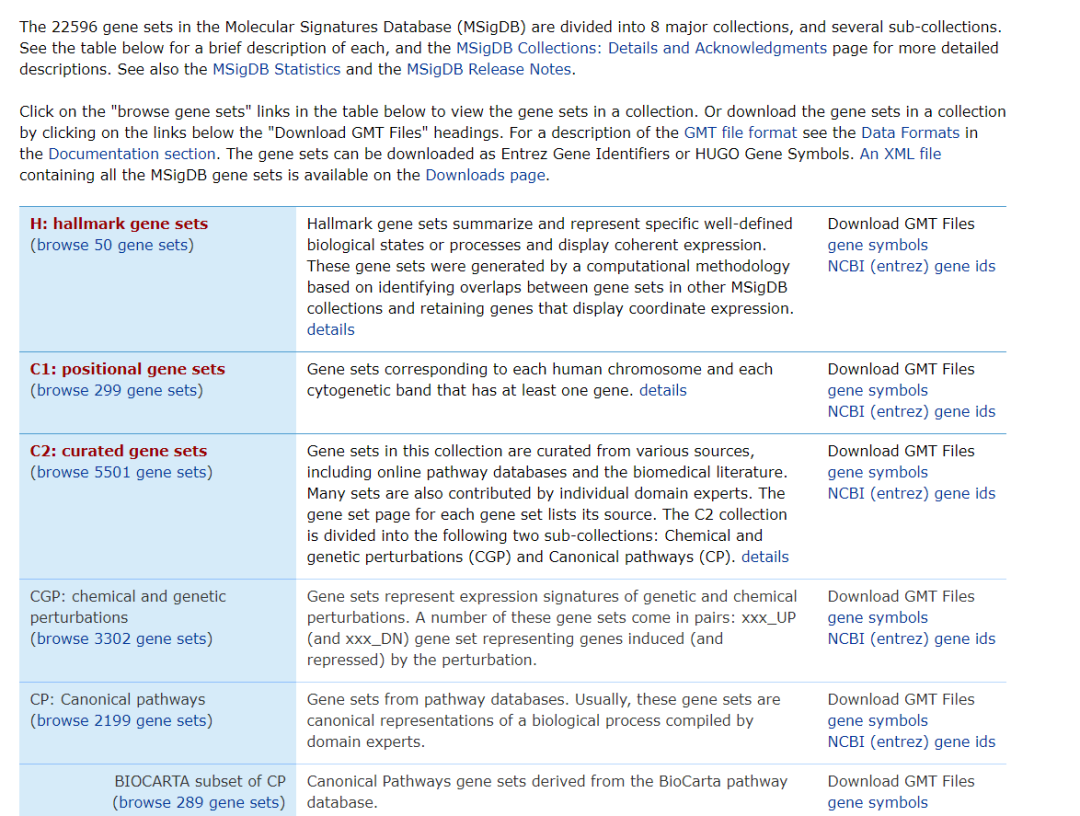
这里我们选择Hallmark gene set作为示例,通过其官方定义,我们知道该基因集是通过共表达模式定义了50个特定生物学过程或状态。为了将其用于后续的分析,我们可以将其下载下来。点击下图红色框中的链接即可下载gmt格式的基因集。

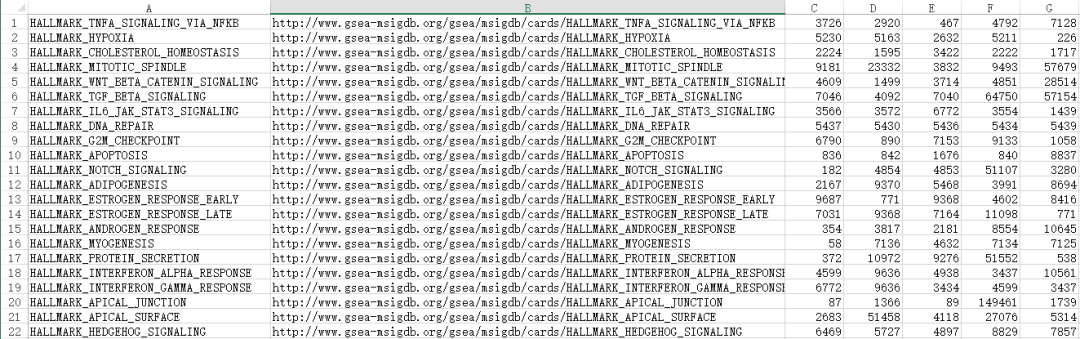
这个gmt文件里面是什么内容呢?我们可以把它用Excel表格打开看一眼。可以看到这个文件中每一行是一个基因集,第一列是基因集的名字,第二列是官网链接,后面的所有列就是该集合中包含的基因了(当然,这里是基因的Entrez ID)。
实例演练
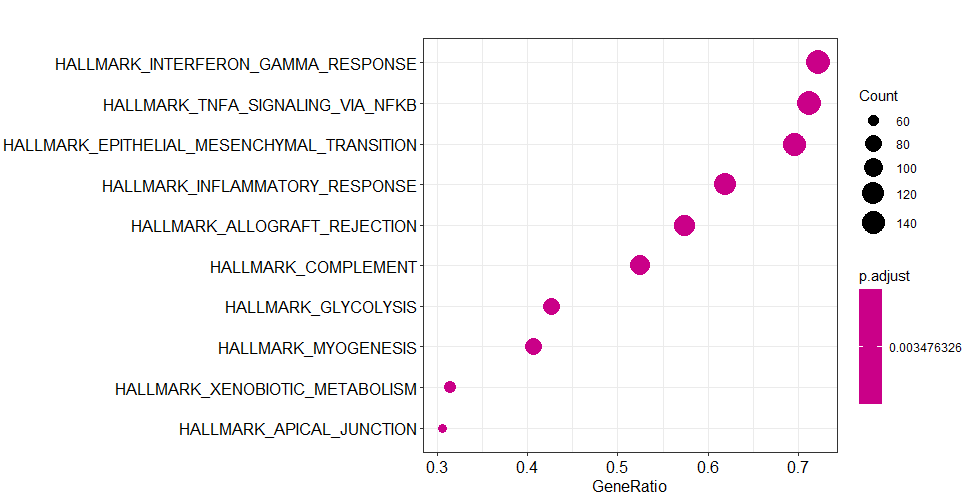
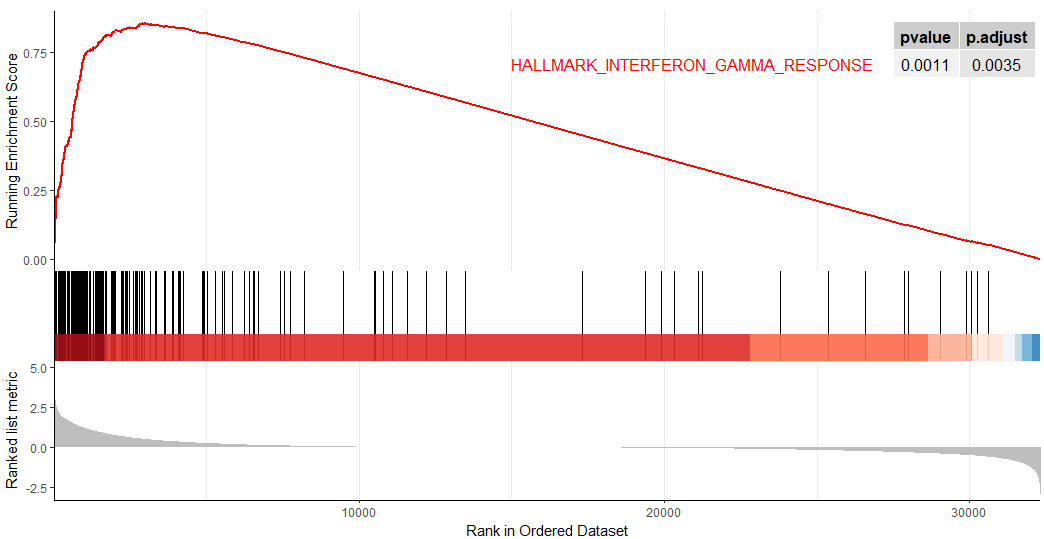
现在给大家演示如何用特定的.gmt文件(基因集合)进行GSEA分析,我们还是用上次的数据集(没有测试数据的同学,可以在文末联系客服小姐姐领取)。我们先讲第一种方法:还是使用clusterProfiler包进行分析,代码如下,在计算出来的结果中,我们可以选择前10个富集出来结果绘制气泡图,同时选择第一个基因集合INTERFERON GAMMA RESPONSE绘制gsea图:
1#clusterprofiler2pathway"h.all.v7.0.entrez.gmt")
3y 4dotplot(y,showCategory=10)
5gseaplot2(y,"HALLMARK_INTERFERON_GAMMA_RESPONSE",color = "red",pvalue_table = T)
如果你看倦了气泡图和GSEA图,在这里我们也给大家提供了另外一种方式,通过fgsea包选择特定基因集合进行分析,代码如下:
1#fgsea2pathways"h.all.v7.0.entrez.gmt")
3fgseaRes 1000,minSize=15, maxSize=500)
4topPathwaysUp 0][head(order(pval), n=10), pathway]
5plot.new()
6plotGseaTable(pathways[topPathwaysUp], gene, fgseaRes, gseaParam=0.5)
绘制出来的图形和结果如下所示,个人认为这种结果看上去更加简洁明了,我们最关心的NES、p value、adjusted p value以及Gene Ranks的缩略图都在图表中了。

好了,关于定制化的万能GSEA分析到这里告一段落,对于我们上次教程提出的问题,如果你想做免疫相关通路的GSEA,现在可以动手尝试起来喽。最后回顾一下,既然通路是由基因集组成的,我们自然可以选择我们想要研究的通路进行富集,同样那么在我们遇到一些崭新问题的时候,是不是也可以自己“制作”参考基因集呢?
本期教程,关于通过R语言进行GSEA分析,我们讲到这里,需要完整代码的同学,关注“科研猫”,联系文末客服领取。
本期干货
·
- 个性化GSEA分析 -
关注“科研猫”公众号,联系客服
胖雨小姐姐
or
折耳猫小姐姐
领取
更多科研新鲜资讯、文献精读和生物信息技能
请关注科研猫公众号

未经许可请勿随意转载,
版权事宜由上海辰明律师事务所提供法务支持




















 9075
9075











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








