单因素和多因素这里有时也会很困惑,在分析中也同样会遇到很多问题,比如当做单因素分析时,得到的 P 值显著,但是在做多因素时却不显著?又比如多因素分析时,选择变量的个数不同,得到的 P 值完全不同,显著的变量反而不显著,各种疑惑,一头雾水,这些问题该怎么解决呢?我感觉每个项目在做多因素分析的时候我都需要尝试十几次变量筛选,其目的就一个希望自己研究的那个因素能够被挑选出来,成为一个独立的预测因子,想必你也有同样的想法吧,我根据各种参考书籍所讲述的方法,总结出来一定的规律,可以节省时间,那下面我们就分析一下吧!
01 多元变量的筛选原则
我们在做多元变量分析时,经常会遇到变量筛选的问题,哪种变量符合哪种指标才能纳入后续的模型中这个问题也是非常困惑,那么我们就看下多元变量的筛选原则,大概有两种:
-
先做单因素分析,P 值显著的变量放入多元回归方程;
-
危险因素研究是根据对暴露因素效应值的影响筛选协变量。
对于 Logistic 回归和 Cox 回归可以使用7种变量筛选的方法,如下:
-
条件参数估计释然比检验(向前:条件);
-
最大似然估计的似然比检验(向前:LR);
-
Wald 卡方检验(向前:Wald);
-
条件参数估计似然比检验(向后:条件);
-
最大似然估计的似然检验(向后:LR);
-
Wald 卡方检验(向后:Wald);
-
Enter 法(变量全部进入,是默认方法)。
现实情况是,在临床研究报告是,大多选择单因素变量放入多因素中的筛选方式,首先单因素回归分析 P<0.1 的纳入最终的回归方程,此处变量筛选的标准也可设为 0.05 或 0.2,一般不会设置 P< 0.05, 也不会设置 P>0.2。而这种方法饱受统计学家的诟病。对于临床医生来说,这两种方法到底该如何选择呢?这个问题没有标准答案,但是变量筛选需考虑以下几点原则:
-
当有效样本量很大,统计学检验能足够时,可以使用上述6种变量自动筛选方法中的任何一种;
-
当不满足上述条件,或者其他原因导致的统计学效能不够时,应该采用大多数临床研究报告中采用的变量筛选方法,即首先逐个变量进行单因素回归分析,把单因素回归分析 P<0.2 的纳入最终的回归方程;
-
基于上述两种变量筛选方法,我们也需要同时考量那些已知确定与某种疾病预后显著相关的变量,即便未达到设定的统计学筛选标准,我们也应该纳入回归模型,这么做就是从临床专业角度筛选变量。
综上所述,第三种变量筛选的方法更加符合临床研究。我们既要统筹考虑统计学上的单因素分析结果与已知临床专业知识,同时还要考虑有效样本量。
02 多元变量的筛选实例分析
在选用单因素,多因素回归分析时需要我们具有一定的样本量,之前我们有讲过样本量的确定原则至少满足 EPV = 10,那么我们发现凡是文章中有这种表格的变量至少也在10个以上,因此样本量至少30以上,越多越好,这样才能满足结果有意义。
这里我们先看一篇文章里面的这种表格该怎么展示?下文是一篇基于国家癌症数据库分析淋巴性侵袭性微乳头状乳腺癌的预后的文章,如下:

Table 1. 是样本数量以及特征分布, Table 2. 先做单因素筛选变量放入多因素回归中,筛选影响有创微乳头切除术患者总体生存的预后因素,如下图:
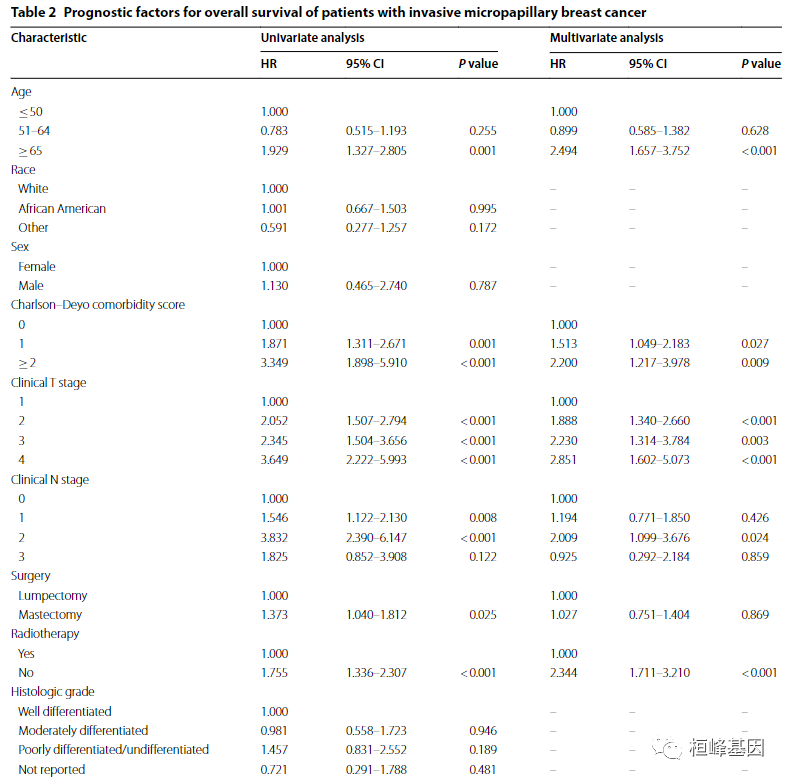
类似的临床研究基本上都会出现 Table 1. 如果预后影响因素过多,大部分都会选择 Table 2.多因素回归分析进一步确定模型的影响因子,拿这张表格利用R语言怎样才能快速解决,就是今天我们要细细道来!
03 批量********单元素回归分析表格实现
我们这里同样选择肺癌作为例子,不过这个实例中变量并不多,之前我们做单因素回归分析已经得到了一张表格,我们可以考虑在这基础上继续分析多因素分析,最后合并单因素分析和多因素分析的表格,即为我们所需要的最终结果。
我们先看数据包括16列,除了id, study, time,status之外,还有12个变量可以选择,如下:
library(survival)
data("colon")
str(colon)
'data.frame': 1776 obs. of 16 variables:
$ id : num 1 1 2 2 3 3 4 4 5 5 ...
$ study : num 1 1 1 1 1 1 1 1 1 1 ...
$ rx : Factor w/ 3 levels "Obs","Lev","Lev+5FU": 3 3 3 3 1 1 3 3 1 1 ...
$ sex : num 1 1 1 1 0 0 0 0 1 1 ...
$ age : num 43 43 63 63 71 71 66 66 69 69 ...
$ obstruct: num 0 0 0 0 0 0 1 1 0 0 ...
$ perfor : num 0 0 0 0 0 0 0 0 0 0 ...
$ adhere : num 0 0 0 0 1 1 0 0 0 0 ...
$ nodes : num 5 5 1 1 7 7 6 6 22 22 ...
$ status : num 0 0 1 1 0 0 0 0 0 0 ...
$ differ : num 2 2 2 2 2 2 2 2 2 2 ...
$ extent : num 3 3 3 3 2 2 3 3 3 3 ...
$ surg : num 0 0 0 0 0 0 1 1 1 1 ...
$ node4 : num 1 1 0 0 1 1 1 1 1 1 ...
$ time : num 1521 968 3087 3087 963 ...
$ etype : num 2 1 2 1 2 1 2 1 2 1 ...
- attr(*, "na.action")= 'omit' Named int [1:82] 127 128 165 166 179 180 187 188 197 198 ...
..- attr(*, "names")= chr [1:82] "127" "128" "165" "166" ...
colon<-na.omit(colon)
table(colon$status)
0 1
876 900
?colon
Usage
colon
data(cancer, package="survival")
Format
id: id
study: 1 for all patients
rx: Treatment - Obs(ervation), Lev(amisole), Lev(amisole)+5-FU
sex: 1=male
age: in years
obstruct: obstruction of colon by tumour
perfor: perforation of colon
adhere: adherence to nearby organs
nodes: number of lymph nodes with detectable cancer
time: days until event or censoring
status: censoring status
differ: differentiation of tumour (1=well, 2=moderate, 3=poor)
extent: Extent of local spread (1=submucosa, 2=muscle, 3=serosa, 4=contiguous structures)
surg: time from surgery to registration (0=short, 1=long)
node4: more than 4 positive lymph nodes
etype: event type: 1=recurrence,2=death
首先进行批量单因素 Cox 回归分析,这个之前已经讲过,但是这里也有些不同,就是连续型和分类型变量得到 summary() 有些区别,所以在写结果提取的时候需要分开,附上源代码,如下:
#######连续型变量
fit<-coxph(Surv(time, status)~age, data = colon)
summary(fit)
Call:
coxph(formula = Surv(time, status) ~ age, data = colon)
n= 1776, number of events= 900
coef exp(coef) se(coef) z Pr(>|z|)
age 0.005894 1.005912 0.003064 1.924 0.0544 .
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
exp(coef) exp(-coef) lower .95 upper .95
age 1.006 0.9941 0.9999 1.012
Concordance= 0.526 (se = 0.012 )
Likelihood ratio test= 3.74 on 1 df, p=0.05
Wald test = 3.7 on 1 df, p=0.05
Score (logrank) test = 3.7 on 1 df, p=0.05
######多分类变量
fit<-coxph(Surv(time, status)~rx, data = colon)
summary(fit)
Call:
coxph(formula = Surv(time, status) ~ rx, data = colon)
n= 1776, number of events= 900
coef exp(coef) se(coef) z Pr(>|z|)
rxLev -0.11487 0.89148 0.08567 -1.341 0.180
rxLev+5FU -0.11439 0.89191 0.08103 -1.412 0.158
exp(coef) exp(-coef) lower .95 upper .95
rxLev 0.8915 1.122 0.7537 1.054
rxLev+5FU 0.8919 1.121 0.7609 1.045
Concordance= 0.516 (se = 0.01 )
Likelihood ratio test= 2.45 on 2 df, p=0.3
Wald test = 2.48 on 2 df, p=0.3
Score (logrank) test = 2.48 on 2 df, p=0.3
批量单因素 Cox 回归分析结果汇总,所以我们通过获取多项分类的子类名称来整理Cox 回归分析结果,先写个指标提取函数,如下:
y<- Surv(time=colon$time,event=colon$status==0)#0为复发
Uni_cox_model<- function(x){
surv <- as.formula(paste0 ("y~",x))
cox<- coxph(surv,data=colon)
sum<-summary(cox)
HR <- round(sum$coefficients[,2],2)
PValue <- round(sum$coefficients[,5],3)
lower <-round(sum$conf.int[,3],2)
upper <-round(sum$conf.int[,4],2)
subchar <-rownames(sum$coefficients)
HRa<-paste0(HR, " (", lower,'-',upper, ")",sep="")
Uni_cox_model<- data.frame('Characteristics' = paste(x,"_",subchar,sep=""),
'HRa'=HRa,
'P-value' = PValue,
'HR' = HR,
'lower' = lower,
'upper' = upper
)
return(Uni_cox_model)
}
整理批量单因素 Cox 回归结果,我们发现结果的表格中出现多分类的结果,其中是由一个分类作为reference,整理表格是需要我们稍微修改一下,如下:
#转换成数据框,并转置
library(plyr)
univ_results <- lapply(covariates, Uni_cox_model)
univ_results <- ldply(univ_results,data.frame)
#最后,将P值=0的变为p<0.0001
univ_results$PValue[univ_results$PValue==0]<-"<0.001"
names(univ_results)=c("Variants","Hazard Ratio (95%CI)","P-value","","","")
univ_results
Variants Hazard Ratio (95%CI) P-value
1 sex_sex 1.12 (0.98-1.28) 0.092 1.12 0.98 1.28
2 age_age 1.01 (1-1.01) 0.054 1.01 1.00 1.01
3 obstruct_obstruct 1.05 (0.89-1.25) 0.545 1.05 0.89 1.25
4 perfor_perfor 1.09 (0.71-1.66) 0.701 1.09 0.71 1.66
5 adhere_adhere 0.94 (0.76-1.15) 0.540 0.94 0.76 1.15
6 nodes_nodes 1.04 (1.01-1.07) 0.004 1.04 1.01 1.07
7 differ_differ2 1.02 (0.83-1.27) 0.822 1.02 0.83 1.27
8 differ_differ3 1.13 (0.86-1.47) 0.377 1.13 0.86 1.47
9 extent_extent2 1.48 (1-2.19) 0.050 1.48 1.00 2.19
10 extent_extent3 1.58 (1.1-2.26) 0.014 1.58 1.10 2.26
11 extent_extent4 1.53 (0.9-2.58) 0.114 1.53 0.90 2.58
12 rx_rxLev 0.89 (0.75-1.05) 0.180 0.89 0.75 1.05
13 rx_rxLev+5FU 0.89 (0.76-1.05) 0.158 0.89 0.76 1.05
14 surg_surg1 0.87 (0.75-1.02) 0.083 0.87 0.75 1.02
15 etype_etype2 0.89 (0.78-1.01) 0.081 0.89 0.78 1.01
16 node4_node41 1.2 (1-1.44) 0.045 1.20 1.00 1.44
04 多元素回归分析表格实现
再来看多因素回归分析,根据上面介绍的多变量筛选原则,我们选择 分别看下P值区域值不同时,变量的个数,这里我们选择 P<0.1 的变量放入多元回归模型中,也可以三种情况都做一次多因素分析,对比结果,如下:
univ_results$Variants[univ_results$`P-value`<0.2]
[1] "sex_sex" "age_age" "nodes_nodes" "extent_extent2" "extent_extent3"
[6] "extent_extent4" "rx_rxLev" "rx_rxLev+5FU" "surg_surg1" "etype_etype2"
[11] "node4_node41"
univ_results$Variants[univ_results$`P-value`<0.1]
[1] "sex_sex" "age_age" "nodes_nodes" "extent_extent2" "extent_extent3"
[6] "surg_surg1" "etype_etype2" "node4_node41"
univ_results$Variants[univ_results$`P-value`<0.05]
[1] "nodes_nodes" "extent_extent3" "node4_node41"
多因素Cox回归结果整理, 入选为 “sex”,“age”,“nodes”,“extent”,“surg”,“etype”,“node4”,还是蛮多的, 如下:
########多因素分析
y<- Surv(time=colon$time,event=colon$status==0)#0为复发
#1.提取单因素p<0.1变量
univ2mul<-univ_results$Variants[univ_results$`P-value`<0.1]
univ2mul
#2.多因素模型建立
mul_Variants=NULL;
for (i in 1:length(univ2mul)) {
mul_Variants[i]=strsplit(univ2mul,split="_")[i][[1]][1]
}
mul_Variants=unique(mul_Variants)
mul_Variants
"sex" "age" "nodes" "extent" "surg" "etype" "node4"
mul_cox_model<- as.formula(paste0 ("y~",
paste0(mul_Variants,
collapse = "+")))
mul_cox<-coxph(mul_cox_model,data=colon)
cox_sum<-summary(mul_cox)
#3.提取多因素回归的信息
HR<- round(cox_sum$coefficients[,2],2)
PValue<- round(cox_sum$coefficients[,5],4)
lower<-round(cox_sum$conf.int[,3],2)
upper<-round(cox_sum$conf.int[,4],2)
#4.多因素结果优化并成表:mul_cox1
HRa<-paste(mul_HR," (", lower,'-',upper,")",sep = "")
mul_results<- data.frame("HRa"=HRa,"P"=PValue,"HR"=HR,"lower"=lower,"upper"=upper)
Variants=data.frame(rownames(mul_results))
rownames(mul_results)=NULL
mul_results<-cbind(Variants,mul_results)
colnames(mul_results)=c("Variants","Hazard Ratio (95%CI)","P-value","","","")
mul_results
Variants Hazard Ratio (95%CI) P-value
1 sex 1.11 (0.97-1.26) 0.1380 1.11 0.97 1.26
2 age 1.01 (1-1.01) 0.0463 1.01 1.00 1.01
3 nodes 1.04 (1-1.09) 0.0580 1.04 1.00 1.09
4 extent2 1.44 (0.97-2.15) 0.0695 1.44 0.97 2.15
5 extent3 1.54 (1.07-2.23) 0.0208 1.54 1.07 2.23
6 extent4 1.38 (0.81-2.35) 0.2368 1.38 0.81 2.35
7 surg1 0.86 (0.73-1) 0.0495 0.86 0.73 1.00
8 etype2 0.89 (0.78-1.02) 0.0900 0.89 0.78 1.02
9 node41 0.95 (0.71-1.27) 0.7392 0.95 0.71 1.27
05 单/多元素回归分析表格合并
最后合并单因素和多因素Cox回归的表格,最后就是我们需要的表格,放在word里面稍微修改一下就完成了整个过程,有些公众虽然有些过,但是过程都不算完美,我在千人的基础上把整个过程又重新调整了一下,如下:
#########合并表格
univ_Variants=NULL
for (i in 1:nrow(univ_results)) {
univ_Variants[i]=strsplit(univ_results$Variants,split="_")[i][[1]][2]
}
univ_results$Variants<-univ_Variants
final_results<-merge(univ_results[,1:3],mul_results[,1:3],by="Variants",all=TRUE)
final_results
Variants Hazard Ratio (95%CI).x P-value.x Hazard Ratio (95%CI).y P-value.y
1 adhere 0.94 (0.76-1.15) 0.540 <NA> NA
2 age 1.01 (1-1.01) 0.054 1.01 (1-1.01) 0.0463
3 differ2 1.02 (0.83-1.27) 0.822 <NA> NA
4 differ3 1.13 (0.86-1.47) 0.377 <NA> NA
5 etype2 0.89 (0.78-1.01) 0.081 0.89 (0.78-1.02) 0.0900
6 extent2 1.48 (1-2.19) 0.050 1.44 (0.97-2.15) 0.0695
7 extent3 1.58 (1.1-2.26) 0.014 1.54 (1.07-2.23) 0.0208
8 extent4 1.53 (0.9-2.58) 0.114 1.38 (0.81-2.35) 0.2368
9 node41 1.2 (1-1.44) 0.045 0.95 (0.71-1.27) 0.7392
10 nodes 1.04 (1.01-1.07) 0.004 1.04 (1-1.09) 0.0580
11 obstruct 1.05 (0.89-1.25) 0.545 <NA> NA
12 perfor 1.09 (0.71-1.66) 0.701 <NA> NA
13 rxLev 0.89 (0.75-1.05) 0.180 <NA> NA
14 rxLev+5FU 0.89 (0.76-1.05) 0.158 <NA> NA
15 sex 1.12 (0.98-1.28) 0.092 1.11 (0.97-1.26) 0.1380
16 surg1 0.87 (0.75-1.02) 0.083 0.86 (0.73-1) 0.0495
##########The end
最后看下放在word里面整理之后的效果,这个三线表的设计也是需要技巧的,看效果吧,如下:
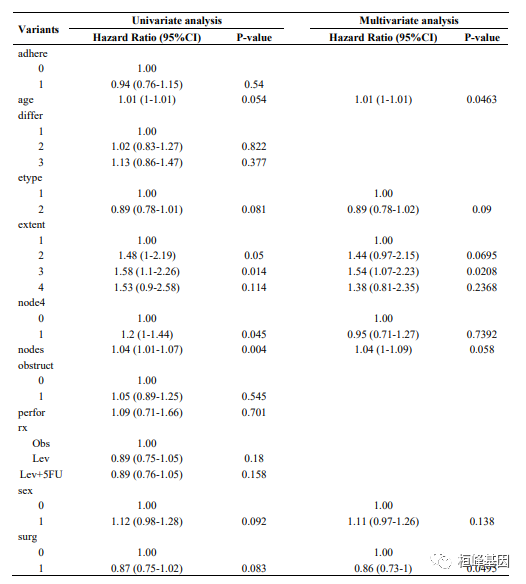
终于完成了,您一定要学会了,因为我也搞了好长时间才弄出来,希望您也能学到,如果自己仍然搞不定,关注公众号,扫码进群,我来教你哦!

Reference:
-
Kang SJ, Cho YR, Park GM, et al. Predictors for functionally significant in-stent restenosis: an integrated analysis using coronary angiography, IVUS, and myocardial perfusion imaging. JACC Cardiovasc Imaging. 2013;6(11):1183-1190.
-
Lewis GD, Xing Y, Haque W, et al. Prognosis of lymphotropic invasive micropapillary breast carcinoma analyzed by using data from the National Cancer Database. Cancer Commun (Lond). 2019;39(1):60.
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
32篇原创内容
公众号









 本文详细介绍单因素和多因素回归分析的选择与应用,探讨变量筛选原则与实例分析,通过R语言实现批量单因素和多因素Cox回归分析,最终合并结果并优化呈现。
本文详细介绍单因素和多因素回归分析的选择与应用,探讨变量筛选原则与实例分析,通过R语言实现批量单因素和多因素Cox回归分析,最终合并结果并优化呈现。

















 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








