
桓峰基因公众号推出单细胞生信分析教程并配有视频在线教程,目前整理出来的相关教程目录如下:
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
SCS【21】单细胞空间转录组可视化 (Seurat V5)
SCS【22】单细胞转录组之 RNA 速度估计 (Velocyto.R)
这期介绍一下单细胞数据整合,也是我们最近根据需求需要将不同时期不同批次不同平台的测序结果进行整合,这其中涉及到很多问题,那么推荐Harmony用于单细胞的数据整合效果还是很不错的,下面就看下例子,数据过大就考虑使用Linux上操作吧,由于软件包自带的例子数据量子集,自己的PC还是可以操作的!
简介
单细胞RNA-seq数据集的多样性允许在各种生物和临床条件下对细胞类型进行完整的转录表征。然而,将它们一起分析是具有挑战性的,特别是当使用不同的技术分析数据集时,因为生物和技术差异是分散的。这期我们介绍Harmony (https://github.com/immunogenomics/harmony),这是一种将单细胞投影到共享嵌入中的算法,其中细胞按细胞类型分组,而不是按数据集特定条件分组。同时解释了多种实验和生物因素。在分析中,展示Harmony的性能优于先前发布的算法,同时需要更少的计算资源。Harmony可以在一台个人计算机上集成约106个细胞。Harmony应用于外周血单个核细胞,这些数据集来自具有较大实验差异的数据集、胰岛细胞的五项研究、小鼠胚胎发生数据集以及scRNA-seq与空间转录组学数据的整合。

特征
快速:分析笔记本电脑上的数千个细;
敏感:不同的细胞类型可能在每批中存在或不存在;
准确:整合来自多个供体、组织甚至不同技术的细胞。
算法介绍
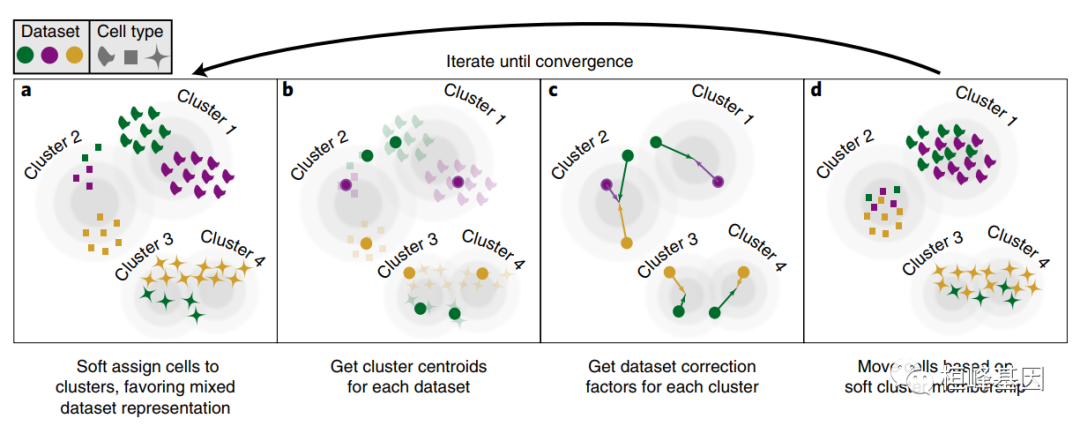
PCA将细胞嵌入到降维空间中。Harmony接受这个简化空间中的单元坐标,并运行迭代算法来调整数据集的特定效果。a, Harmony使用模糊聚类将每个单元分配给多个聚类,而惩罚项确保每个聚类内数据集的多样性最大化。b, Harmony计算每个集群的全局质心,以及每个集群的数据集特定质心。c, 在每个聚类中,Harmony根据质心计算每个数据集的校正系数。d, 最后Harmony用一个特定于细胞的因子来校正每个细胞:一个数据集校正因子的线性组合,由步骤a中对细胞的软聚类分配加权。Harmony重复步骤a到d,直到收敛。聚类分配与数据集之间的依赖性随着每一轮的进行而减弱。数据集用颜色、不同形状的单元格类型表示。
软件包安装
harmony 软件包有两种方式,都非常方便:
install.packages('harmony')
#or
library(devtools)
install_github("immunogenomics/harmony")加载软件包:
library(harmony)## Warning: 程辑包'harmony'是用R版本4.2.3 来建造的## 载入需要的程辑包:Rcpp数据读取
整合来自10X的细胞系数据集下面的例子是手稿中的图2。我们从10X网站下载了3个细胞系数据集。前两个(jurkat和293t)来自纯细胞系,而“一半”数据集是jurkat和HEK293T细胞50:50的混合物。我们用标准标记XIST推断细胞类型,因为这两个细胞系分别来自一个男性和一个女性供体。support.10xgenomics.com/single-cell-gene-expression/datasets/1.1.0/jurkat *
support.10xgenomics.com/single-cell-gene-expression/datasets/1.1.0/293t *
support.10xgenomics.com/single-cell-gene-expression/datasets/1.1.0/jurkat:293t_50:50
我们对细胞进行文库归一化,对计数进行对数转换,并对基因进行缩放。然后进行PCA,保留前20位PC。
data(cell_lines)
V <- cell_lines$scaled_pcs
meta_data <- cell_lines$meta_data例子实操
最初,细胞按数据集(左)和单细胞类型(右)聚类。
这里面需要注意一下 do_scatter()来自vignettes上的函数,以及 colors_use <- c(jurkat = '#810F7C', t293 = '#D09E2D',half = '#006D2C')
p1 <- do_scatter(V, meta_data, "dataset") + labs(title = "Colored by dataset")
p2 <- do_scatter(V, meta_data, "cell_type") + labs(title = "Colored by cell type")
cowplot::plot_grid(p1, p2)
让我们运行Harmony从嵌入中去除原始数据集的影响。缺省情况下,Harmony接受规范化的基因表达矩阵并执行PCA。因为这里已经有了pc,所以指定do_pca=FALSE。矩阵harmony_embeddings是经过Harmony校正的PCA嵌入矩阵。
harmony_embeddings <- harmony::HarmonyMatrix(V, meta_data, "dataset", do_pca = FALSE,
verbose = FALSE)在Harmony之后,数据集现在是混合的(左),细胞类型仍然是分开的(右)。
p1 <- do_scatter(harmony_embeddings, meta_data, 'dataset') +
labs(title = 'Colored by dataset')p2 <- do_scatter(harmony_embeddings, meta_data, 'cell_type') +
labs(title = 'Colored by cell type')cowplot::plot_grid(p1, p2, nrow = 1)
与软件包的接口还可以将Harmony作为几个软件包(如Seurat、MUDAN和scran)中已建立的管道的一部分运行。有关这些小插图,请访问我们的网站。运行Harmony最常见的方式是在降维上运行,比如主成分分析(PCA)中的PC嵌入。如果使用低维嵌入,则设置do_pca=FALSE。
cell_lines$meta_data[1:5, ]
## # A tibble: 5 × 5
## cell_id dataset nGene percent_mito cell_type
## <chr> <chr> <int> <dbl> <chr>
## 1 half_GTACGAACCACCAA half 1508 0.0148 jurkat
## 2 t293_AGGTCATGCACTTT t293 4009 0.0232 t293
## 3 half_ATAGTTGACTTCTA half 3545 0.0153 jurkat
## 4 half_GAGCGGCTTGCTTT half 2450 0.0170 jurkat
## 5 jurkat_CTGATACTCCGTAA jurkat 2388 0.0601 jurkat
cell_lines$scaled_pcs[1:5, 1:5]
## X1 X2 X3 X4 X5
## 1 0.002806741 -0.0014502980 -0.0063901369 0.0002824677 0.001444069
## 2 -0.011669691 0.0008773044 0.0008972694 0.0013239566 -0.003291763
## 3 0.009334467 -0.0069718865 -0.0025989384 0.0018824674 -0.003803677
## 4 0.006339979 -0.0025175931 -0.0043900882 0.0002737979 -0.002497476
## 5 0.008545583 0.0070866873 -0.0022541011 0.0016793801 0.004554607
harmonized_pcs <- HarmonyMatrix(data_mat = cell_lines$scaled_pcs, meta_data = cell_lines$meta_data,
vars_use = "dataset", do_pca = FALSE)
harmonized_pcs[1:5, 1:5]
## X1 X2 X3 X4 X5
## [1,] 0.004311941 0.0023307199 -4.274589e-03 0.0004508426 0.001550416
## [2,] -0.011651843 0.0001980328 3.223454e-04 0.0010981036 -0.003421640
## [3,] 0.010933557 -0.0024395897 -7.736527e-05 0.0019918176 -0.003595491
## [4,] 0.008169854 0.0019220194 -1.537521e-03 0.0006239379 -0.002250048
## [5,] 0.006953862 0.0031422373 -4.574271e-03 0.0013961197 0.004478406归一化基因矩阵
您还可以在库大小规范化表达式计数的稀疏矩阵上运行Harmony。HarmonyMatrix()函数将缩放表达式数据、运行PCA并运行Harmony集成算法。
my_harmony_embeddings <- HarmonyMatrix(data_mat = normalized_counts, meta_data = meta_data,
vars_use = "dataset")在 Seurat 中运行 Harmony
您可以在 Seurat 工作流中运 行Harmony。您只需要对代码进行两处更改。使用 RunHarmony() 函数运行 Harmony 在下游分析中,使用 Harmony 嵌入代替PCA。例如,分两行分别运行Harmony和UMAP:
seuratObj <- RunHarmony(seuratObj, "dataset")
seuratObj <- RunUMAP(seuratObj, reduction = "harmony")Reference
Korsunsky, I., Millard, N., Fan, J. et al. Fast, sensitive and accurate integration of single-cell data with Harmony. Nat Methods 16, 1289–1296 (2019).
这个软件包代码量还是很多的,需要具有一定 R 语言编程基础,并不是看起来那么简单,所有好多老师想直接自己学习教程来分析,但是实质上没有基础还是很难实现,每步报错都不知道该怎样处理,是最崩溃的,所以有需求的老师可以联系桓峰基因,提供最优质的服务!!!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”!!!
























 5259
5259

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








