欢迎关注”生信修炼手册”!
GWAS中的Gene Set Analysis, 简称GSA分析,是从基因或者通路水平来进行关联分析,是建立在SNP水平的的GWAS分析结果基础上的,在更高的层次进行深入挖掘,以发现更加有用的信息。MAGMA是进行GSA分析的一款工具,其官网如下
https://ctg.cncr.nl/software/magma
该软件的安装过程如下
wget https://ctg.cncr.nl/software/MAGMA/aux_files/NCBI37.3.zip
unzip NCBI37.3.zip其分析步骤共分为以下三大步
1. annotation
第一步是一个预处理步骤,将SNP位点映射到基因上去。根据SNP的染色体位置进行判断,如果落在了某个基因内,则映射到该基因上,当然也支持基因区间的延伸,比如想将基因启动区的SNP也纳入研究范围,可以通过参数将基因的区间在上下游进行拓展,基本用法如下

需要两个输入文件,第一个文件是SNP的染色体位置, 对应参数snp-loc, 这个文件可以有两种格式,一种就像上述示例一样,直接采用plink中后缀为.bim的文件,当我们有plink格式的原始数据时,采用这种方法非常方便,第二种是纯文本格式,要求前3列分别为SNP ID, 染色体名称,染色体位置,有这3列就够了,其他列信息会被忽略。
第二个是基因的染色体位置,对应参数gene-loc, 对于human而言,官网提供了3种基因组版本的该文件

该文件的内容示意如下
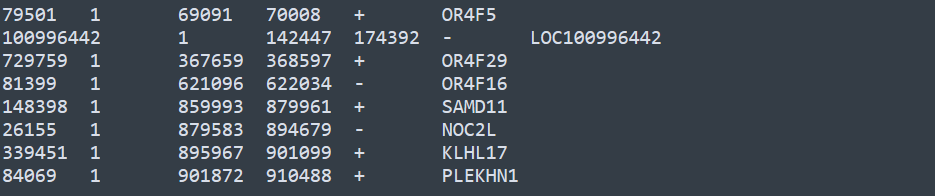
第一类为基因的Entrez ID, 第二列为染色体,第三列为转录起始,第四列为转录终止,前四列信息是必须的,第五列是基因的正负链,第六列是gene symbol。
运行成功后,会生成后缀为genes.annot的文件,内容如下

第一列为基因的Entrez ID, 第二列为染色体位置,其他列为对应的SNP ID,该软件的文本文件都用制表符\t分隔。如果需要拓展基因的区间,用法如下

上述用法表示在原来的基础上,上游延伸5kb, 下游延伸1.5kb。
2. gene analysis
基因水平的分析建立在SNP分析的基础上,该软件支持两种模式,第一种直接从原始的分型结果开始,第二种从GWAS分析结果,也就是SNP的P值开始,基本用法如下

bfile参数表示原始的分型结果,对于从SNP开始的分析,这个参数使用对应人群的分型结果即可,官网提供了1000G的数据供下载

gene-annot参数为第一步产生的SNP和基因的映射关系,pval参数为SNP对应的p值,格式如下

制表符分隔的两列,第一列为SNP的ID,第二列为对应的p值,输出文件后缀为genes.out, 内容示意如下
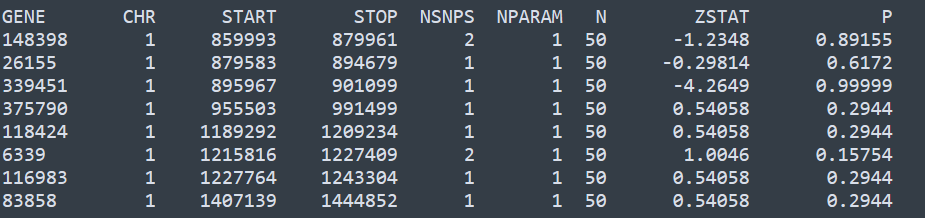
同时还会产生一个后缀为genes.raw的文件,用于后续的gene set分析。
3. gene set analysis
在基因分析的基础上,进行基因集的分析,基本用法如下

gene-results参数为第二步产生的文件,set-annot代表基因集,有如下两种格式

SET1表示基因集的名称,可以是pathway的编号,对应的基因集合用Entrez ID表示,输出结果后缀为.gsa.out, 内容示意如下

通过以上3步,就可以搞定GSA分析,该软件还支持添加协变量以及自变量间的相互作用,更多用法请参考官方文档。
·end·
—如果喜欢,快分享给你的朋友们吧—
往期精彩
基因型填充
CN








 本文介绍了GWAS中的Gene Set Analysis(GSA),它是在SNP分析基础上的更高层次挖掘。MAGMA是一款进行GSA分析的工具,分析包括3步:1. annotation,预处理SNP映射到基因;2. gene analysis,基于SNP分析结果进行基因水平分析;3. gene set analysis,进行基因集分析。通过这些步骤,可以在基因和通路层面发现更多关联信息。
本文介绍了GWAS中的Gene Set Analysis(GSA),它是在SNP分析基础上的更高层次挖掘。MAGMA是一款进行GSA分析的工具,分析包括3步:1. annotation,预处理SNP映射到基因;2. gene analysis,基于SNP分析结果进行基因水平分析;3. gene set analysis,进行基因集分析。通过这些步骤,可以在基因和通路层面发现更多关联信息。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 1973
1973

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








