点击蓝字 关注我们
甜樱桃T2T基因组及多组学方法揭示其进化和加倍后果色变化的候选基因

研究论文
● 原文链接DOI: https://doi.org/10.1002/imo2.13
●2024年6月30日,北京市农林科学院林业果树研究所张开春团队在iMetaOmics在线发表了题为“Insights into the evolution and fruit color change-related genes of chromosome doubled sweet cherry from an updated complete T2T genome assembly”的文章。
● 本研究完成了甜樱桃完整T2T基因组,为基因组进化和染色体加倍过程中的基因组变化以及果色相关基因的潜在变化提供了新的见解。
● 第一作者:张欣
● 通讯作者:张开春(kaichunzhang@126.com)
● 合作作者:段续伟、王晶、张晓明、闫国华、吴传宝、周宇
● 主要单位:北京市农林科学院林业果树研究所
亮 点
● 甜樱桃的完整T2T基因组组装;
● 甜樱桃的染色体加倍;
● 影响甜樱桃多倍体果色基因及突变的多组学挖掘。
摘 要
鉴于缺乏更高质量的基因组信息,为了促进甜樱桃基因组学研究,我们使用Pruns avium L.cv.Tieton在Hi-C校正后组装了完整的341.62Mb端粒-到-端粒(T2T)基因组。通过对这一最新的完整基因组的分析,研究了甜樱桃的进化特征和染色体加倍后果实颜色变化相关基因。
视频解读
Bilibili:https://www.bilibili.com/video/BV1iE4m1R7gm/
Youtube:https://youtu.be/0QeLT43bSV8
中文翻译、PPT、中/英文视频解读等扩展资料下载
全文解读
引 言
甜樱桃美早(Prunus avium L.cv.Tieton)是一个著名的商业品种,以其高营养和高保健价值的果实而闻名。目前,甜樱桃的基础研究主要基于基因组草图。然而,从未进行过基于具有较大的共线区域的高质量基因组的进化研究。同时,对染色体加倍品种的基因组和表型变化的研究还有待进一步深入。鉴于缺乏高质量的基因组来促进甜樱桃基因组研究,我们使用Tieton在Hi-C校正后组装了完整的341.62Mb端粒到端粒(T2T)基因组。我们使用先进的第三代循环一致性测序(CCS)和高通量染色体构象捕获(Hi-C)测序技术来组装高质量的基因组。此外,利用该T2T基因组研究了甜樱桃的进化特征和染色体加倍后果实颜色变化相关基因。我们分析了该基因组的重复序列和编码基因,并研究了甜樱桃与其他蔷薇科植物的遗传变异和进化关系。基于RNA-seq和重测序分析,我们能够识别加倍后果实发育过程中的差异表达基因(DEG)以及甜樱桃染色体加倍时发生的突变,总结了其影响表达的基因并揭示染色体加倍后潜在的果色相关基因的变化规律。总之,我们的研究完成了甜樱桃完整T2T基因组,为基因组进化和染色体加倍过程中的基因组变化以及果色相关基因的潜在变化提供了新的见解。
结果与讨论
基因组组装和注释
为了组装该基因组,使用了18.46 Gb(50.02×)MGISEQ数据、20.94 Gb(61.32×)PacBioHiFi数据和39.73 Gb(96.79×)Hi−C数据(表S1)。在去除污染、细胞器序列和重复重叠群后,通过整合已发表的基因组和Hi−C数据,产生了总大小为341,620,392bp、N50长度为39.81Mb的最终组装。该组装版本由8个重叠群组成,按已报道的染色体命名为Chr1−Chr8。BUSCO评估显示,基因组完整性为98.40%(图A、B;表S2),约含有59.29%的重复序列(包括TE(转座子))(表S3−S4)。整合不同的方法预测了58,204个基因,全部分布在8条染色体上,其中56,822个(97.63%)具有功能注释(表S5)。在所有8条染色体中鉴定到了端粒序列,长度从1,448到3,297bp(图S2;表S6)。根据长串联重复序列和Hi−C数据预测了着丝粒(图S2−S3)。此外,着丝粒区通常缺乏基因,不同着丝粒区域的重复序列类型各不相同,如LTR(长末端重复序列)/Copia或Gypsy重复序列(图S4−S5)的分布各不相同。所有先前报道的2,152个缺口在该基因组中都很好地补齐了(图A,F)。此外,与先前发表的基因组相比,新基因组新发现了205个新的DEG,512个增加的SV(结构变体)和9.55增加的LAI(LTR组装指数值)指数(图A),表明该新基因组的质量高(表S2)。
比较基因组分析
利用这个高质量的基因组,构建了进化树(图C)。根据分子钟分析,Prunus于6,404万(Mya)年前从Malus分化而来,其他物种从Pruns avium逐渐分化的时间约为48.39 Mya(图C)。这种大的时间跨度(95%置信区间)是由于其他基因组的低质量和没有更多的化石校准信息,与其他物种相比,我们推断出的分化时间相较整个进化史较近。我们进一步进行了基因家族扩张和收缩分析,在Pruns avium发现了3,836个扩张家族和2,087个收缩家族(图S6−S7),而在Prunus armeniaca发现了1,506个扩张和4,054个收缩家族(图C)。在樱桃的基因组中没有发现最近的WGD(全基因组复制)事件(除蔷薇科共有的历史加倍事件外),这与4DTv曲线一致,而略高的曲线可能是由于基因家族的扩张(图S8−S9)。
本研究中15个被分析物种的基因家族分布(图C)如图S10-S11所示,我们在新基因组中发现了322个独特的基因家族。共线性分析表明,李属中共线性基因的比例较高,表明李属中物种间的亲缘关系密切。在图C(图S12)中的10种李属植物中检测到几个在果实成熟中发挥作用的共线性基因,因此我们进一步分析了果实成熟和着色相关基因。
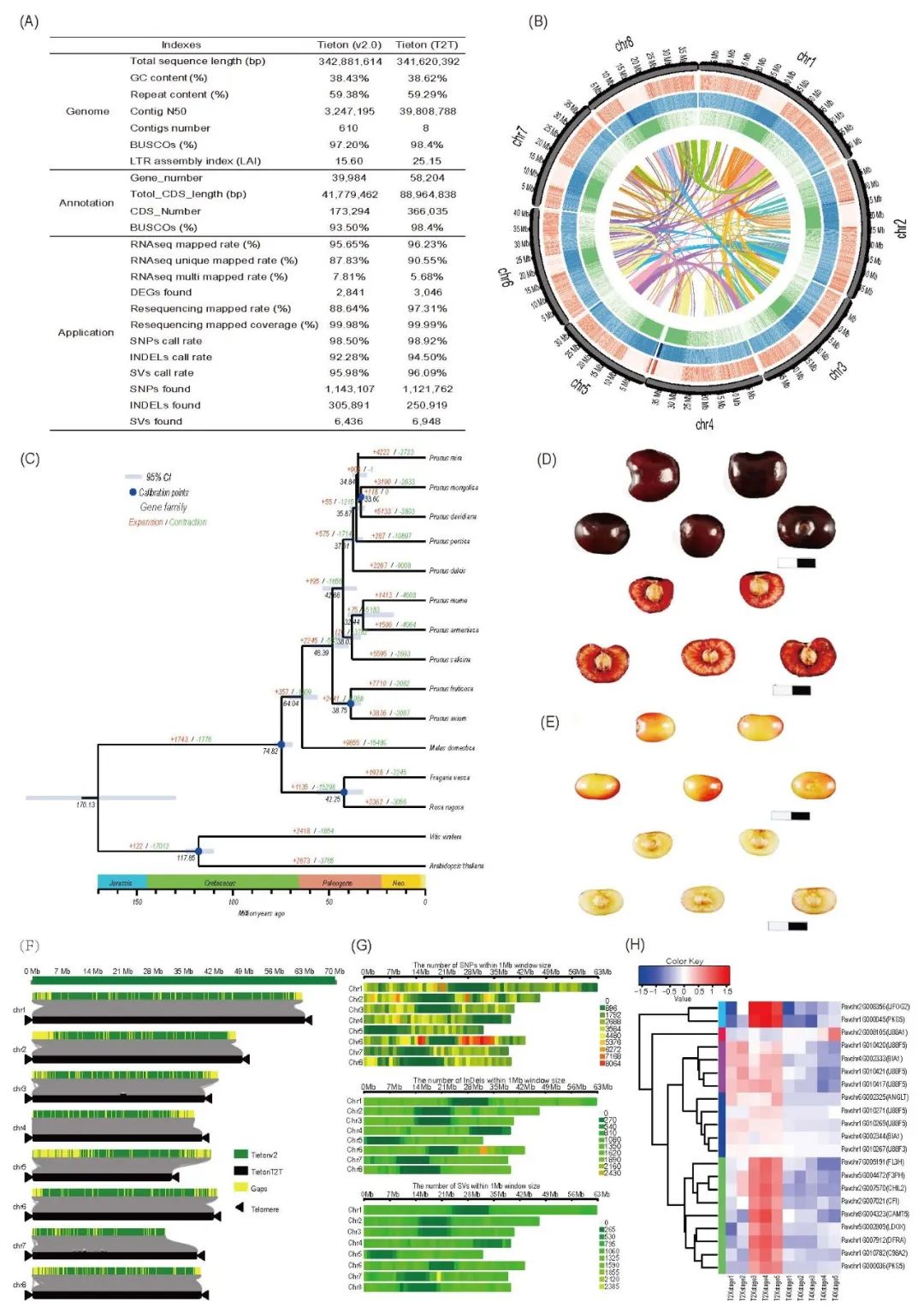
图 甜樱桃的最新完整T2T基因组组装
(A) Tieton基因组数据。对‘v2.0’和‘T2T’版本的Tieton基因组进行了比较;(B) Tieton基因组染色体的环形图。从外到内:8条染色体与其实际长度;基因密度;基因组GC含量;重复序列含量;全基因组共线性区块;(C)进化树显示了15个物种与分化时间的关系;(D)二倍体(T2X)成熟期果实的横截面,标尺为2厘米;(E)四倍体(T4X)成熟期果实的横截面,标尺为2厘米;(F)Tieton ‘v2.0’和‘T2T’版本基因组之间的共线性。共线区域由灰线表示。所有闭合的缺口区域显示为黄色块;(G)四倍体(T4X)Tieton中已鉴定到的突变分布和密度;(H) 突变影响的DEG热图(果色变化相关基因,括号中为基因名称)。
染色体加倍、转录组和突变分析
甜樱桃栽培中最紧迫的任务之一是创建四倍体甜樱桃种质,通过该种质可以从同倍性的中国樱桃资源中引入抗病性、风味和颜色基因。因此,研究染色体加倍的基因组变化不仅有助于鉴定性状相关基因,而且对创造新的甜樱桃种质具有重要的现实意义。我们首先获得了Tieton二倍体(T2X)和四倍体(T4X)个体的重测序和转录组数据,这些新种质被创制和观察了近10年(图D、E和S1;表S1)。使用该数据集和我们新的无间隙基因组(图F),在T2X和T4X的果实之间发现了3,046个DEG(1,718个下调和1,328个上调)(图A,S13−S14)。通过时间序列、WGCNA和KEGG分析进一步研究了这些DEG涉及的关键通路(图S15−S21)。为了探索四倍体发生的突变在多大程度上影响所发现的黄酮类和花青素类代谢基因的表达,我们将T4X重测序数据与新的T2X基因组进行比对,鉴定了1,121,762个高质量SNPs、250,919个INDEL和6,948个SV(图G;表S7),观察到85.71%的黄酮类和花青素DEG受到影响(沉默)(图H;表S8)。这些新发现的影响共表达转录因子的突变(表S9)将为进一步阐明染色体加倍甜樱桃颜色变化机制提供潜在的新靶点,填补染色体加倍植物基因组研究的空白。
数据和方法可用性
该项目的所有数据都在国家基因库(CNGBdb)生物项目下的数据库中:https://db.cngb.org/search/project/CNP0004619/ 和http://www.cherries.org.cn。补充材料(方法、图表、表格、图形摘要、幻灯片、视频、中文翻译版和更新材料)可在在线DOI或iMeta Science中找到http://www.imeta.science/imetaomics/。
引文格式:
Xin Zhang, Xuwei Duan, Jing Wang, Xiaoming Zhang, Guohua Yan, Chuanbao Wu, Yu Zhou, Kaichun Zhang. 2024. "Insights into the evolution and fruit color change-related genes of chromosome doubled sweet cherry from an updated complete T2T genome assembly." iMetaOmics e13. https://doi.org/10.1002/imo2.13
课题组简介
北京市农林科学院樱桃研究室设立于北京市农林科学院下设的林果所,成立于1997年,是“国家林业草原樱桃工程技术研究中心” “国家樱桃资源圃北京分库”和“中国园艺学会樱桃分会”挂靠单位,“樱桃产业国家创新联盟”副理事长单位。主要从事樱桃资源收集与评价、甜樱桃品种、砧木选育、栽培技术以及生物技术辅助育种等研究。主持或完成国家科技支撑、公益性行业专项、国家自然基金以及北京市科委、农委项目和课题50余项。现收集保存国内外樱桃资源350余份;选育8个甜樱桃品种,其中国审品种1个,获植物新品种权保护2个;选育砧木品种6个,其中具有新品种保护权品种4个;获得专利6项;发表学术论文150余篇,撰写论著11部;获北京市科技进步二等奖1项,三等奖1项,神农中华农业科技二等奖1项,北京市技术推广三等奖1项。在陕西澄城、河北三河、云南昆明等地建有区域试验基地9个。

主要研究方向为:
1. 甜樱桃新品种选育:自交结实、早果、丰产;大果、硬肉、酸甜适口;特殊品质和抗性(适合观光采摘、短低温、耐褐斑病、抗寒等);不同熟期,尤其极早熟和极晚熟
2. 樱桃砧木育种:易于繁殖、嫁接亲和性好;根系发达、固地性强;综合抗性强(抗根癌病、抗褐斑病、抗盐碱、耐涝、耐瘠薄等);早果矮化
3. 樱桃产业新技术研究:现代高效树型;早果丰产技术;樱桃设施栽培;病虫害防治、土肥水一体化等果园配套管理技术;以防裂果、防冻害和避雨为主的防灾减灾研究。
iMetaOmics
更多资讯
● iMeta姊妹刊iMetaOmics(定位IF>10)欢迎投稿!(2024.2.27)
● iMeta姊妹刊iMetaOmics编委招募 (定位IF>10) (2024.3.2)
● iMeta姊妹刊iMetaOmics电子版和印刷版ISSN申请获批(2024.4.1)
● iMeta姊妹刊iMetaOmics投稿系统正式上线(2024.4.17)
● iMeta姊妹刊iMetaOmics主编正式官宣(2024.4.22)
● 出版社iMetaOmics主页正式上线!(2024.4.28)
● iMetaOmics | 浙江大学宗鑫组揭示两猪种宿主-肠道菌群互作差异
● iMetaOmics | 罗鹏/袁硕峰/苗凯/程全发表STAGER: 生成式人工智能可靠性的标准化测试和评估推荐
● iMetaOmics | 徐州医科大杨欢组揭秘沙门氏菌-宿主-微生物群在免疫与代谢中的相互作
● iMetaOmics | 中科院动物所金坚石组综述16S rRNA基因扩增子测序技术的“前世今生”
● iMetaOmics | 浙大张天真组完成二倍体棉种泛基因组构建
● iMetaOmics | 张勇/李福平-先进糖蛋白组学在男性生殖研究中的潜在应用
●iMetaOmics | 暨南大学潘永勤/杨华组-炎症蛋白联合检测有利于诊断甲状腺乳头状癌和结节性甲状腺肿
更多推荐
(▼ 点击跳转)
iMeta | 引用13000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
3卷1期
2卷2期封底
2卷4期封底
3卷2期
3卷3期
3卷3期封底
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百千华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 20)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、前3年免出版费、50万用户的社交媒体宣传等。2022年2月正式创刊发行!发行后相继被Google Scholar、ESCI、PubMed、DOAJ、Scopus等数据库收录!2024年6月获得首个影响因子23.7,位列全球SCI期刊前千分之五(107/21848),微生物学科2/161,仅低于Nature Reviews,同学科研究类期刊全球第一,中国大陆11/514!
“iMetaOmics” 是“iMeta” 子刊,主编由中国科学院北京生命科学研究院赵方庆研究员和香港中文大学于君教授担任,定位IF>10的高水平综合期刊,欢迎投稿!
联系我们
iMeta主页:
http://www.imeta.science
姊妹刊iMetaOmics主页:
http://www.imeta.science/imetaomics/
出版社iMeta主页:
https://onlinelibrary.wiley.com/journal/2770596x
出版社iMetaOmics主页:
https://onlinelibrary.wiley.com/journal/29969514
iMeta投稿:
https://wiley.atyponrex.com/journal/IMT2
iMetaOmics投稿:
https://wiley.atyponrex.com/journal/IMO2
邮箱:
office@imeta.science







































 619
619

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








