
摘要
细胞色素是一类以铁卟啉(或血红素)作为辅基的电子传递蛋白,其b类细胞色素的辅基是原血红素。NCBI下载的13个物种的细胞色素b蛋白的氨基酸序列数据,通过MEGA软件构建系统发育树研究,能够为物种间的进化关系提供重要的参考信息。结果表明:虹鳟和泥鳅亲缘关系密切,蓝鲸和长须鲸进化上更接近鱼类,拟鳄龟、青蛙与原鸡关系密切,而与绿头鸭有一定亲缘关系。爬行动物和鸟类与鸵鸟的距离较远,而它们与非洲爪蟾的关系更为疏远。
关键词:细胞色素b蛋白、NCBI、MEGA、系统发育树
Abstract
Cytochromes are a class of electron transfer proteins with heme (or hemoglobin) as their prosthetic group, and the prosthetic group of type b cytochromes is heme. The amino acid sequence data of cytochrome b proteins from 13 species downloaded from NCBI were used to construct a phylogenetic tree using MEGA software, which can provide important reference information for the evolutionary relationships between species. The results show that rainbow trout and loach are closely related, blue whales and humpback whales are closer to fish in evolution, crocodiles, frogs, and chickens are closely related, and they are somewhat related to mallards. Reptiles and birds are more distant from ostriches, while their relationship with African clawed frogs is more distant.
Keywords:Cytochrome b protein,NCBI、MEGA、phylogenetic tree
一、背景
细胞色素b(CYTB)作为复合体III中唯一,一个线粒体DNA编码的转录物,参与了线粒体的电子传递链,在生物体的能量代谢中起重要作用[1]。它的进化速度适中,一个较小的基因片段就包含着从种内到种间,属间乃至科间的进化遗传信息,被认为是解决分类及系统进化问题可信的分子标记之一[2],其编码序列的系统发育分析已广泛应用于探讨生物之间的亲缘关系。NCBI,即美国国家生物技术信息中心,是一个提供生物信息学服务的国家级机构。它提供了广泛的生物信息资源,包括基因组数据库、生物样本数据库、文献数据库等。本研究旨在通过MAGE分析从NCBI上获取的13种生物包括哺乳动物、鸟类动物、爬行动物、两栖动物、鱼类动物等的细胞色素b蛋白序列,构建系统发育树, 系统发育树是一种图形化的表示方式,用于展示不同物种或生物个体之间的进化关系。它通过比较物种或个体的DNA、RNA或蛋白质序列等遗传信息来构建,该研究使用的是这13种物种的蛋白质序列信息,揭示这些生物在进化过程中的关系。
二、材料与方法
2.1数据来源与方法
以每一个物种的蛋白质登录号在NCBI中搜索
如图表1
表格 1 13种生物的基本信息

在NCBI中搜索下载其基因序列fasta格式如图表1,

图表 1使用蛋白质序列号再NCBI中搜索fasta格式
Fasta格式包含注释信息和序列信息,将13个物种的fasta格式整合在一个文本中,为了方便后续在使用MEGA构建进化树的时候查看各个物种间的关系,我将Fasta格式中注释信息修改为了每个物种的名称的拼音,如图表2。
图表 2 13个物种所有蛋白质序列fasta格式
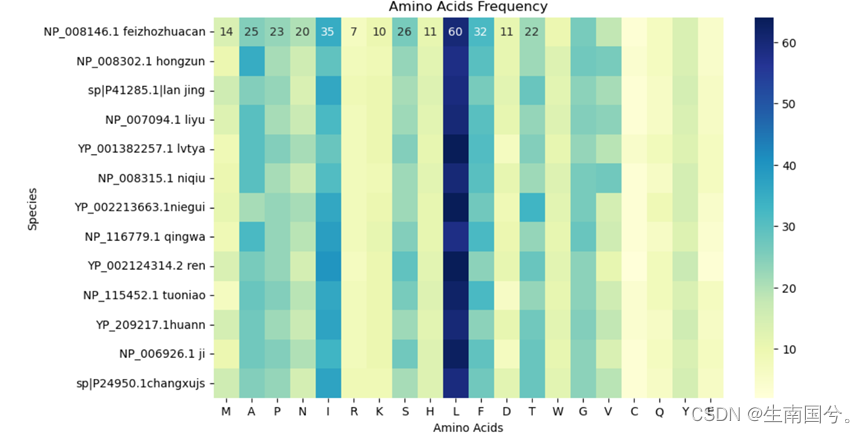
图表 3 编程实现提取13个物种氨基酸频次热图
2.2实验方法与参数
实验流程如下:
第一步:在MEGA中打开,存放的fasta格式将其保存为meg格式。 如图表4

图表 4将fasta格式在MEGA中打开
第二步:构建进化树,打开meg在models-find best models的Reslts中选择BIC参数最小的model进行构建。
这里选择“Number of Threads=7”如图表5 这个参数通常是指在并行计算中使用的线程数。线程是操作系统能够进行运算调度的最小单位。在多线程计算中,一个程序可以被分成多个线程来同时运行,这样可以加快程序的执行速度,尤其是在处理大型数据集或者复杂的计算任务时。

图表 5参数Number of Threads设置
该研究模型选择中,选择BIC最小的模型,如图表6。BIC(Bayesian Information Criterion,贝叶斯信息准则)是一种常用的统计指标,用于比较不同模型的拟合优度和复杂度。BIC考虑了两个因素:模型的拟合优度和模型的复杂度。在选择最佳模型时,我们希望找到一个既能很好地拟合数据又不会过度复杂的模型。
BIC的计算公式为:
BIC = -2 * ln(L) + k * ln(n)
其中,ln(L)是模型的对数似然函数值,k是模型的参数数量,n是样本数量。BIC值越小,说明模型的拟合优度越好,同时考虑到了模型的复杂度。因此,在选择构建进化树时,选择BIC最小的模型意味着选择了在给定数据集下既能较好地拟合数据又不会过度复杂的模型,从而提高了模型的准确性和可解释性。
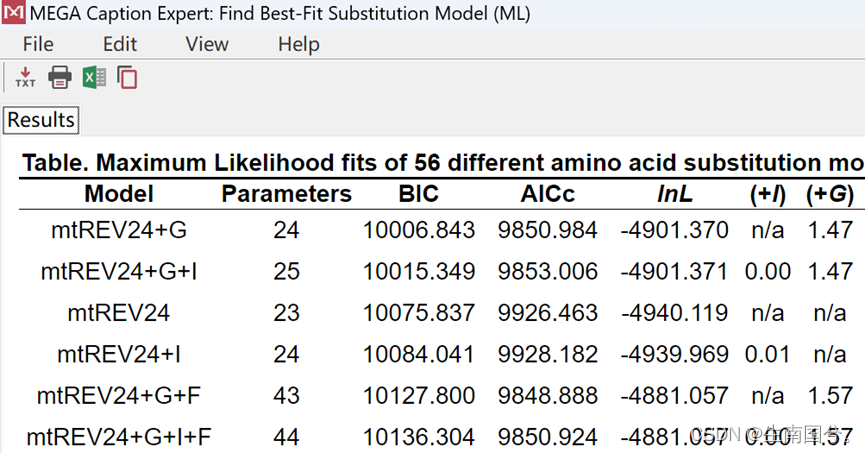
图表 6选择最小BIC
第三步:选择PHYLOGENY-Construct/Test XXX Tree,参数选择:Bootstrap,重复1000次。
如图表7,在构建进化树时,Bootstrap是一种常用的统计方法,用于评估进化树的可靠性。它通过从原始数据集中随机抽取一定数量的样本,并基于这些抽样数据构建多个进化树来估计进化树的稳健性和可信度。
重复1000次的意思是进行1000次Bootstrap重抽样,每次都从原始数据集中随机抽取样本来构建进化树。通过进行大量的重复,可以得到多个进化树,从而可以对进化树的拓扑结构和分支支持度进行统计分析,提高对真实生物进化关系的理解和可信度。

图表 7 Bootstrap 参数选择1000
第四步:进化树构建完成,进行调整与美化
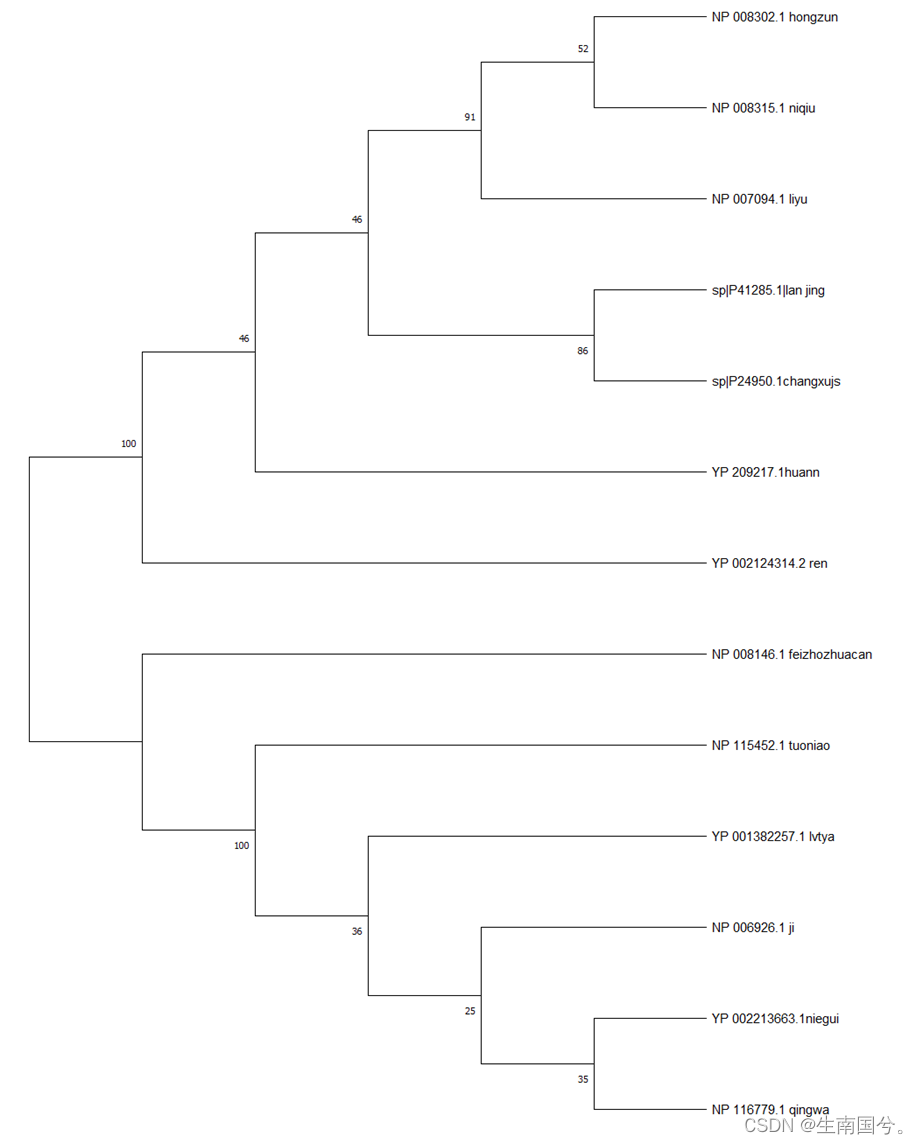
结果表明:
1.虹鳟和泥鳅的自展值有52%,如果按照自展值小于50%来表明,两个物种间的关系不稳定,那么虹鳟和泥鳅之间的进化关系应该是比较稳定的且关系较近。把虹鳟、泥鳅看作整体,与鲤鱼之间的自展值是91%,表明进化关系稳定,亲缘关系也较近。其中虹鳟、泥鳅、鲤鱼均是鱼类动物。
2.若将上述过程看作一个整体,将蓝鲸和长须鲸看作另一个整体,其中蓝鲸和长须鲸的自展值为86%,且他们在同一个分支处,表明他们亲缘关系接近。但是这两个整体的间的自展值为46%,表明他们之间的亲缘关系可能接近也可能存在差异。
3.再将上述所有物种看作一个整体,他们与西藏黄牛的自展值是46%,加上西藏黄牛,他们与人之间的自展值是100%,这表明他们与人都有一定亲缘关系。其中蓝鲸、长须鲸、人、西藏黄牛都是哺乳动物。
4.拟鳄龟和青蛙,虽然再同一分支上,但是他们的自展值却小于50%,表明他们之间的关系可能不太稳定,也具有一定亲缘关系,连同原鸡,与他们的自展值也不高。若把拟鳄龟、青蛙、原鸡看作整体,绿头鸭与他们 之间的自展值是100%,表明他们之间的关系稳定。上述涉及到的物种包括两栖动物和爬行动物。
四、总结
本研究通过分析13个生物物种的CYTB蛋白序列,构建了系统发育树,初步揭示了这些物种在进化过程中的亲缘关系。似乎鱼类动物与哺乳动物的亲缘关系近,两栖动物与爬行动物的亲缘关系更近。CYTB作为一个适中进化速率的分子标记,在探讨生物进化史方面具有重要价值。未来可进一步扩大样本量,结合其他基因序列数据,对生物进化的历史轨迹进行更加深入和全面的研究。
- Hu, Z., Yang, L., Zhang, M., Tang, H. (2024). A novel protein CYTB-187AA encoded by the mitochondrial gene CYTB modulates mammalian early development. Cell Metabolism, 发表日期:2024.4.12.
- Yao, L., Ju, X., James, T. Y., Qiu, J. Z., Liu, X. Y. (2018). Relationship between saccharifying capacity and isolation sources for strains of the Rhizopus arrhizus complex. Science Direct, 发表日期:2018.2.11.
























 1036
1036

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










