AutoDock Vina与LeDock对接口袋参数文件互转
1. AutoDock Vina与LeDock简介
AutoDock Vina 是一个用于进行分子对接的开源程序。它最初是由分子图形实验室(现为CCSB)设计和实现。与 AutoDock 4 相比,AutoDock Vina 显着提高了结合模式预测的平均准确度和对接速度。受到X-Score启发,其打分方程是一种混合函数(经验+基于知识的函数)。
LeDock是苏黎世大学ZHAO Hongtao博士开发的一款分子对接软件,在构象搜索和打分方程中均呈现出优势。LeDock采用模拟退火-遗传算法交叉的算法进行构象搜索,对接打分涵盖范德华相互作用(vdw),静电相互作用(Coulombic interaction),氢键贡献(Hbond)以及分子间(inter-)和配体分子内(intra-)的冲突(clash)几项的和做为打分方程。
2. AutoDock Vina与LeDock口袋参数
2.1. AutoDock Vina口袋参数:vian.in
receptor=5tf9_pro.pdbqt
center_x=21.408
center_y=12.224
center_z=8.102
size_x=22.374
size_y=16.942
size_z=22.009
2.2. LeDock口袋参数:dock.in
Receptor 5tf9_pro.pdb
RMSD
1.0Binding pocket
11.67 33.078
10.83 23.054
17.958 26.06Number of binding poses 20
Ligands list ligands
END
2.3 口袋选取原理(基于配体)
以小分子配体为中心,向外扩展一定距离(extending)形成的口袋位置。首先获取小分子的坐标后取得X/Y/Z的最大(max)、最小值(min),然后用最小值减去扩展距离、最大值加扩展距离。LeDock对接软件的口袋参数文件如2.2中所示,在Binding pocket行后的三行中,以最终加减过扩展距离后X/Y/Z各自的“最小值 最大值”定义了最终的口袋大小。而AutoDock Vina是在此基础上对最大值和最小值进行换算获得最终的Size和Center值。如下面图片来自GetBox-PyMOL-Plugin。
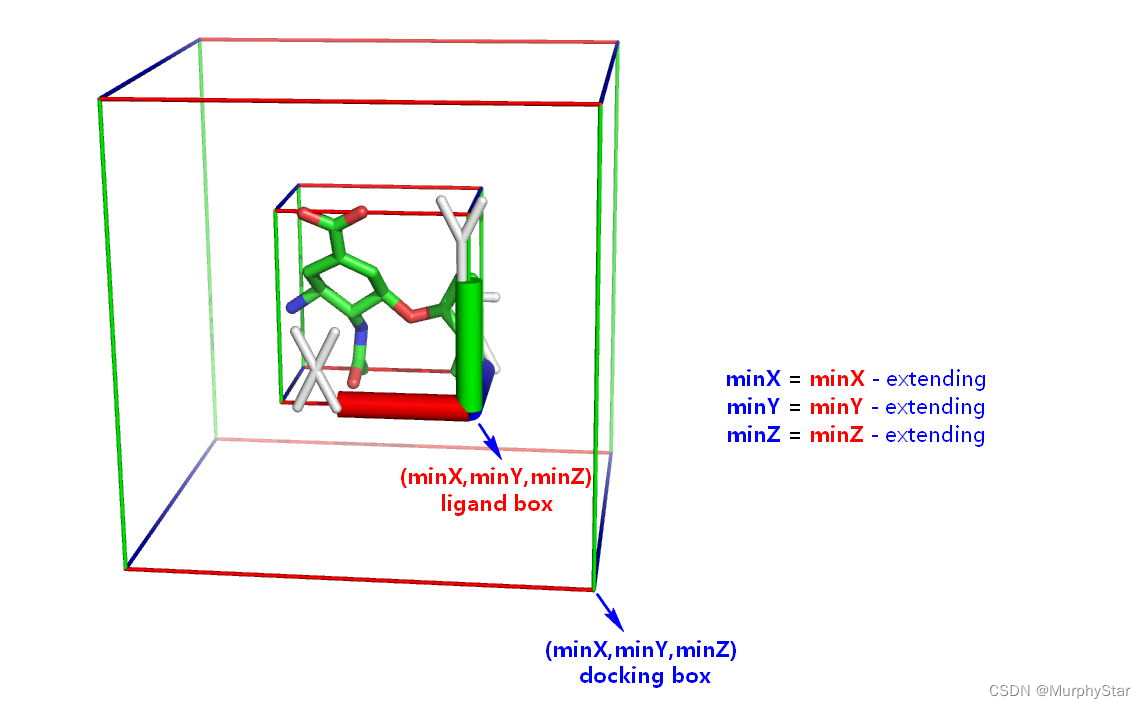
a. LeDock对接口袋计算原理
minX = minX - float(extending)
minY = minY - float(extending)
minZ = minZ - float(extending)
maxX = maxX + float(extending)
maxY = maxY + float(extending)
maxZ = maxZ + float(extending)
b. AutoDock Vina对接口袋计算原理
SizeX = maxX - minX
SizeY = maxY - minY
SizeZ = maxZ - minZ
CenterX = (maxX + minX)/2
CenterY = (maxY + minY)/2
CenterZ = (maxZ + minZ)/2
3. AutoDock Vina口袋参数文件转换为LeDock
3.1 转化命令
python vina2ledock.py vina.in
3.2 转化脚本vina2ledock.py
import sys
with open(sys.argv[1], 'r') as f:
pocket = f.readlines()
receptor_QT=pocket[0].split('=')[1]
receptor=receptor_QT.split('qt')[0]
center_x=pocket[1].split('=')[1]
center_y=pocket[2].split('=')[1]
center_z=pocket[3].split('=')[1]
size_x=pocket[4].split('=')[1]
size_y=pocket[5].split('=')[1]
size_z=pocket[6].split('=')[1]
f.close()
max_x=float(center_x)+float(size_x)/2
min_x=float(center_x)-float(size_x)/2
max_y=float(center_y)+float(size_y)/2
min_y=float(center_y)-float(size_y)/2
max_z=float(center_z)+float(size_z)/2
min_z=float(center_z)-float(size_z)/2
with open('./dock.in','w+') as file_out:
print('Receptor\n{}'.format(receptor), file=file_out)
print('\nRMSD\n1.0\n\nBinding pocket', file=file_out)
print('{}'.format(round(min_x,3)), '{}'.format(round(max_x,3)), file=file_out)
print('{}'.format(round(min_y,3)), '{}'.format(round(max_y,3)), file=file_out)
print('{}'.format(round(min_z,3)), '{}'.format(round(max_z,3)), file=file_out)
print('\nNumber of binding poses\n20\n\nLigands list\nligands\n\nEND', file=file_out)
file_out.close()
4. LeDock口袋参数文件转换为AutoDock Vina
4.1 转化命令
bash docktovian.sh dock.in
4.2 转化脚本docktovian.sh
#!/bin/bash
rcpt=`cat $1 |grep -i -A 1 "Receptor"`
rcpt_name=`echo ${rcpt}|awk {'print $2'}`
echo "receptor = " ${rcpt_name}qt
bdst=`cat $1 |grep -i -A 3 "Binding pocket"`
min_x=`echo ${bdst}|awk '{print $3}'`
max_x=`echo ${bdst}|awk '{print $4}'`
min_y=`echo ${bdst}|awk '{print $5}'`
max_y=`echo ${bdst}|awk '{print $6}'`
min_z=`echo ${bdst}|awk '{print $7}'`
max_z=`echo ${bdst}|awk '{print $8}'`
center_x_1=`expr ${max_x%.*} + ${min_x%.*}`
center_x=`expr ${center_x_1} / 2`
echo center_x = ${center_x}
center_y_1=`expr ${max_y%.*} + ${min_y%.*}`
center_y=`expr ${center_y_1} / 2`
echo center_y = ${center_y}
center_z_1=`expr ${max_z%.*} + ${min_z%.*}`
center_z=`expr ${center_z_1} / 2`
echo center_z = ${center_z}
size_x=`expr ${max_x%.*} - ${min_x%.*}`
echo size_x = ${size_x}
size_y=`expr ${max_y%.*} - ${min_y%.*}`
echo size_y = ${size_y}
size_z=`expr ${max_z%.*} - ${min_z%.*}`
echo size_z = ${size_z}


























 3779
3779

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








