微生物群落研究中,扩增子测序技术就是针对微生物中具有物种特异性序列进行测序。细菌群落的研究更多关注的是16S rRNA的序列,这段序列保守区夹杂着9个高变区构成(下图),不同物种的高变区具有较大差异,因此可作为物种识别的特征性序列开展研究。

相信主攻微生物领域研究的老师们,一定纠结过到底测细菌16S的哪个区,V3+V4,或者V4+V5?细菌的16S一共有V1-V9九个区,全长约1500+bp,受限于NGS的读长,只能对其中的个别高变区域开展,在属水平进行物种注释,而都无法将其准确注释到种水平。
有没有方法能够测得16S全长的同时兼顾测序准确率、通量和价格呢?
伴随着三代测序技术的发展,长读长测序技术越来越成为组学研究的主流。PacBio平台3-5kb的平均读长可以轻而易举地覆盖16S rRNA的9个高变区域,16S rRNA全长测序因此成为多样性研究的新热点技术,具有许多明显的优势:
1、三代16sr RNA全长扩增子长读长
PacBio全长16S rRNA基因测序采用27F-1492R引物扩增全长片段(覆盖V1-V9区),采用PacBio SMRT测序平台CCS(Circular Consensus Sequencing)模式进行测序分析,获得的序列更长,信息量更多更准确!

2、三代16s rRNA全长物种鉴定高精准
HiFi reads(High Fidelity reads)是2019年由PacBio推出的基于环化共有序列(Circular Consensus Sequencing,CCS)模式产生的既兼顾长读长(10-20kb)又具有高精度(>99%准确率)的测序结果。PacBio数据下机后,使用仪器自带的SMRTLINK(v9)进行一致性CCS序列(circular consensus sequencing)的获得,CCS序列的准确性达到QV20(99%准确率)水平。

由此可见,微生物16S rRNA三代全长HiFi扩增子兼顾了二代测序准确率、长读长以及数据高通量,对于微生物群落物种鉴定更加灵敏、准确!
从分类鉴定的状况看,基于三代测序,在同一分类水平下,也可以实现更多的物种分类,更是在“种”水平物种鉴定中实现了大幅提升,减少了很多在种属水平上的错误物种分类。这一点就很大程度满足了微生物学研究者的迫切需求。

3、三代16S rRNA全长PCR扩增无GC偏好性
由于二代测序建库过程中有PCR扩增的步骤,难以避免由于扩增偏好性造成的物种丰度检测的偏差。在多个研究中,人们都发现基于二代测序部分高变区的研究,可能造成某些特定微生物类群含量的检测偏差(下图),这就难以实现对样本中微生物群落分布的真实还原。而三代测序由于不需要PCR的过程,无GC偏好性,就很大程度上降低了结果的偏差,数据结果更准确!
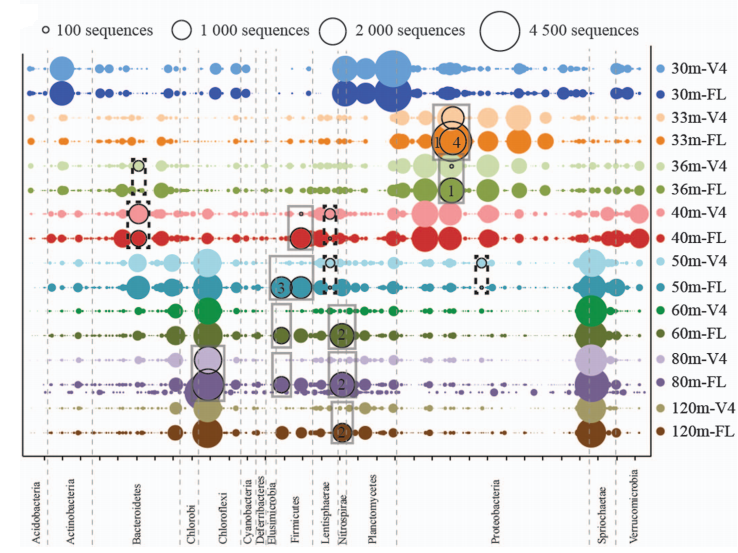
4、三代16S全长能够更真实的还原微生物群落结构
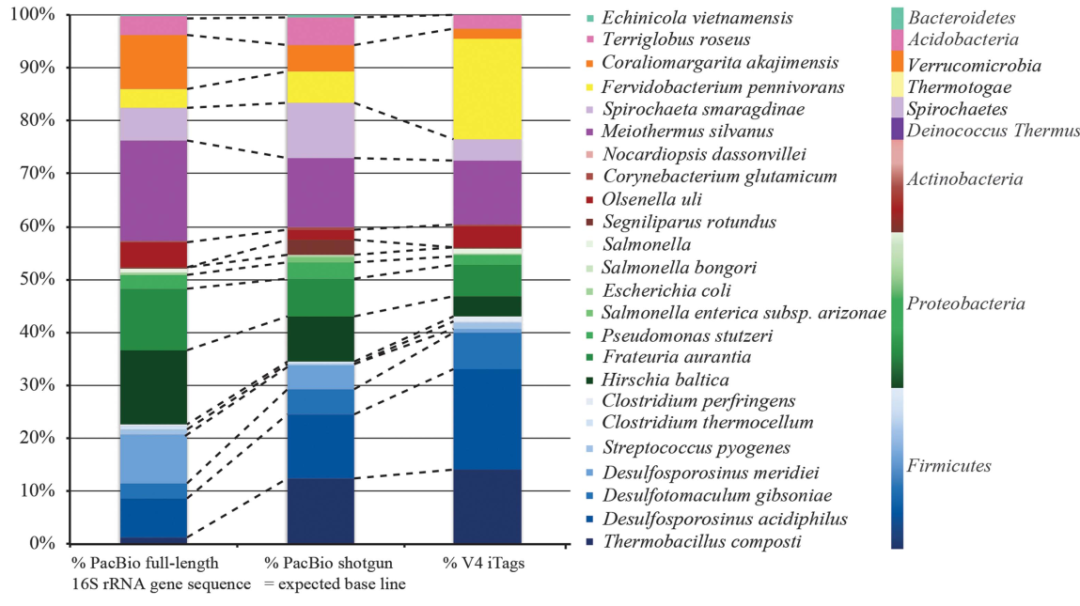
2019年有学者在《Nature Communications》期刊报道了基于16S不同高变区和16S全长研究的差异。基于数据库已有信息和计算模拟,评价了不同区域引物在物种识别和还原样本信息上的差异。

下图展示的是扩增子序列不能被识别的序列比例。不难看出V1-V9区引物很少有序列不能被明确分类,相比之下,仅仅开展V4区的研究,则有极高比例的序列无法实现明确分类。

基于模式物种库进行进化树构建,并用红色表示其中不能被明确鉴定的物种,亦可看出,基于部分区域的V4区研究,有很大比例的物种无法实现明确分类。此外,由于研究区域的选择,在一定程度上也造成了分类识别的偏好性。
通过聚类OTU,并与模拟物种库进行比较,研究发现,V1-V9区研究实现了对样本的最佳还原。此外,基于三代技术开展16S全长扩增子研究,有更多的序列得到了明确的物种划分,尤其是针对样本中含量低微的物种,分类鉴定的效果更好。

三代16s rRNA全长扩增子测序能够提高微生物组成鉴定精确度,更真实还原样本微生物群落结构。更长,更稳,更精准!
总之,与二代测序相比,三代PacBio平台可满足16S全长研究需要,特别是PacBio HiFi测序同时兼具通量高、测序时间短、准确性高、无需扩增等优势,伴随着三代测序成本的下降,势必将取代二代测序,成为环境、医口研究领域高分科研论文的不二之选!
重磅好消息来咯!
凌恩生物6.18聚惠来袭!!
三代16S全长测序一万条以上序列仅需249元,三万条以上序列仅需299元
低价赶超二代扩增子研究,有没有很心动呢!
再也不必纠结9个高变区该怎么选,
16s全长扩增子,更精确,更灵敏,更保真!
更有凌恩生物特惠支持,再也不用焦头烂额,研究烦恼统统解决!

欢迎参与活动的老师同学联系当地销售或微信公众号后台留言!
参考文献
1. High-resolution Phylogenetic Microbial Community Profiling. The ISME Journal, 2016. doi: 10.1038/ismej.2015.249
2. Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis. Nature Communications, 2019. doi: 10.1038/s41467-019-13036-1




























 778
778

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










