Yanglan Gan, Xingyu Huang, Guobing Zou, Shuigeng Zhou, Jihong Guan, Deep structural clustering for single-cell RNA-seq data jointly through autoencoder and graph neural network, Briefings in Bioinformatics, Volume 23, Issue 2, March 2022, bbac018, Deep structural clustering for single-cell RNA-seq data jointly through autoencoder and graph neural network | Briefings in Bioinformatics | Oxford Academic
代码地址:DHUDBlab/scDSC (github.com)
摘要
单细胞RNA测序(scRNA-seq)可以研究细胞异质性和多样性的复杂机制。无监督聚类对于分析scRNA-seq数据至关重要,因为它可以用于识别潜在的细胞类型。然而,由于噪声影响、高维度和普遍的缺失事件,scRNA-seq数据的聚类分析仍然是一个计算挑战。本文提出了一种新的用于scRNA-seq数据的深度结构聚类方法,命名为scDSC,它将结构信息整合到单细胞的深度聚类中。所提出的scDSC包括基于零膨胀负二项式(ZINB)模型的自编码器,图神经网络(GNN)模块和相互监督模块。为了从稀疏和零膨胀的scRNA-seq数据中学习数据表示,将ZINB模型添加到基本自编码器(AE)中。引入GNN模块以捕获细胞之间的结构信息。通过将基于ZINB的自编码器与GNN模块相结合,模型将自动编码器学习到的数据表示转移到相应的GNN层。此外,采用相互监督策略来统一这两种不同的深度神经架构,并引导聚类任务。
模型图
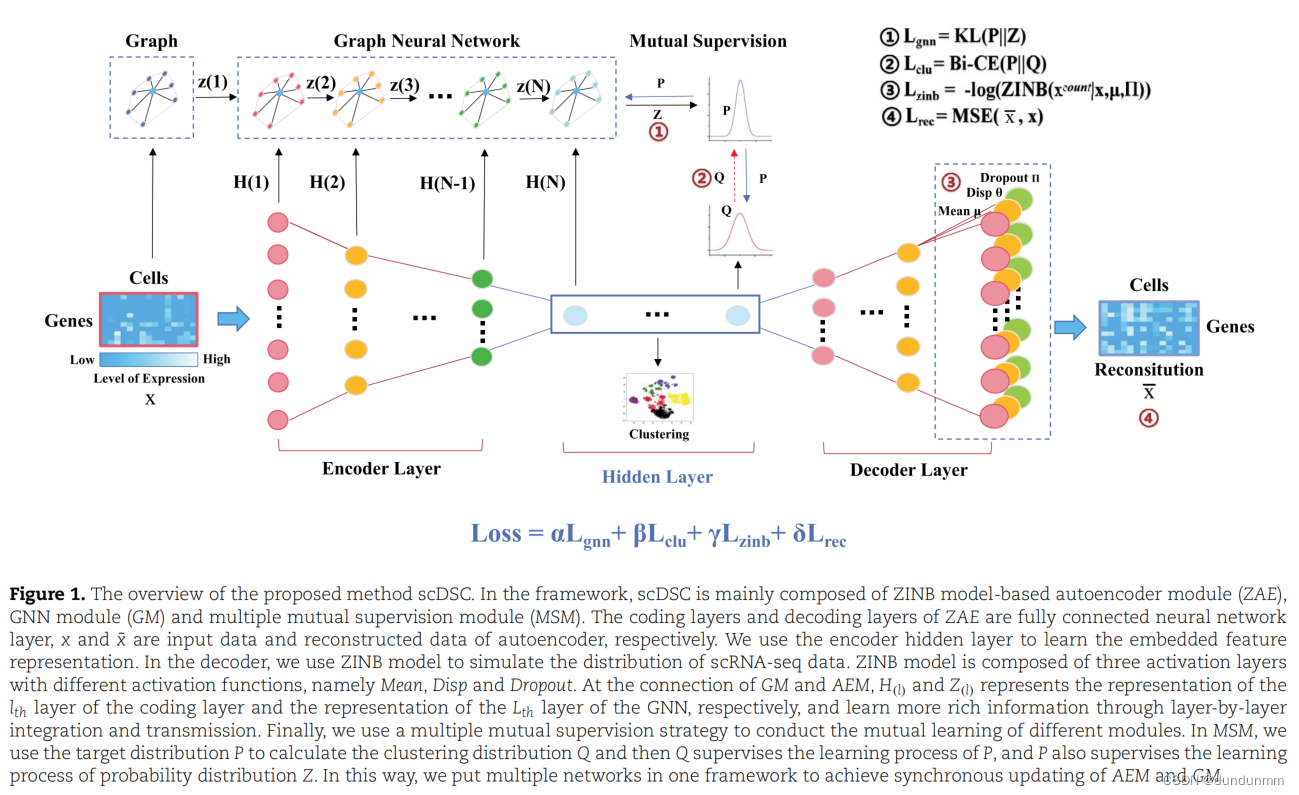
看过SDCN模型的同学,可以发现这一篇的思路就是将SDCN模型用于单细胞RNA测序任务。为了适应单细胞RNA数据的复杂分布,将常规的AE改为基于ZINB的AE。
论文的写法也与SDCN论文类似。如果想把深度聚类模型融入该领域中,可以借鉴一下该文的切入角度和实验角度。






















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








