根据GEO的GSE数据集编号自动下载和处理GEO数据教程
NCBI GEO网站介绍
NCBI GEO数据库(Gene Expression Omnibus)是一个全球最大的生物医学领域的公共数据库平台,用于存储基因组学数据,包括基因表达数据、染色质状态和基因组变异等。研究人员可以在该平台上查找、下载和分析各种基因表达数据集,以便进行生物信息学和生物医学研究。NCBI GEO数据库提供了丰富的数据资源和分析工具,是生命科学领域的重要研究工具之一。大量生物医学领域发表的论文使用的公共数据集也一般是来自GEO数据库的数据集。
GEO数据库中的数据集按照一系列预定义的格式和标准进行提交和组织,主要包括以下几种格式:
- GSE(Gene Series):表示一个或多个实验的数据集,通常包含多个样本。
- GSM(Sample Series):表示单个实验中的一个样本。
- GPL(Platform):描述用于实验的数据采集平台的详细信息,包括探针序列、实验条件等。
- GDS(Series Matrix File):包含一个GSE或GSM数据集的详细矩阵表格,其中行表示基因,列表示样本,单元格中的值表示基因在样本中的表达水平。
配置aria2c高效下载支持断点续传的下载神器来加速GEO的数据下载
该部分的教学视频链接
Aria2c软件下载
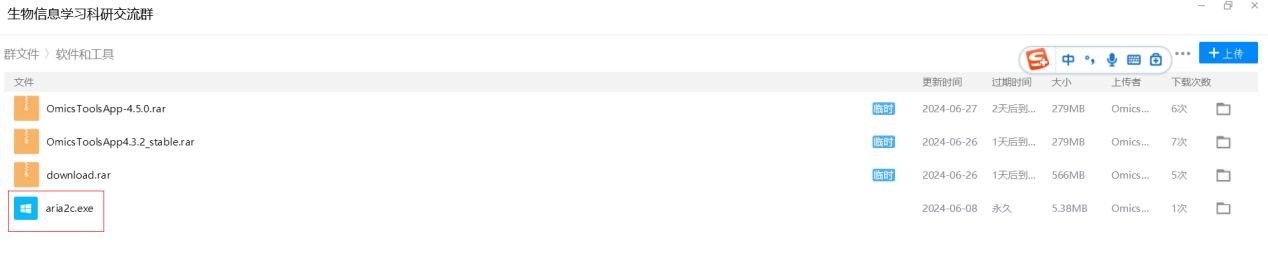
在D:/omics_tools目录下创建一个aria2的目录,从我的生信q群931846486里下载aria2c.exe软件包到D:/omics_tools/aria2目录下就可,不需要安装,只需要把这个aria2c.exe放到该目录就行了。
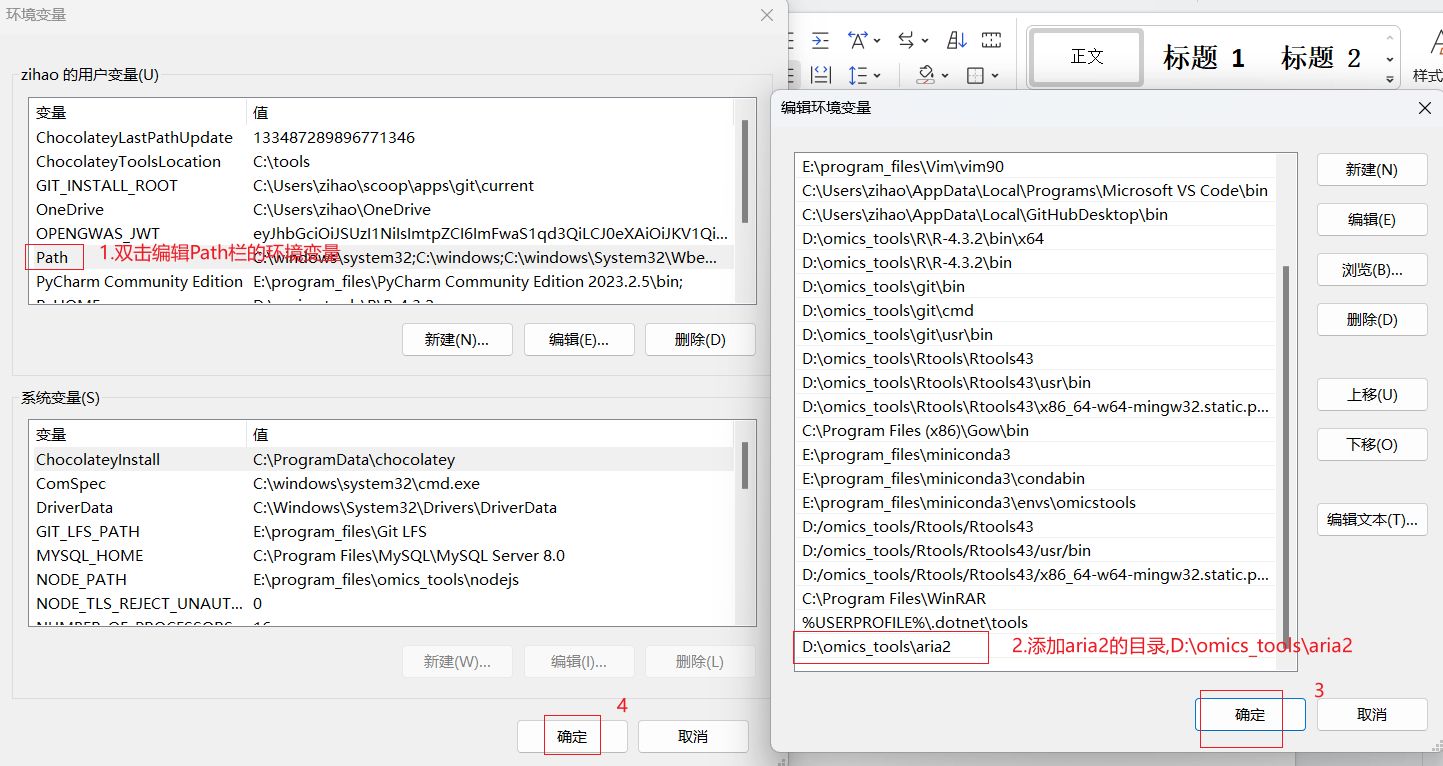
配置aria2c的环境变量
环境变量的配置示意图
检测aria2c的环境变量是否配置成功
新打开一个cmd命令提示符,输入which aria2c, 看看是否会返回aria2c的路径
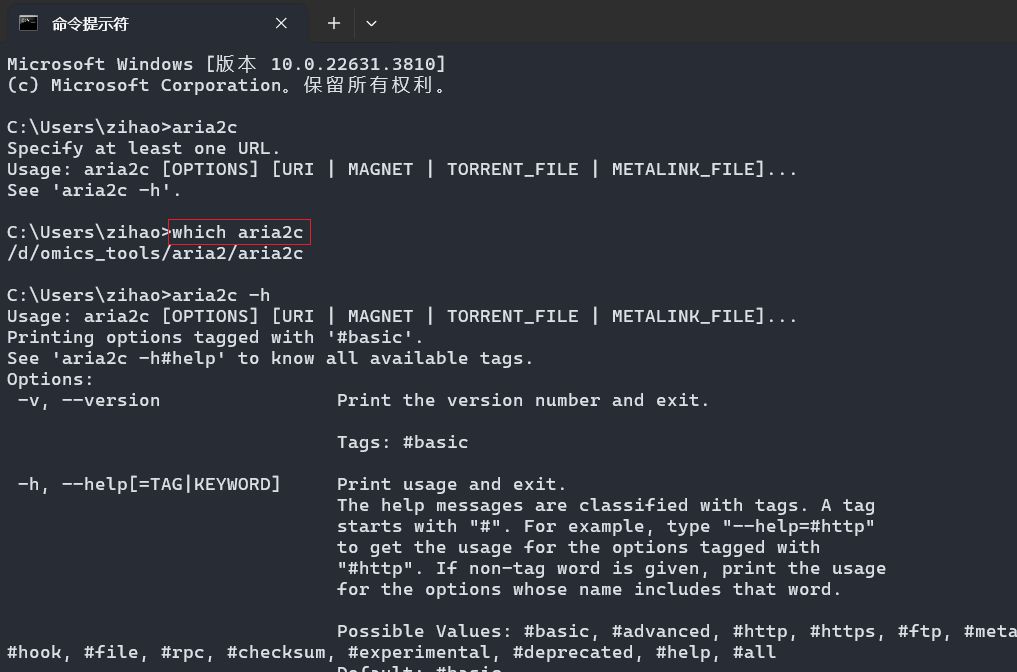
如果成功返回了aria2c的路径,至此,aria2c下载工具就配置完成了。
根据GSE id自动下载处理GEO数据(必须要运行的模块)
该模块的教学视频链接
- 根据GSE id自动下载处理GEO数据:根据GSE编号自动下载处理GEO数据提取出表达矩阵和样本分组信息_哔哩哔哩_bilibili根据GSE编号自动下载处理GEO数据提取出表达矩阵和样本分组信息, 视频播放量 376、弹幕量 0、点赞数 4、投硬币枚数 2、收藏人数 15、转发人数 2, 视频作者 邢博士谈科教, 作者简介 OmicsTools 生信电脑软件作者,每晚11点在线直播论文复现教学,提供生信个性化分析科研作图服务,淘宝店:生信研究,相关视频:根据GSE id和文件名构建下载链接批量下载和处理GEO数据,单细胞基本分析流程学习总结,第四课:单细胞通讯分析汇总,2024第三课:单细胞CNV分析及CNV聚类,现在还能收纯生信的SCI杂志太难得了,综述、生信强烈建议先码住!,11(附代码)GTEx数据库下载与整理,GTEx合并TCGA分析。保姆级教程,一键出图,R语言各种数据类型相互转换操作教程讲解,nature发表的文章润色指令,非常好用!,本人医学博士已发15篇sci,最近有空想带几个徒弟,学习生信分析!,肺腺癌2-文献复现流程
 https://www.bilibili.com/video/BV1HE421G7cD
https://www.bilibili.com/video/BV1HE421G7cD - GEO分析流程视频讲解:GEO数据的下载和各种处理挖掘作图全流程分析教程讲解_哔哩哔哩_bilibiliGEO数据的下载和各种处理挖掘作图全流程分析教程讲解, 视频播放量 113、弹幕量 0、点赞数 3、投硬币枚数 0、收藏人数 5、转发人数 1, 视频作者 邢博士谈科教, 作者简介 OmicsTools 生信电脑软件作者,每晚11点在线直播论文复现教学,提供生信个性化分析科研作图服务,淘宝店:生信研究,相关视频:读取数据目录下的idat文件的甲基化全流程一键分析,XRD数据处理-Jade分析、Origin堆积图、PDF卡片导出与插入,43单细胞分析,细胞聚类和注释,细胞轨迹分析,关键基因标注,【教程】4907血浆蛋白孟德尔随机化分析,正反双向本地芬兰数据,骨肉瘤为例,11(附代码)GTEx数据库下载与整理,GTEx合并TCGA分析。保姆级教程,一键出图,第四课:单细胞通讯分析汇总,2024第三课:单细胞CNV分析及CNV聚类,R语言各种数据类型相互转换操作教程讲解,9+神仙二区Top期刊!干湿结合单基因思路,学到就是赚到,CAD2024下载+安装+激活带字幕版详细教程(附带下载链接)
 https://www.bilibili.com/video/BV18r421A7Po
https://www.bilibili.com/video/BV18r421A7Po
分析参数详细解释
func_gse__id:要下载的GSE数据集
nested_function:是否嵌套函数
run_file_path: 数据要下载到的目录
run_read_file:是否要读取文件
run_add_save_file_prefix:是否要添加结果保存文件的前缀
提交
参数指定的默认值
func_gse__id: GSE61763 ;
nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/res_dir/renal_cancer ;
run_read_file: FALSE ;
run_add_save_file_prefix: FALSE
窗口截图

窗口截图
运行中的显示信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir\res_dir; 运行结果日志保存的路径为: D:/omics_tools/demo_data/res_dir\res_dir\renal_cancer_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir/renal_cancer; 分析结果日志保存的路径为: D:/omics_tools/demo_data/res_dir/renal_cancer\renal_cancer_last_final_run_res_log.csv
下载整理到的结果文件展示
结果文件列表
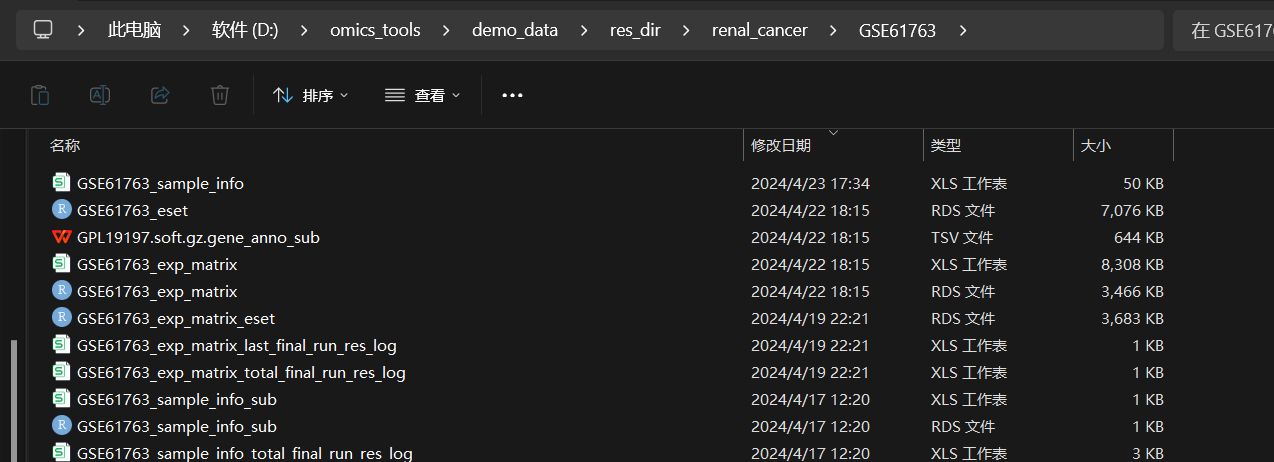
结果文件列表
样本注释文件信息
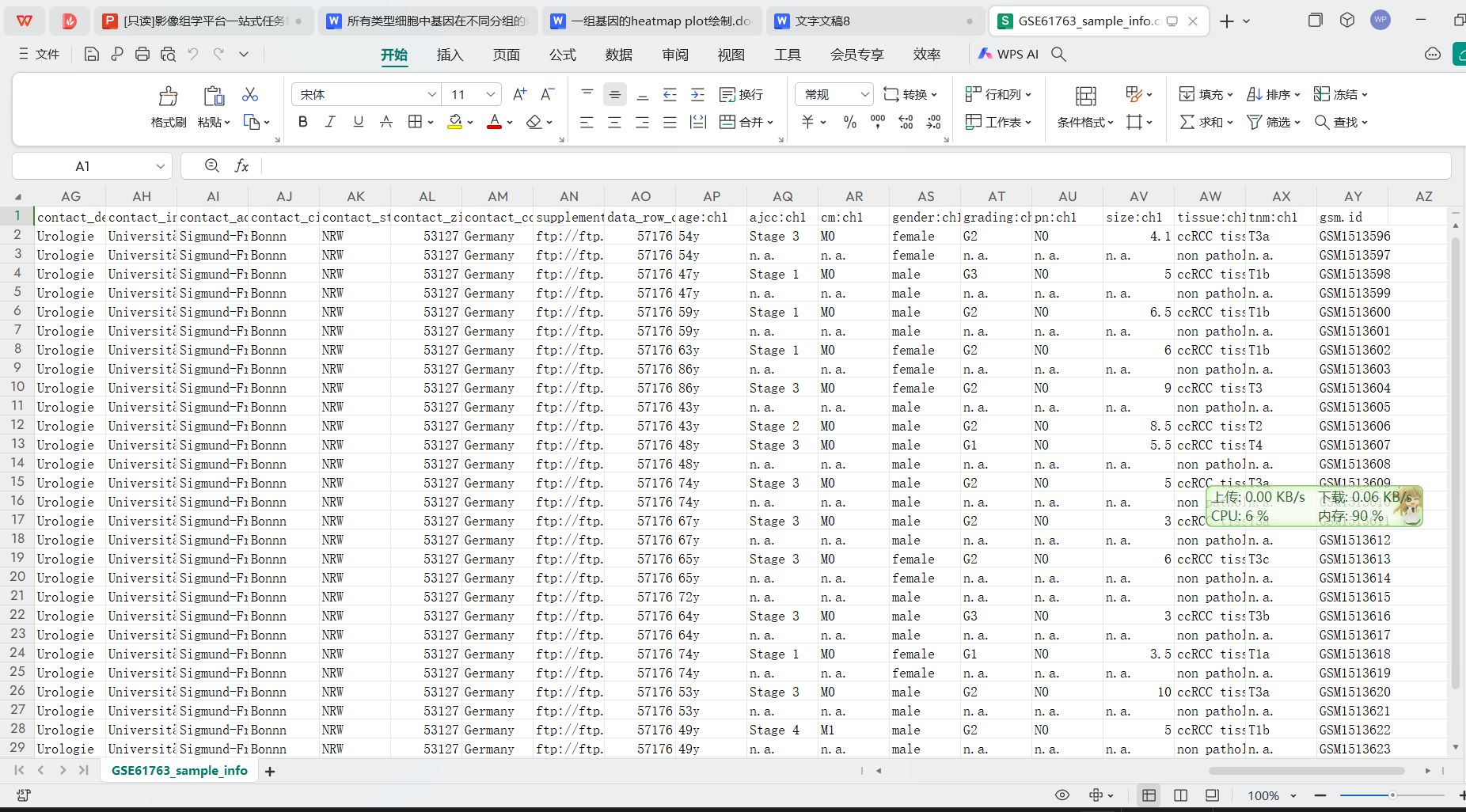
样本的注释信息文件
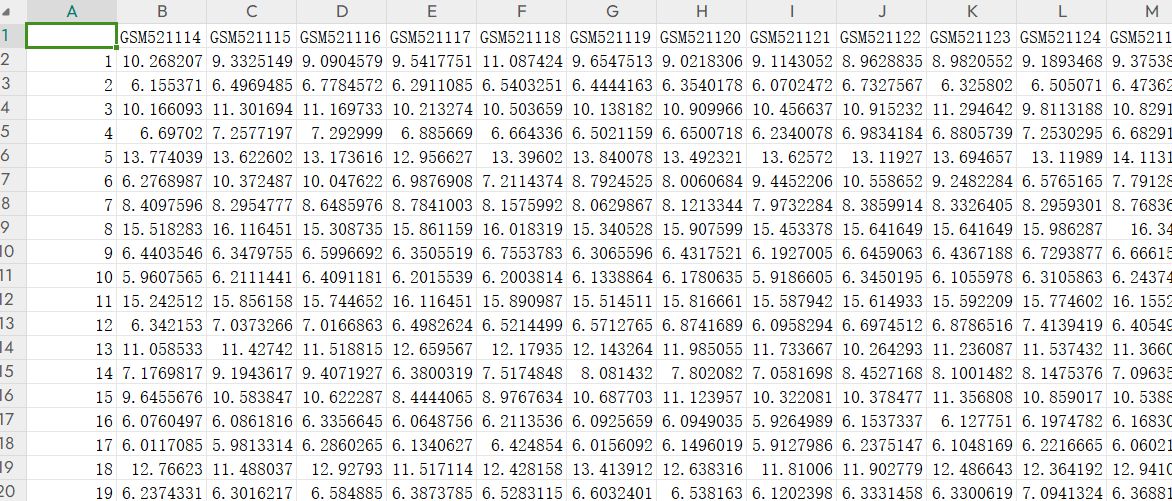
基因表达矩阵
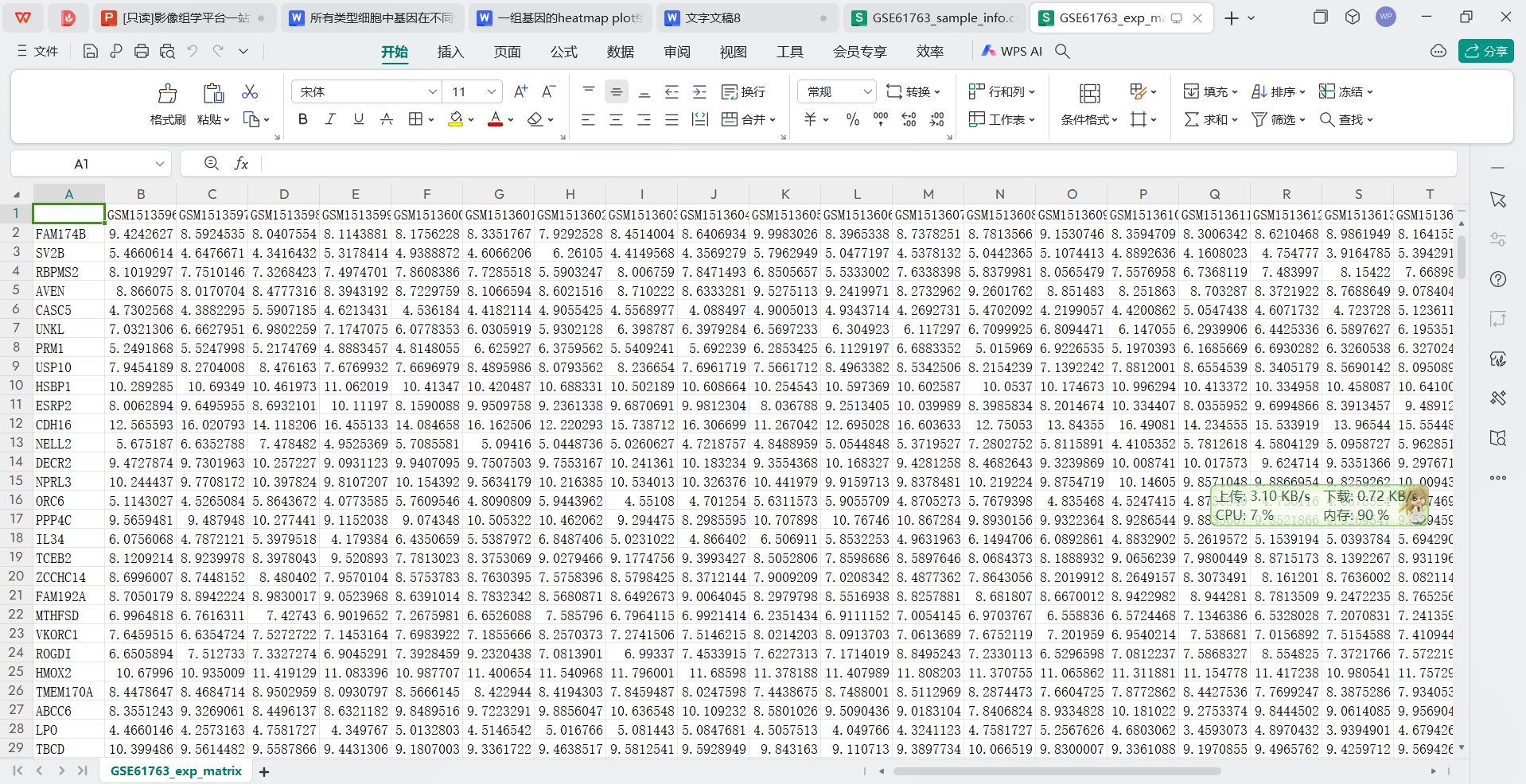
基因表达矩阵文件
该根据GSE id自动下载处理GEO数据的GEO下载模块的常见问题答疑详解
GEO的数据集格式多种多样,使用该模块下载GEO数据集最好的情况下得到的结果是什么样的?
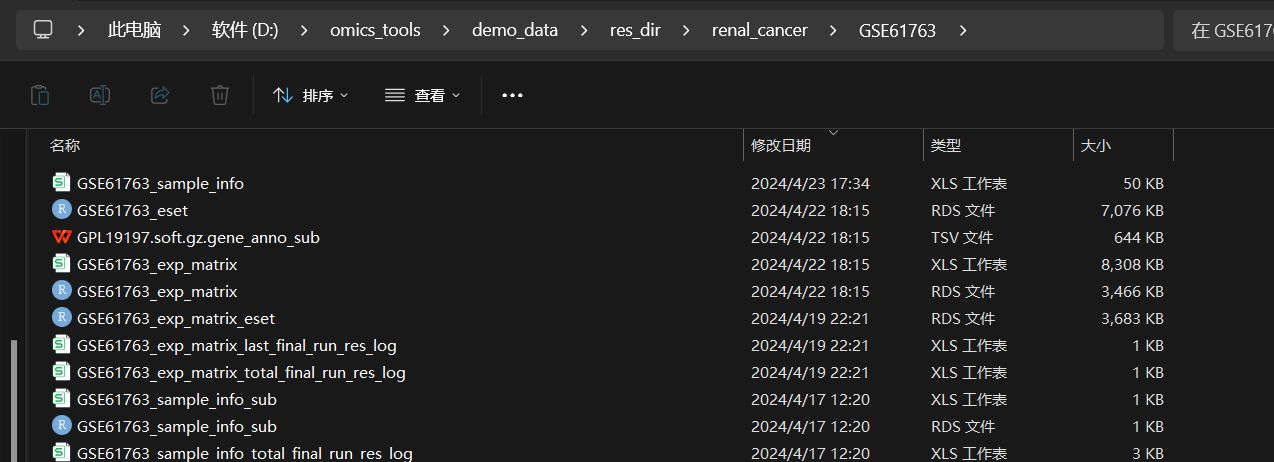
最好的结果就是下图这样,直接通过该模块点击下载后,会得到下面这些下载提取整理好的文件:
- 一次性得到注释好基因名称的表达矩阵文件,如GSE61763_exp_matrix.csv,GSE61763_exp_matrix.rds
- 得到样本的注释信息文件: 如GSE61763_sample_info.csv,
- 得到从GPL soft注释文件中提取到的基因探针跟基因名称的对应关系的两列数据文件,如GPL191197.soft.gz.gene_anno_sub.tsv文件
- 得到了使用表达矩阵,基因注释信息,样本注释信息等数据构建的ExpressionSet对象格式文件,如GSE61763_eset.rds 文件
结果文件列表
GEO的数据集格式多种多样,使用该模块下载GEO数据集第二好的情况下得到的结果是什么样的?遇到这种情况该怎么处理?
下载的结果文件列表
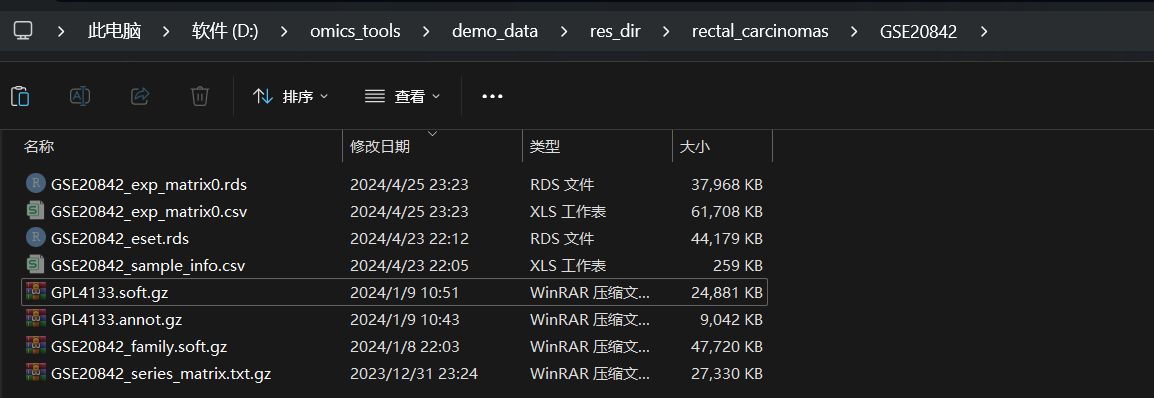
- 提取到了GEO数据集的表达矩阵文件,如GSE20842_exp_matrix0.csv,GSE20842_exp_matrix0.rds
- 得到样本的注释信息文件: 如GSE20842_sample_info.csv,
- 得到了使用表达矩阵,探针信息,样本注释信息等数据构建的ExpressionSet对象格式文件,如GSE20842_eset.rds 文件‘
- 但是这个表达矩阵有个问题就是,行名不是基因名称,而是探针名称,所以我对这个文件名称做了一些修饰,如果文件名后缀是exp_matrix0.csv,那么这样的表达矩阵里面基本上是没有注释好基因名称,需要在手动下载一下GEO的GPL文件从GPL文件中提取出基因的注释信息再跟表达矩阵合并,这样的处理作为我也有详细的处理教程来教大家怎么做,如果文件后缀就是exp_matrix.csv,那么就是已经成功把基因名称提取出来并整合到表达矩阵中了。对于没有基因名称只有基因探针的表达矩阵,可以用下面这两个模块进行处理:
GEO的数据集格式多种多样,使用该模块下载GEO数据集得到的文件比较差的情况是什么样的?遇到这种情况该怎么处理?
比如像这个例子中,只下载到了GSE158055_sample_info.csv这样的样本分组注释信息文件,这种GEO的GSE数据集一般都是没有提供有效的series_matrix文件,导致没法使用GEO下载工具下载,只能进入该GSE数据集的GEO网页下进行手动下载表达数据文件或一个GSEXXX_RAW.tar的压缩包文件。
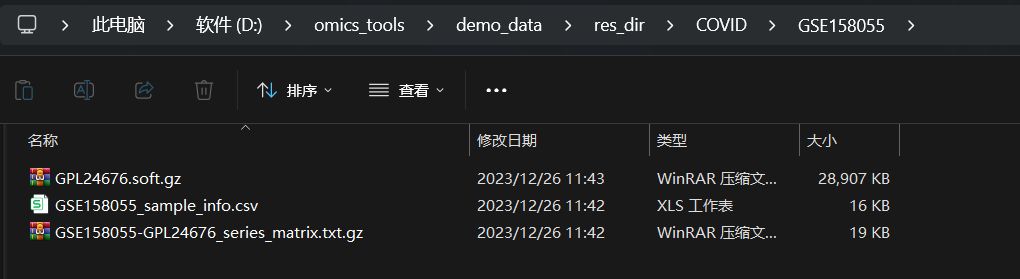
我这里有四个模块可以对转录组或各种平台的基因芯片的RAW.tar压缩包内的多样本表达数据进行整合和处理。

2.根据多个GSE id自动批量下载处理GEO数据
如果大家一次要下载很多个GSE数据集的数据的话,可以使用这个模块进行批量下载多个GSE数据集中的数据。
该模块的教学视频链接
根据多个GSE id自动批量下载处理GEO数据:根据GSE id自动批量下载处理GEO数据_哔哩哔哩_bilibili
根据GSE id自动批量下载处理GEO数据
分析参数详细解释
func_write_download_status:将下载和数据处理是否成功的信息写入到file_path中,默认是TRUE
nested_function:是否嵌套函数
run_file_path:要自动批量下载和处理的带有gse_id列表的文件路径,例如:D:/omics_tools/demo_data/gse_list_info.csv
run_read_file:是否要读取文件
run_add__res__dir:是否要新建结果目录
run_add_save_file_prefix:是否要添加结果保存文件的前缀
run_add__parent__dir:是否在上一级目录下创建目录或保存结果
提交
参数给定的默认值
func_write_download_status: TRUE ;
nested_function: TRUE ;
run_file_path: D:/omics_tools/demo_data/gse_list_info.csv ;
run_read_file: FALSE ;
run_add__res__dir: FALSE ;
run_add_save_file_prefix: FALSE ;
run_add__parent__dir: FALSE
输入文件信息
窗口截图
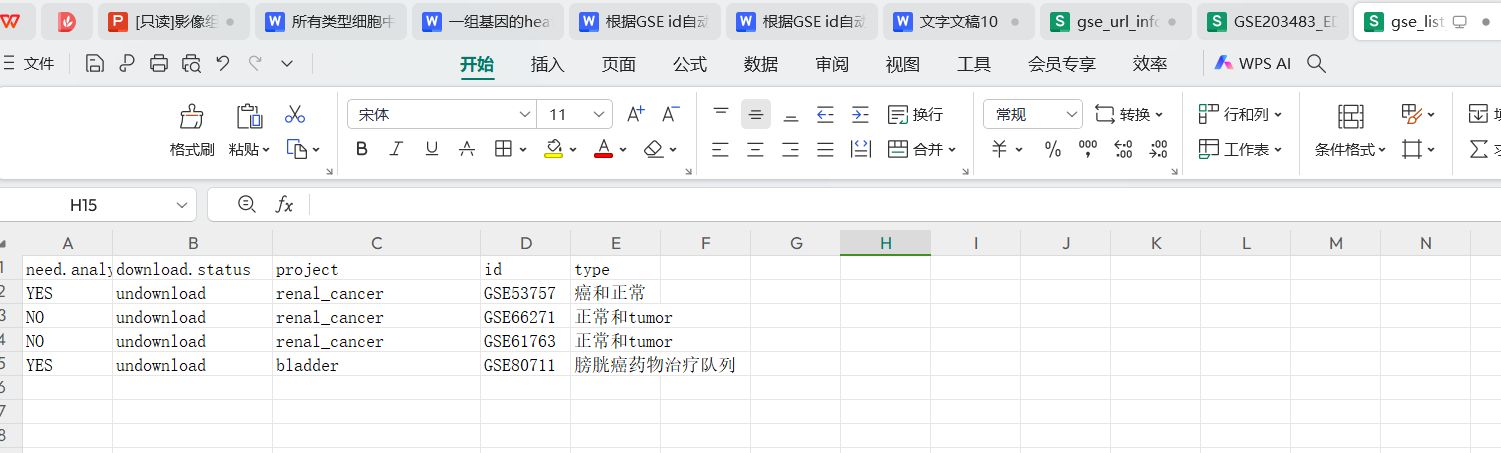
窗口截图
运行中的显示信息
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data; 分析结果日志保存的路径为: D:/omics_tools/demo_data\gse_list_info_last_final_run_res_log.csv
运行已完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data; 分析结果日志保存的路径为: D:/omics_tools/demo_data\gse_list_info_last_final_run_res_log.csv
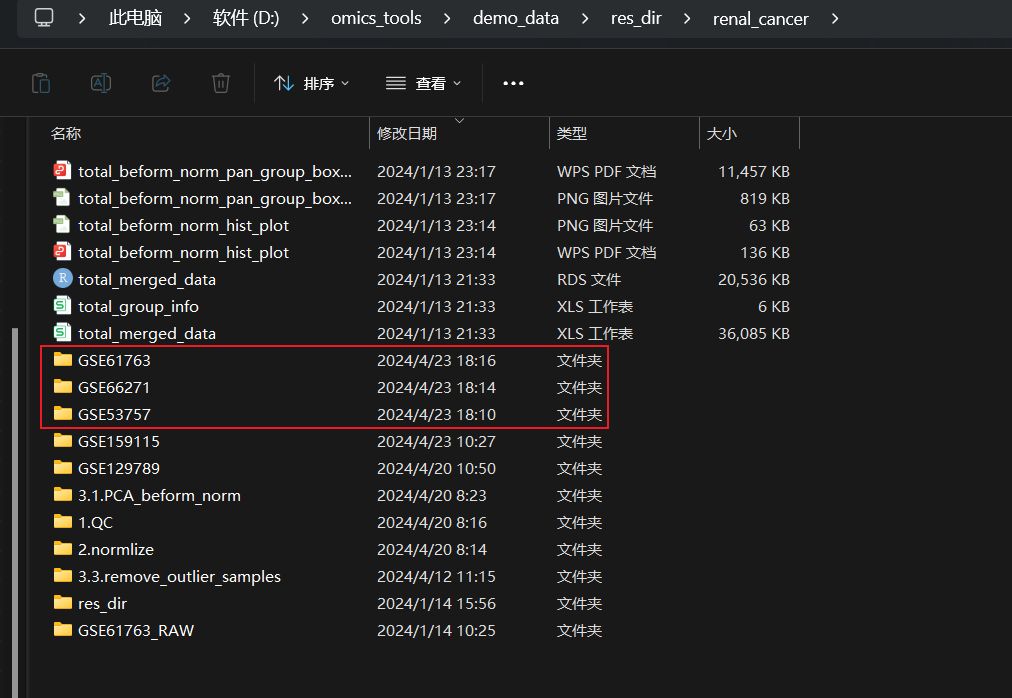
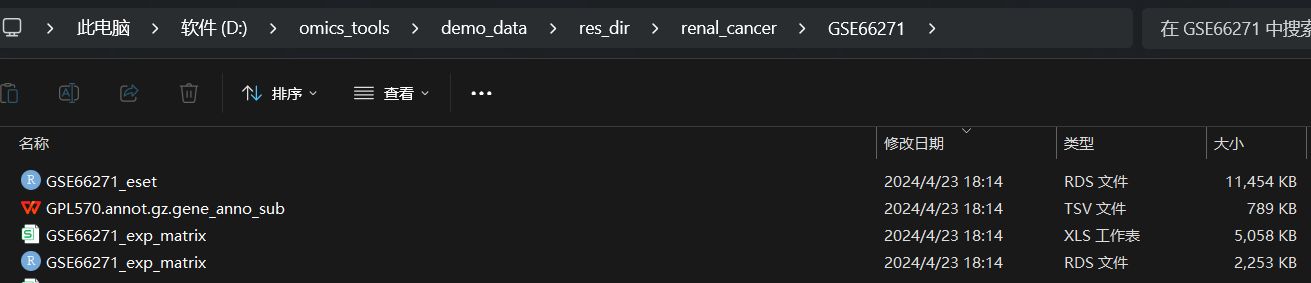
下载结果展示
结果文件列表




























 5192
5192











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










