GEO上所有数据集数据的下载和提取的最详细教程
0. GEO数据库讲解和准备
NCBI GEO网站介绍
NCBI GEO数据库(Gene Expression Omnibus)是一个全球最大的生物医学领域的公共数据库平台,用于存储基因组学数据,包括基因表达数据、染色质状态和基因组变异等。研究人员可以在该平台上查找、下载和分析各种基因表达数据集,以便进行生物信息学和生物医学研究。NCBI GEO数据库提供了丰富的数据资源和分析工具,是生命科学领域的重要研究工具之一。大量生物医学领域发表的论文使用的公共数据集也一般是来自GEO数据库的数据集。
GEO数据库中的数据集按照一系列预定义的格式和标准进行提交和组织,主要包括以下几种格式:
1.GSE(Gene Series):表示一个或多个实验的数据集,通常包含多个样本。
2.GSM(Sample Series):表示单个实验中的一个样本。
3.GPL(Platform):描述用于实验的数据采集平台的详细信息,包括探针序列、实验条件等。
4.GDS(Series Matrix File):包含一个GSE或GSM数据集的详细矩阵表格,其中行表示基因,列表示样本,单元格中的值表示基因在样本中的表达水平。
配置aria2c高效下载支持断点续传的下载神器来加速GEO的数据下载
为了在国内也能稳定下载GEO的数据,必须要配置一下aria2c这个下载神器工具。OmicsTools的GEO数据下载模块会调用aria2c来实现在国内对美国的NCBI GEO数据库上的数据集进行文档下载和断点续传。

aria2c配置的教学视频链接

Aria2c软件下载
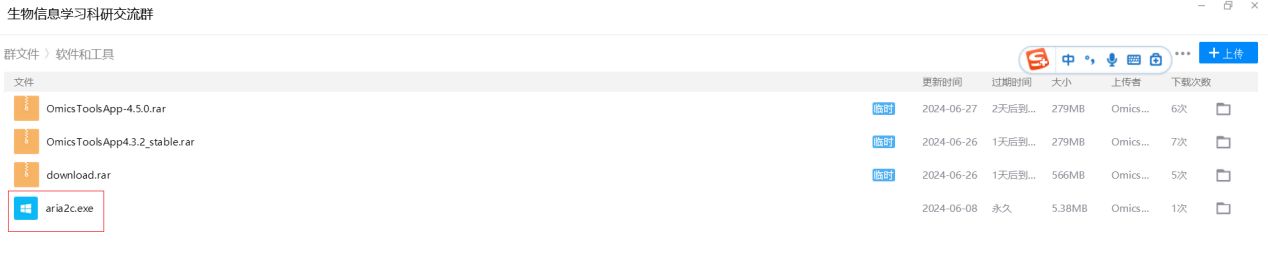
在D:/omics_tools目录下创建一个aria2的目录,从我的生信群里下载aria2c.exe软件包到D:/omics_tools/aria2目录下就可,不需要安装,只需要把这个aria2c.exe放到该目录就行了。
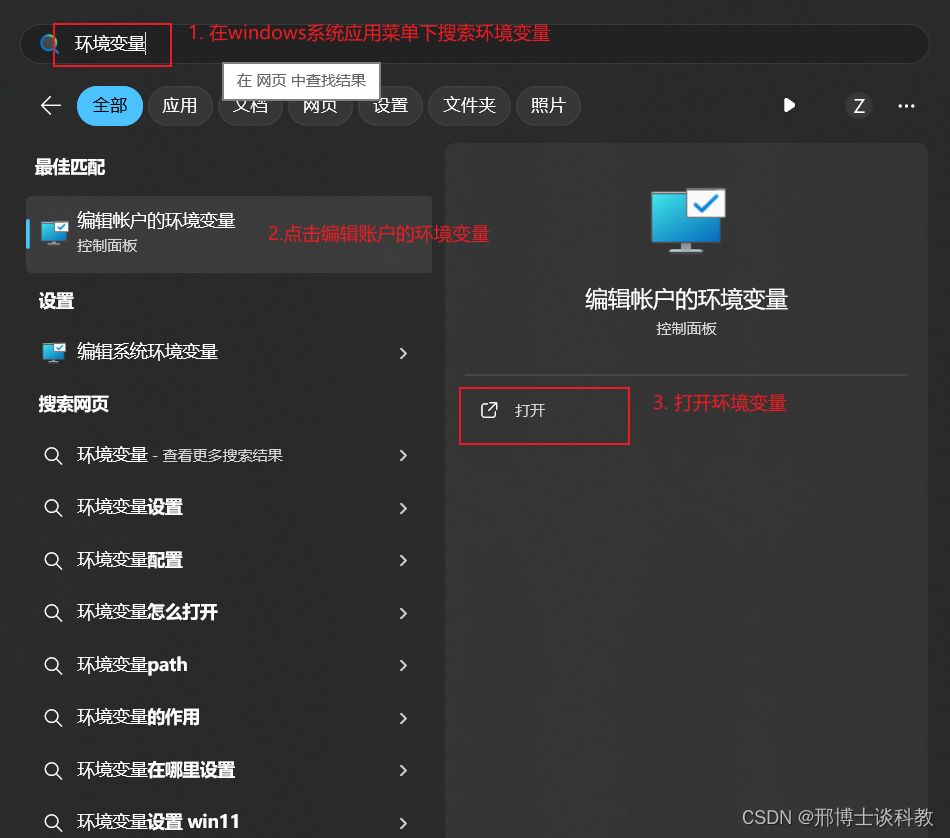
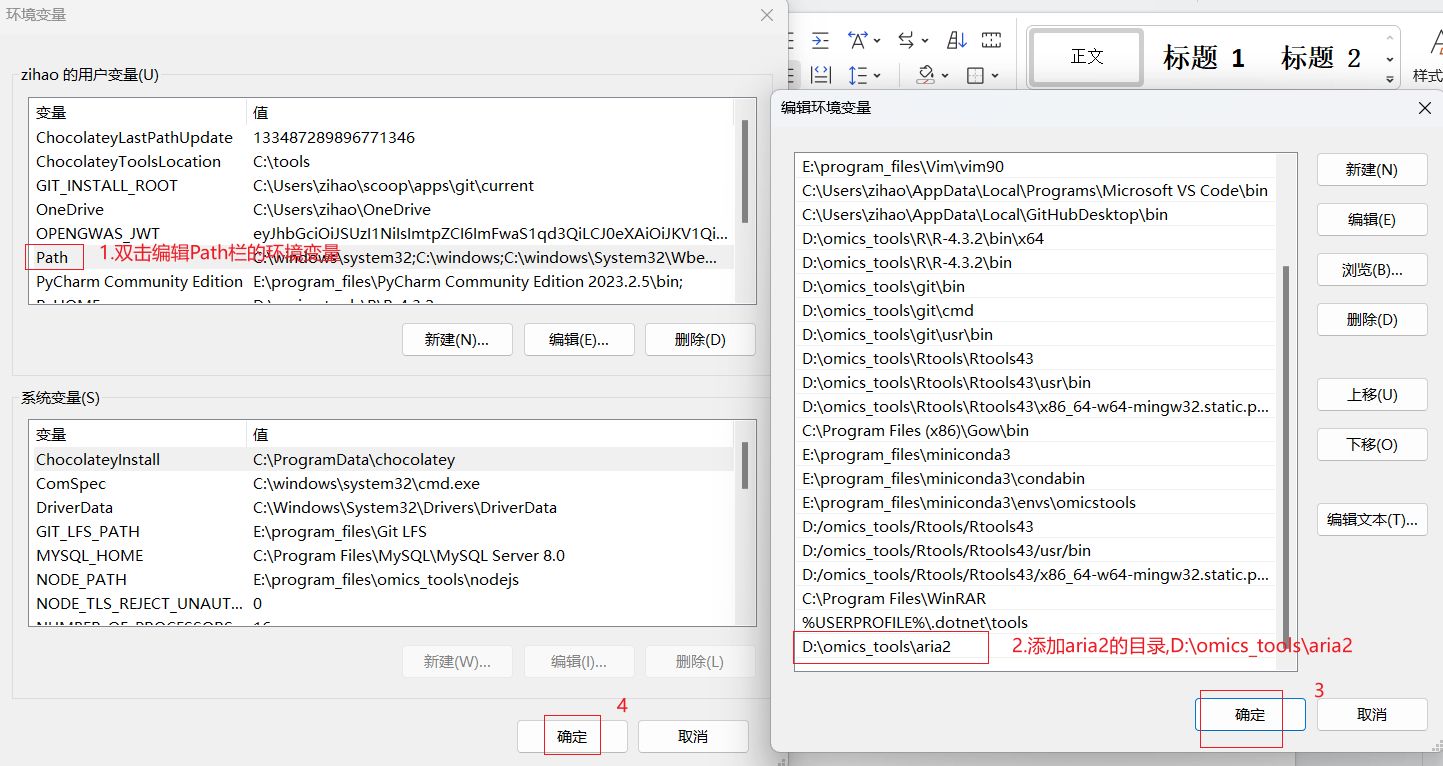
配置aria2c的环境变量
环境变量的配置示意图
检测aria2c的环境变量是否配置成功
新打开一个cmd命令提示符,输入which aria2c, 看看是否会返回aria2c的路径
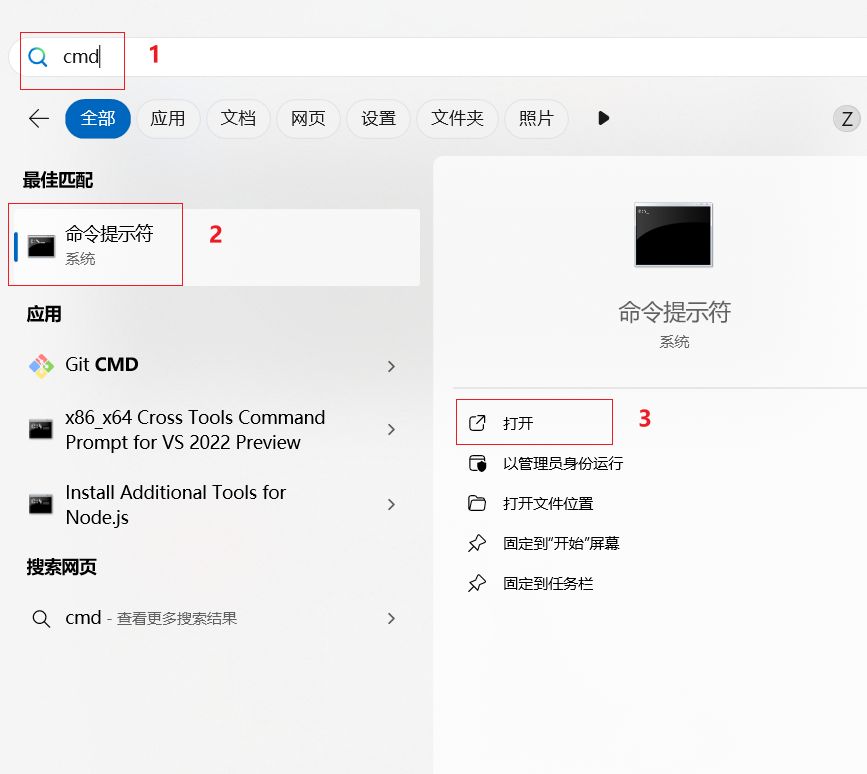
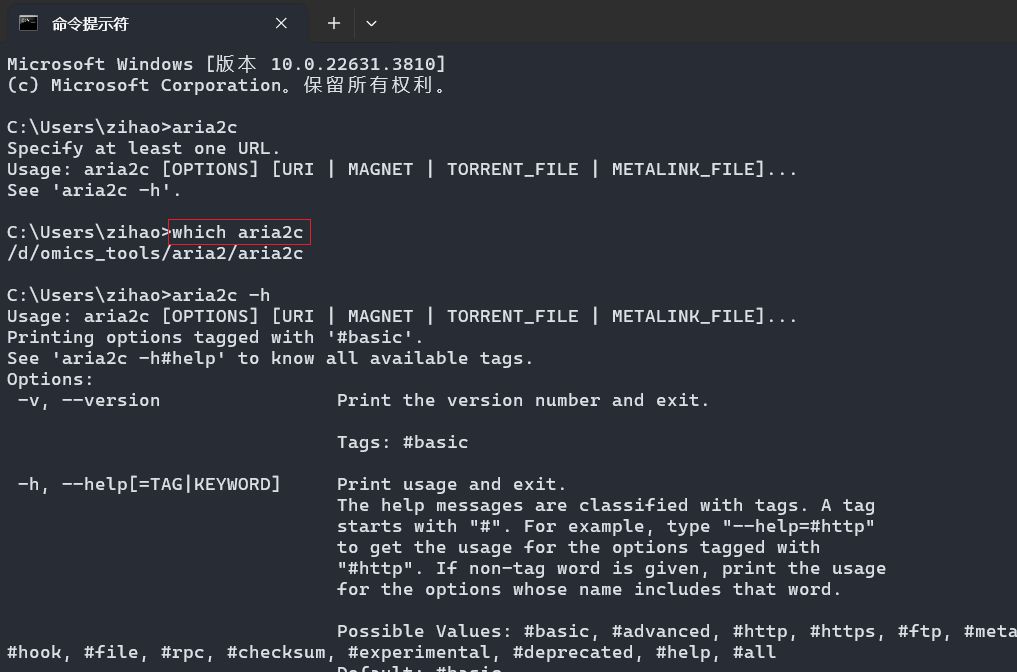
如果成功返回了aria2c的路径,至此,aria2c下载工具就配置完成了。
GEO数据下载教程
1.1根据GEO的GSE数据集编号自动下载和处理GEO数据教程(必须要运行的模块,GEO数据下载要首先运行这个模块)
该模块所在的位置和软件界面
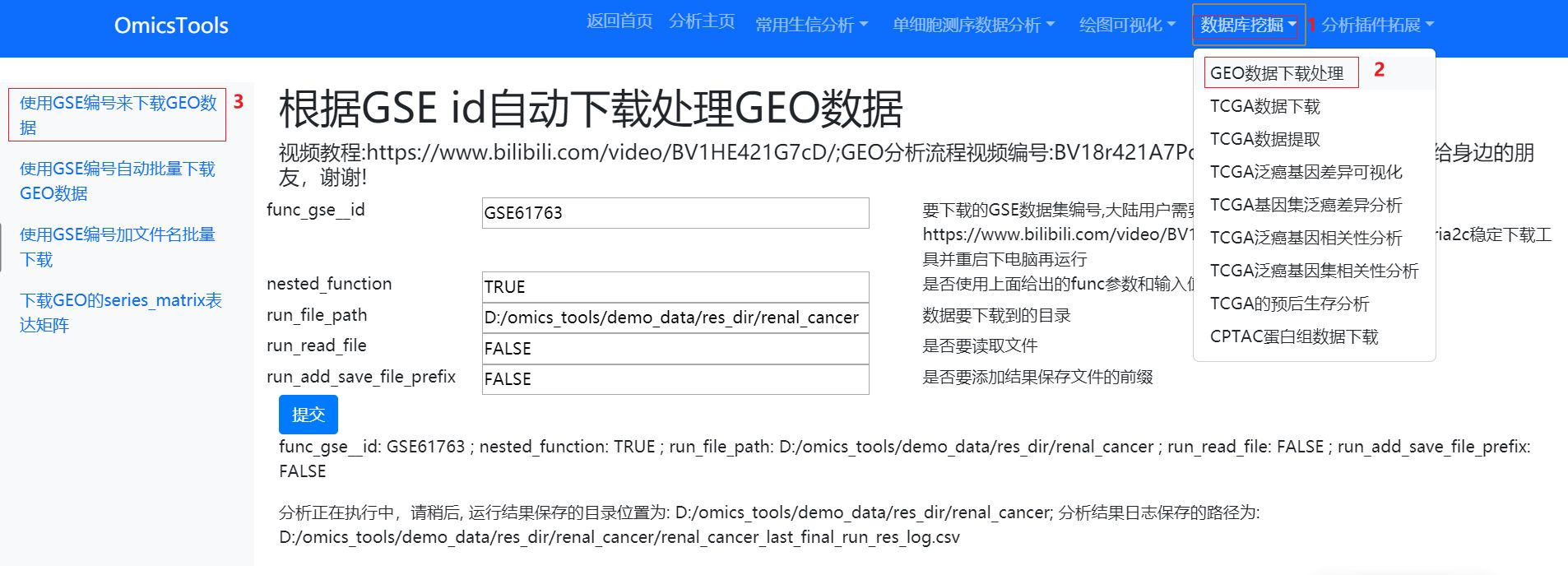
该模块的教学视频链接

运行中的显示信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir\res_dir; 运行结果日志保存的路径为: D:/omics_tools/demo_data/res_dir\res_dir\renal_cancer_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data/res_dir/renal_cancer; 分析结果日志保存的路径为: D:/omics_tools/demo_data/res_dir/renal_cancer\renal_cancer_last_final_run_res_log.csv
下载整理到的结果文件展示
结果文件列表
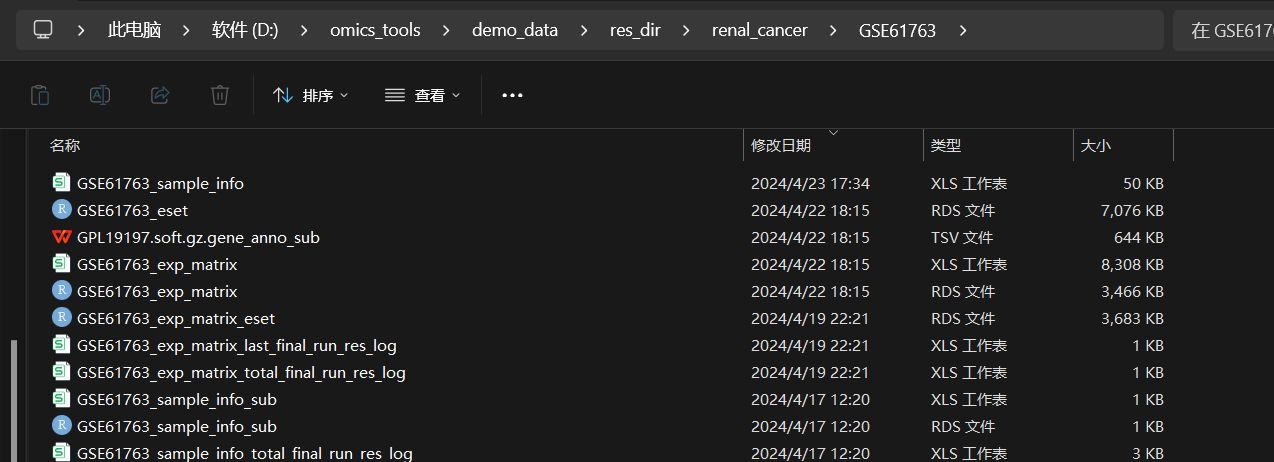
结果文件列表
样本注释文件信息
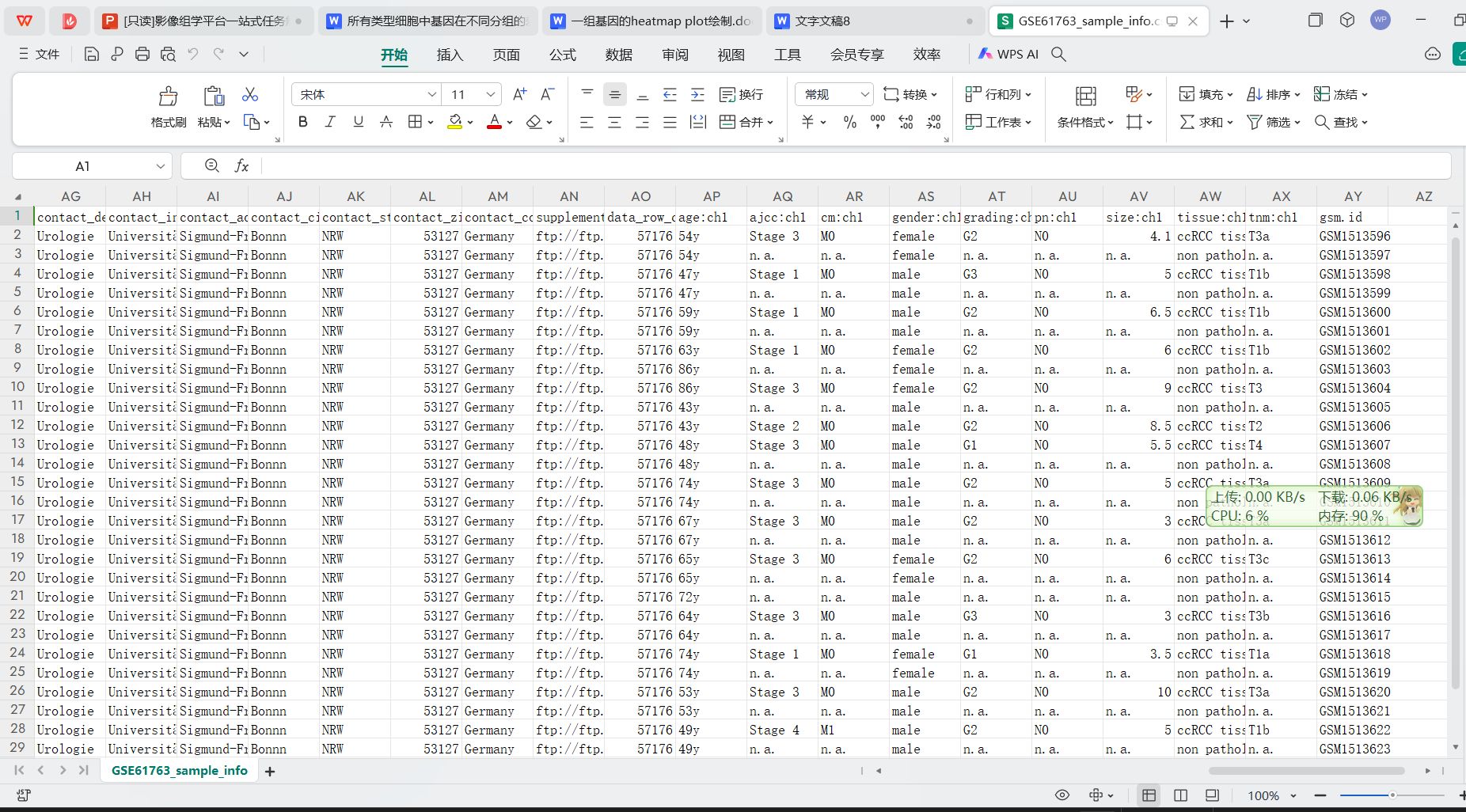
样本的注释信息文件
基因表达矩阵
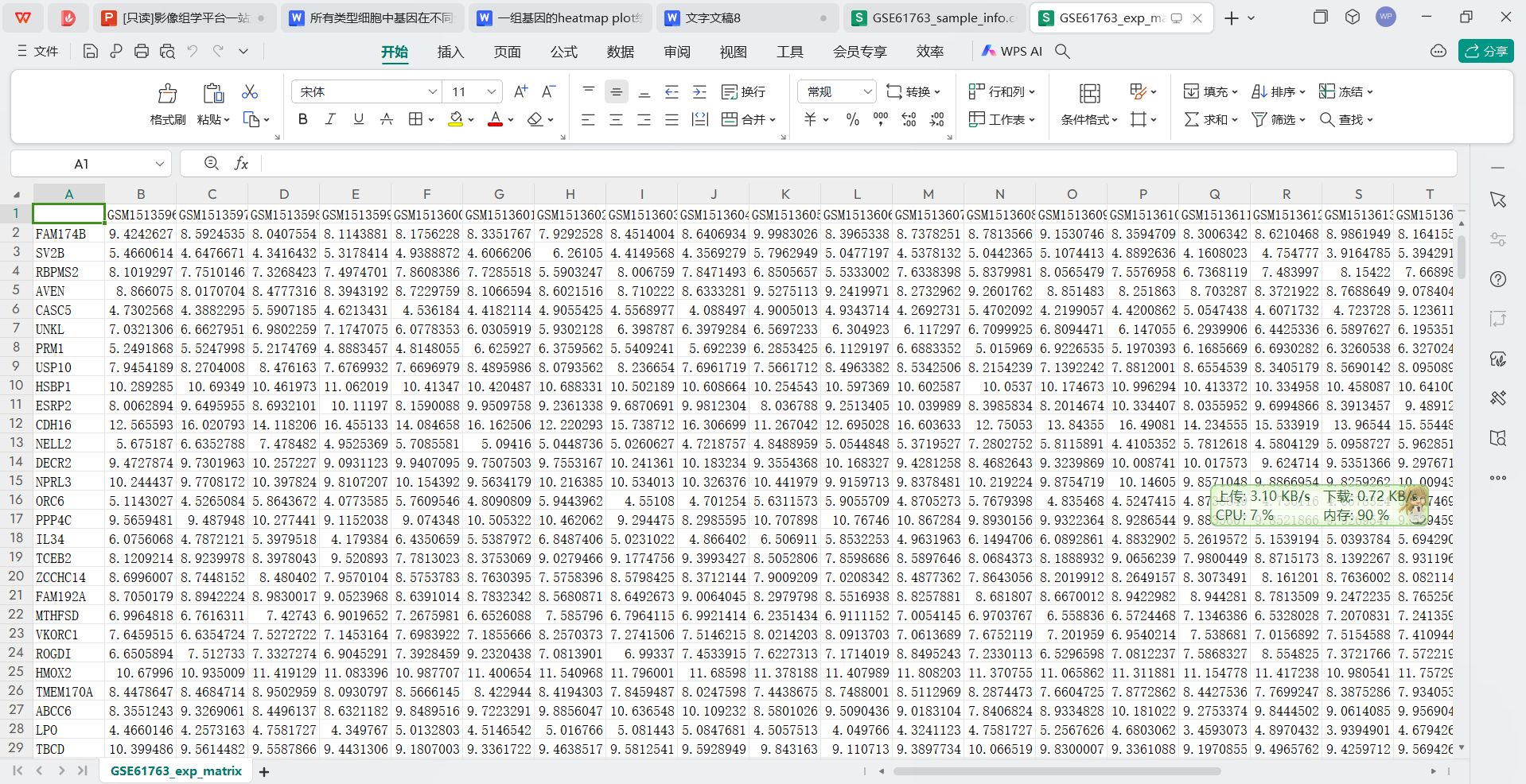
基因表达矩阵文件
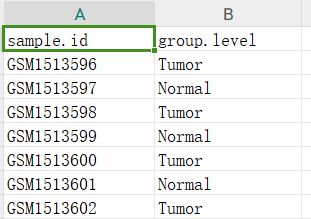
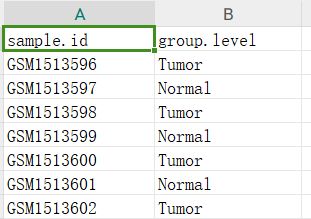
下载GEO数据一定要先运行下这个模块,部分数据集不一定能下载提取出表达矩阵,但是一般能下载到非常完整的样本的注释信息等数据文件,这在对GEO数据集临床信息分析或分组差异分析是非常重要的,当然如果少数GSE数据集遇到没有提取出GSEXXX_sample_info.csv的情况,也可以看看我b站的教学视频,根据GEO网页中的样本编号的分组情况,自建一个这样的分组文件,分组文件一般是长这个样子的,有两列,一列列名叫sample.id,是GSM编号信息,另一列是group.level,存放的是分组信息,当然sample.id这一列的样本名还是要以表达矩阵的样本名为准,这样才能让表达矩阵后面跟样本分组信息按照相同的列名整合在一起:
对于部分没有下载出表达矩阵的数据集,如果该数据集的GEO网页中有GSEXXX_RAW.tar格式的压缩包,可以运行下面的1.2这一步对RAW.tar的压缩包文件进行提取和处理。
该GEO下载模块下载不同GSE数据集得到的不同下载结果详细答疑:
GEO的数据集格式多种多样,使用该模块下载GEO数据集最好的情况下得到的结果是什么样的?
最好的结果就是下图这样,直接通过该模块点击下载后,会得到下面这些下载提取整理好的文件:
- 一次性得到注释好基因名称的表达矩阵文件,如GSE61763_exp_matrix.csv,GSE61763_exp_matrix.rds
- 得到样本的注释信息文件: 如GSE61763_sample_info.csv,
- 得到从GPL soft注释文件中提取到的基因探针跟基因名称的对应关系的两列数据文件,如GPL191197.soft.gz.gene_anno_sub.tsv文件
- 得到了使用表达矩阵,基因注释信息,样本注释信息等数据构建的ExpressionSet对象格式文件,如GSE61763_eset.rds 文件
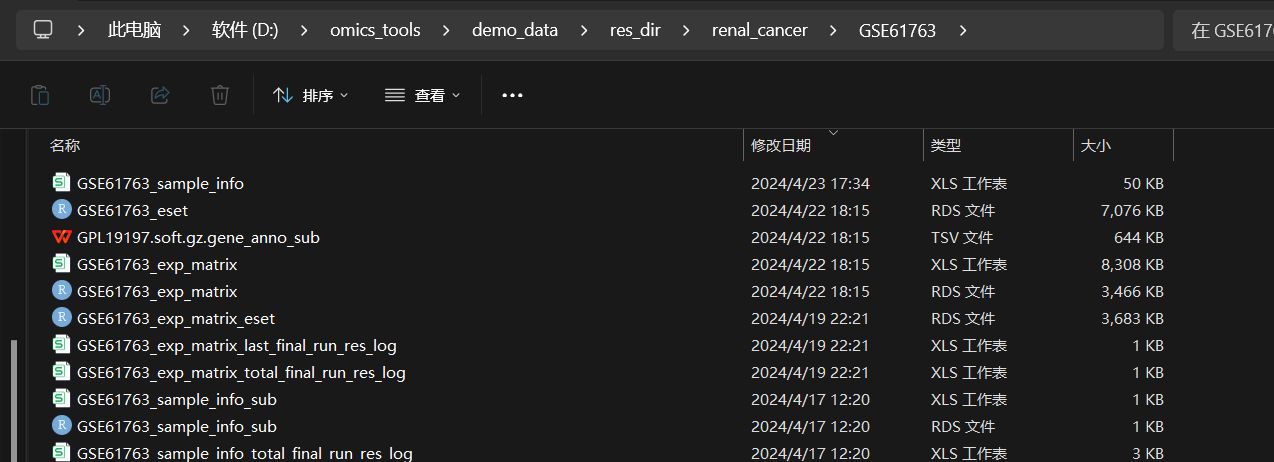
结果文件列表
GEO的数据集格式多种多样,使用该模块下载GEO数据集第二好的情况下得到的结果是什么样的?遇到这种情况该怎么处理?
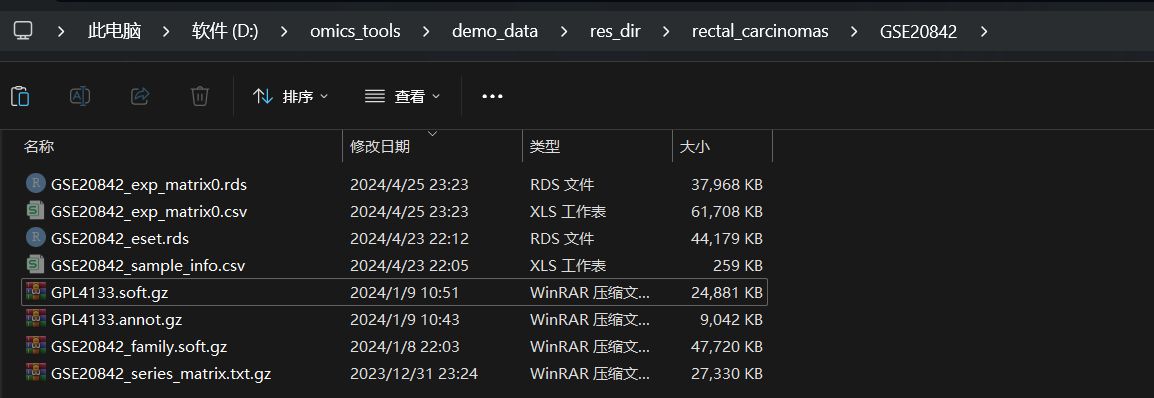
- 提取到了GEO数据集的表达矩阵文件,如GSE20842_exp_matrix0.csv,GSE20842_exp_matrix0.rds
- 得到样本的注释信息文件: 如GSE20842_sample_info.csv,
- 得到了使用表达矩阵,探针信息,样本注释信息等数据构建的ExpressionSet对象格式文件,如GSE20842_eset.rds文件
但是这个表达矩阵有个问题就是,行名不是基因名称,而是探针名称,所以我对这个文件名称做了一些修饰,如果文件名后缀是exp_matrix0.csv,那么这样的表达矩阵里面基本上是没有注释好基因名称,需要在手动下载一下GEO的GPL文件从GPL文件中提取出基因的注释信息再跟表达矩阵合并,这样的处理作为我也有详细的处理教程来教大家怎么做
如果文件后缀就是exp_matrix.csv,那么就是已经成功把基因名称提取出来并整合到表达矩阵中了。对于没有基因名称只有基因探针的表达矩阵,可以用下面这两个模块进行处理:

GEO的数据集格式多种多样,使用该模块下载GEO数据集得到的文件比较差的情况是什么样的?遇到这种情况该怎么处理?
比如像这个例子中,只下载到了GSE158055_sample_info.csv这样的样本分组注释信息文件,这种GEO的GSE数据集一般都是没有提供有效的series_matrix文件,导致没法使用GEO下载工具下载,只能进入该GSE数据集的GEO网页下进行手动下载表达数据文件或一个GSEXXX_RAW.tar的压缩包文件。
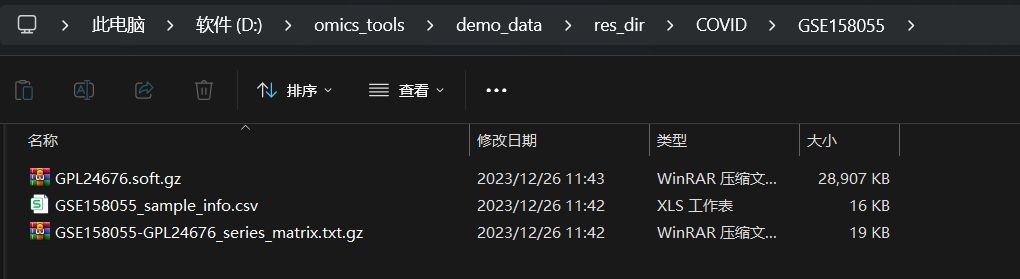
我这里有四个模块可以对转录组或各种平台的基因芯片的RAW.tar压缩包内的多样本表达数据进行整合和处理。
这里的详细教程请查看我这里的文章教程:
转录组和基因芯片GSEXXX_RAW.tar压缩包下载和多样本整合处理教程
GEO的数据集格式多种多样,如果少数GSE数据集遇到没有提取出GSEXXX_sample_info.csv的情况该怎么处理?
如果少数GSE数据集遇到没有提取出GSEXXX_sample_info.csv的情况,也可以看看我b站的教学视频,根据GEO网页中的样本编号的分组情况,自建一个这样的分组文件,分组文件一般是长这个样子的,有两列,一列列名叫sample.id,是GSM编号信息,另一列是group.level,存放的是分组信息,当然sample.id这一列的样本名还是要以表达矩阵的样本名为准,这样才能让表达矩阵后面跟样本分组信息按照相同的列名整合在一起:
关于OmicsTools根据GSE编号自动下载和提取GEO表达数据适用的场景和不适用的场景和原因讲解
series_matrix.txt.gz文件体积太小,用OmicsTools自动下载模块无法提取出表达矩阵
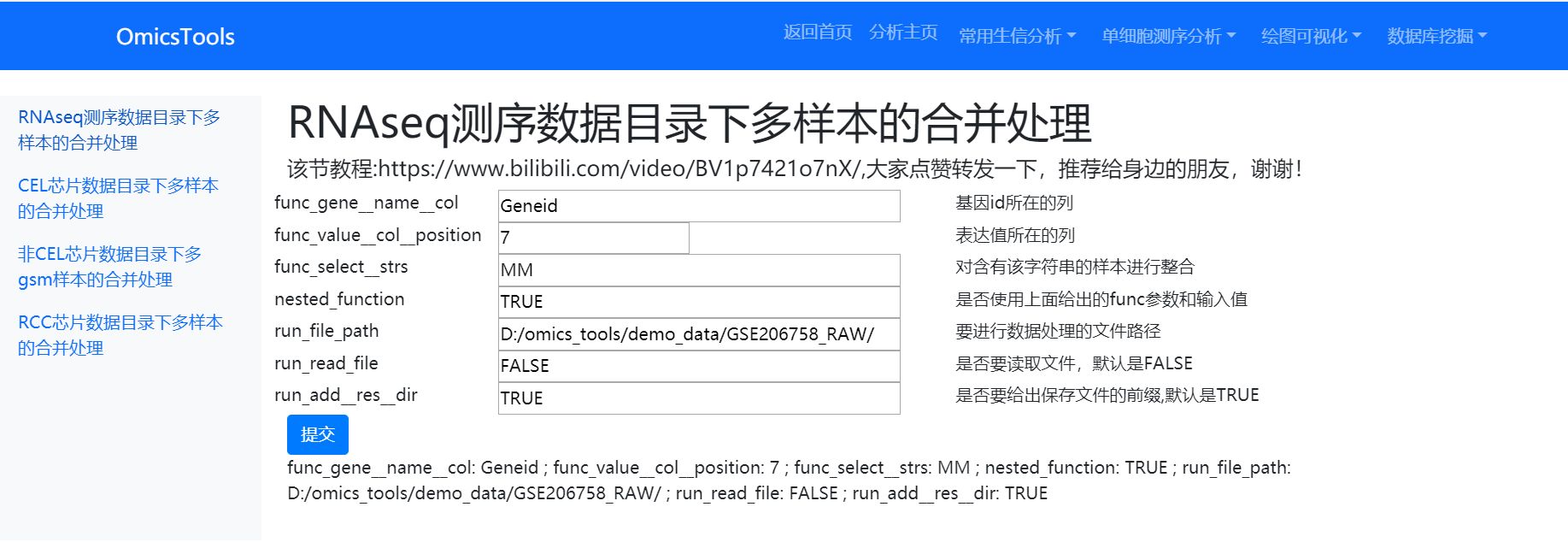
但是对于GEO平台没有给出一个有效的series_matrix表达矩阵,使用OmicsTools的自动下载模块或者GEOquery R包都是没法直接下载提取到数据集的基因表达矩阵的,比如给的该series_matrix的表达矩阵文件大小只有几KB,这种情况下的series_matrix里基本上是没有基因表达矩阵数据的,文件大小太小了,一个有效的series_matrix.txt.gz文件大小应该在1M以上,这样的文件里才可能有有效的基因表达矩阵内容。我们以GSE206758这个数据集来跟大家解释一下,这样的Series_matrix.txt.gz文件,为啥提取不到基因表达矩阵?
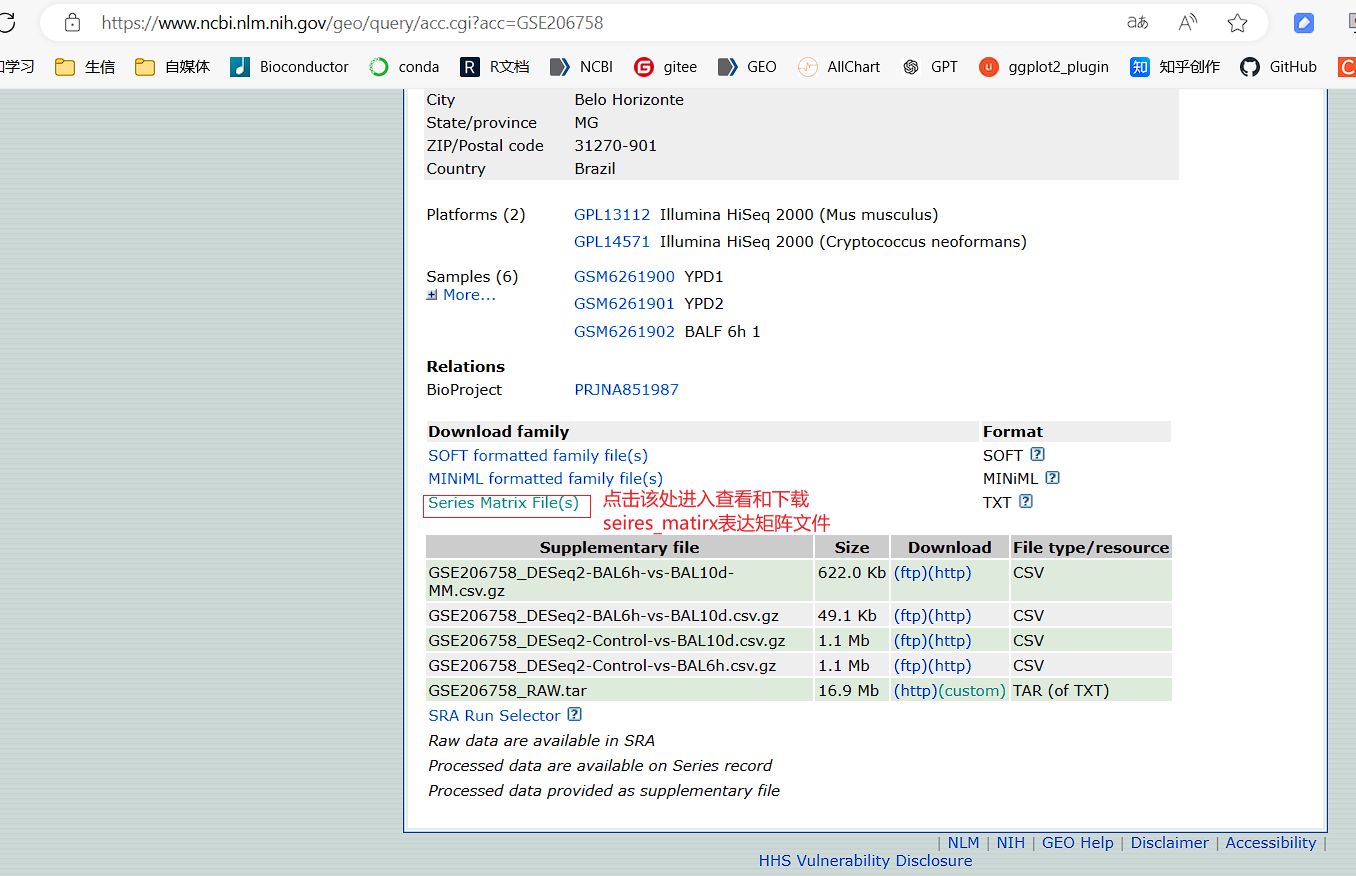
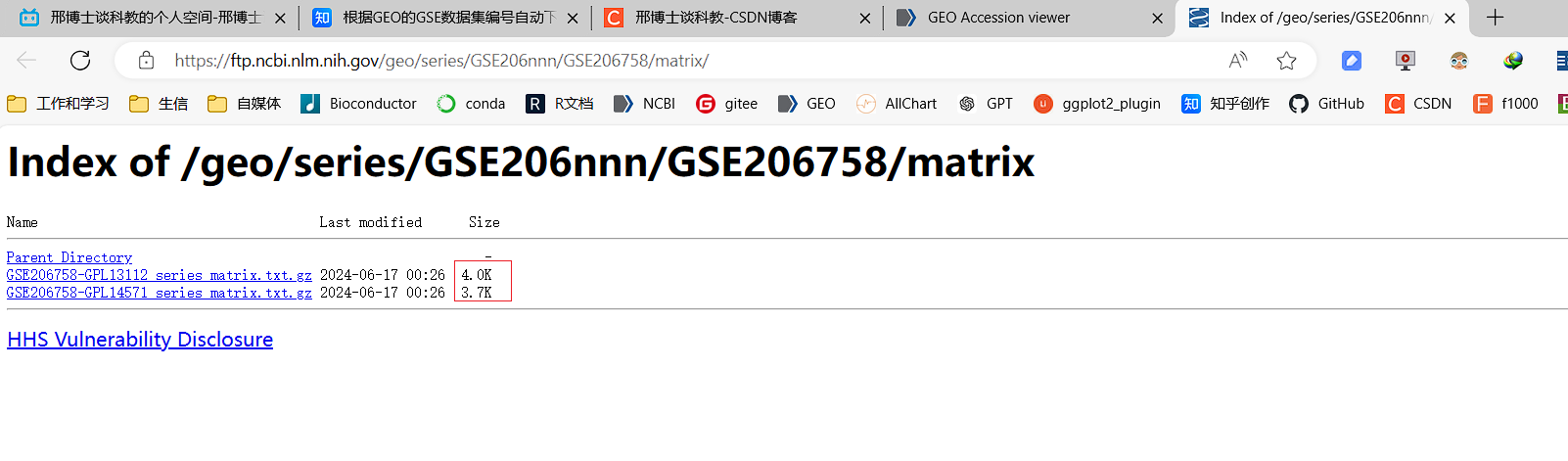
可以看到这里的series_matrix.txt.gz表达矩阵文件只有几KB大小,直接点击上面有蓝色下划线的series_matrix文件名我们就能直接下载到这样的series_matrix矩阵文件,在下载完后,我在解压一下这个gz文件给大家看看里面有没有表达矩阵内容可以被获取。
这里的series_matrix.txt.gz下载解压后的内容如下:

可以看到这里面除了有一些数据集的注释信息外,是基本上没有任何表达矩阵的数据存在的,所以这种情况是无法自动提取出数据集的基因表达矩阵的。
series_matrix.txt.gz文件体积不小,用OmicsTools自动下载模块可以提取出表达矩阵
而对于更大一点的series_matrix.txt.gz的矩阵文件,是可以用我们的OmicsTools生信软件自动下载提取出基因表达矩阵的,以GSE61763数据集为例:
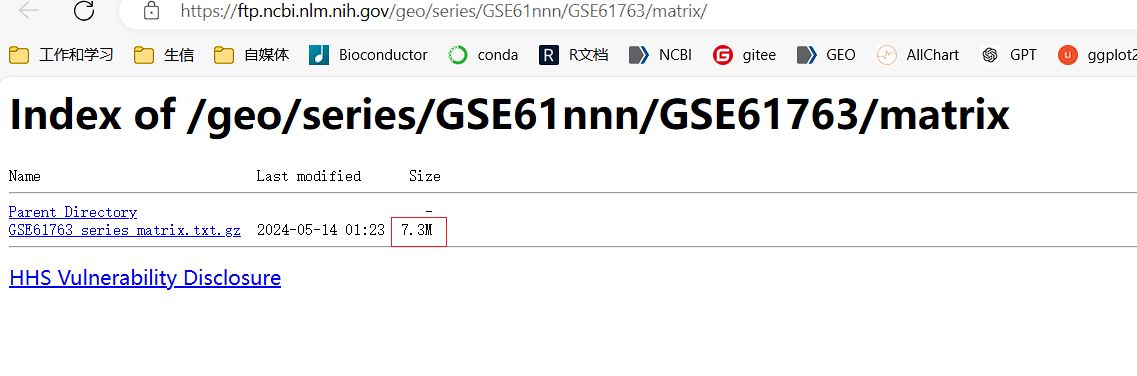
我们看到GSE61763的series_matrix矩阵的文件有7.3M,这个就比前面的那种几KB的文件大的多了,一般这种稍大的series_matrix文件我们都是能直接提取出表达矩阵的。
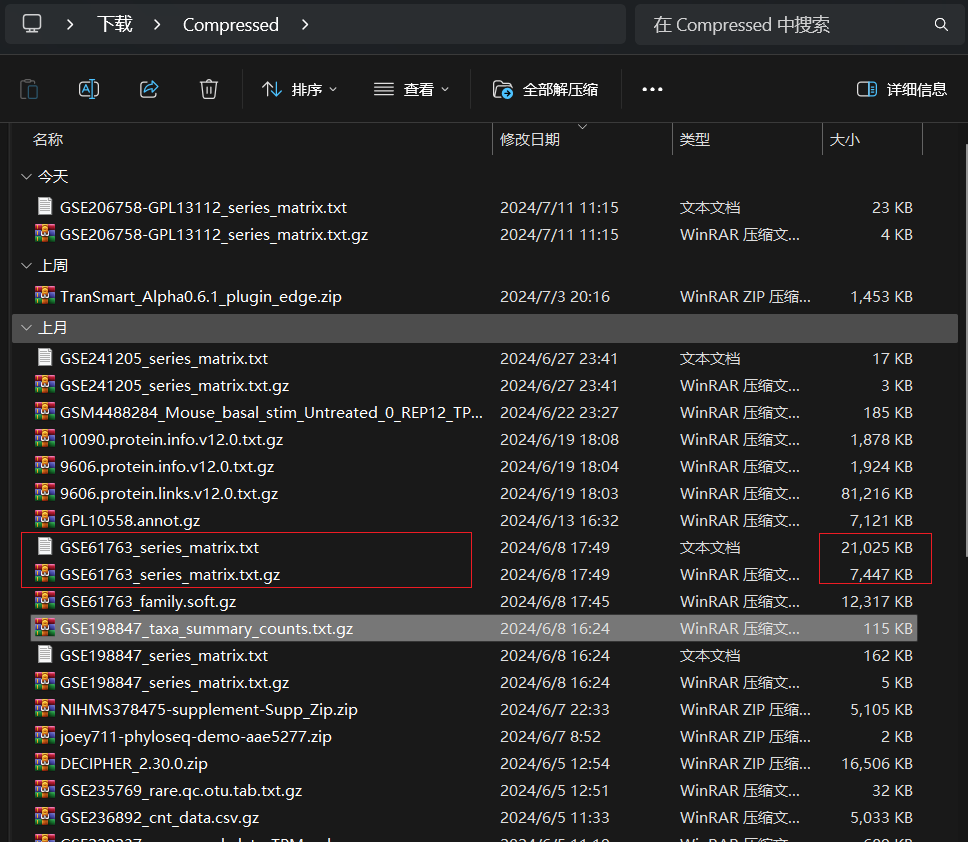
我们看到这个GSE61763数据集的series_matrix的压缩包下载后是7.3M,解压后是21M,也是比较大的文件了。
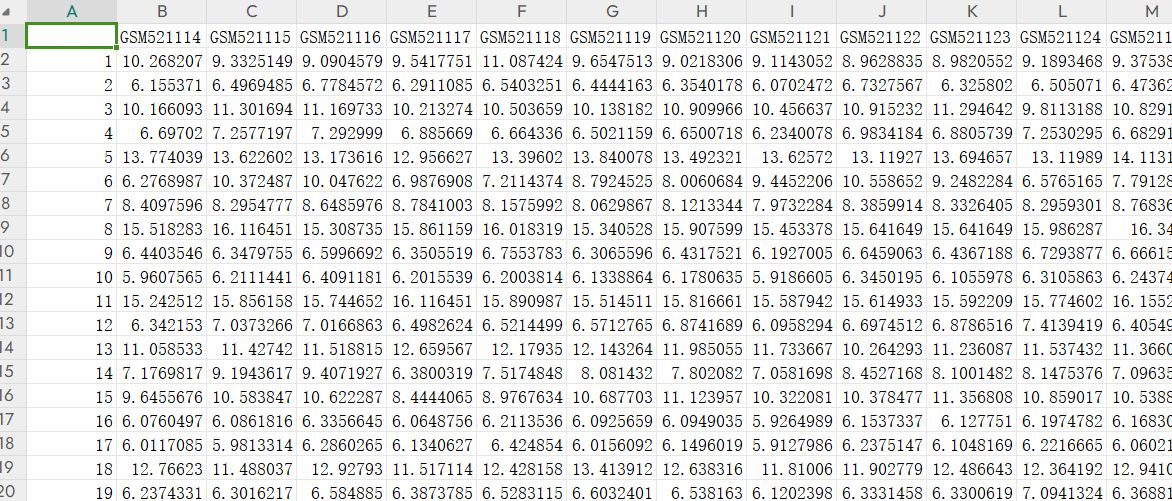
我们解压后用excel表打开并看看里面的内容:
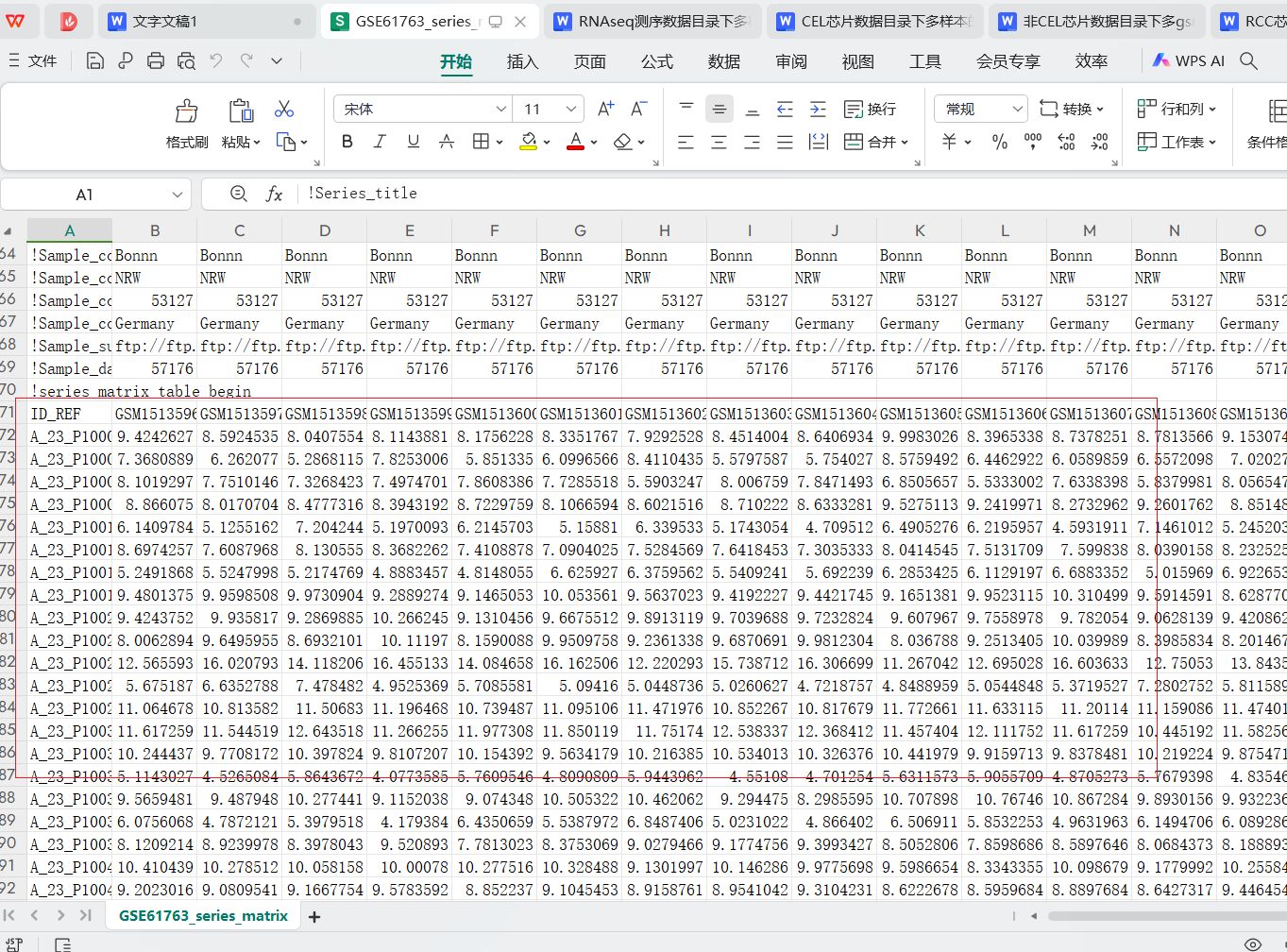
可以看到,像GSE61763这样更大一些的series_matrix表达矩阵,里面除了前面一些行的注释信息外,还包括了每个样本的表达数据信息,这样的series_matrix矩阵就会被我的OmicsTools根据GSE数据集编号自动下载和提取出数据集的基因表达矩阵的。
对于series_matrix.txt.gz文件太小,没有有效的表达数据情况的下载和处理讲解
对于GEO平台上托管的很多GSE数据集而言,一般对于series_matrix.txt.gz文件太小,没有有效的表达数据情况,都会在该GSE数据集的网页下方有表达文件的下载链接,虽然文件名可能千奇百怪,各种各样,一般我们都可以手动下载这些文件后,对这些文件进行进一步的处理,来提取出这些文件的表达矩阵。
而是在GEO网页中给了一个GSEXXX_RAW.tar压缩包的这种或是在GEO中给了一个FPKM表达矩阵文件或counts表达文件的这类,用我的OmicsTools生信电脑软件的自动下载模块是没法直接下载到文件和提取出表达矩阵的,这类文件都是需要手动下载的。
从GEO中手动指定要下载的表达矩阵文件的情况
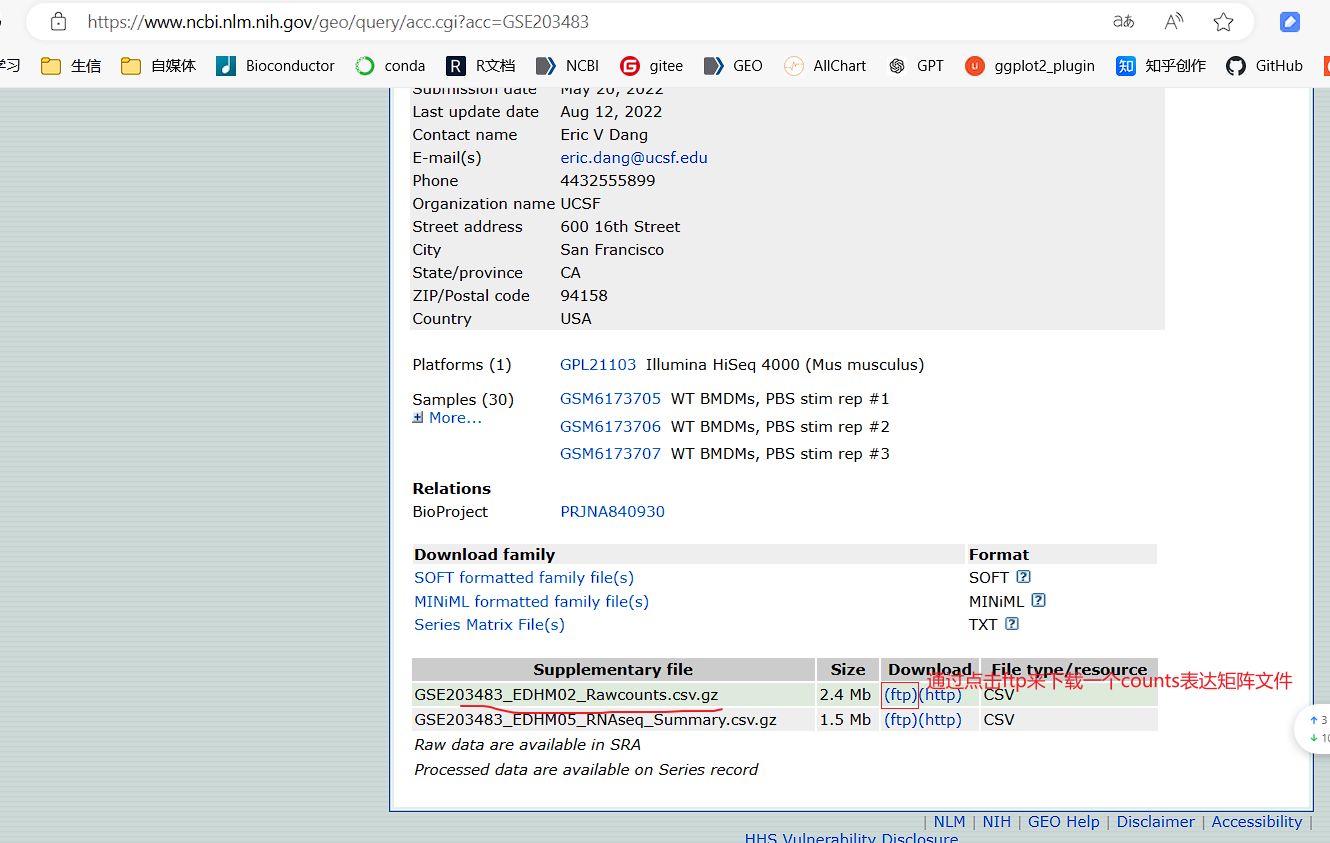 GEO网页中一般这样的情况可以手动直接下载到一个表达矩阵的文件,也可以通过下面这个模块对网页中的表达矩阵文件进行下载
GEO网页中一般这样的情况可以手动直接下载到一个表达矩阵的文件,也可以通过下面这个模块对网页中的表达矩阵文件进行下载

从GEO中手动GSEXXX_RAW.tar压缩包文件的情况
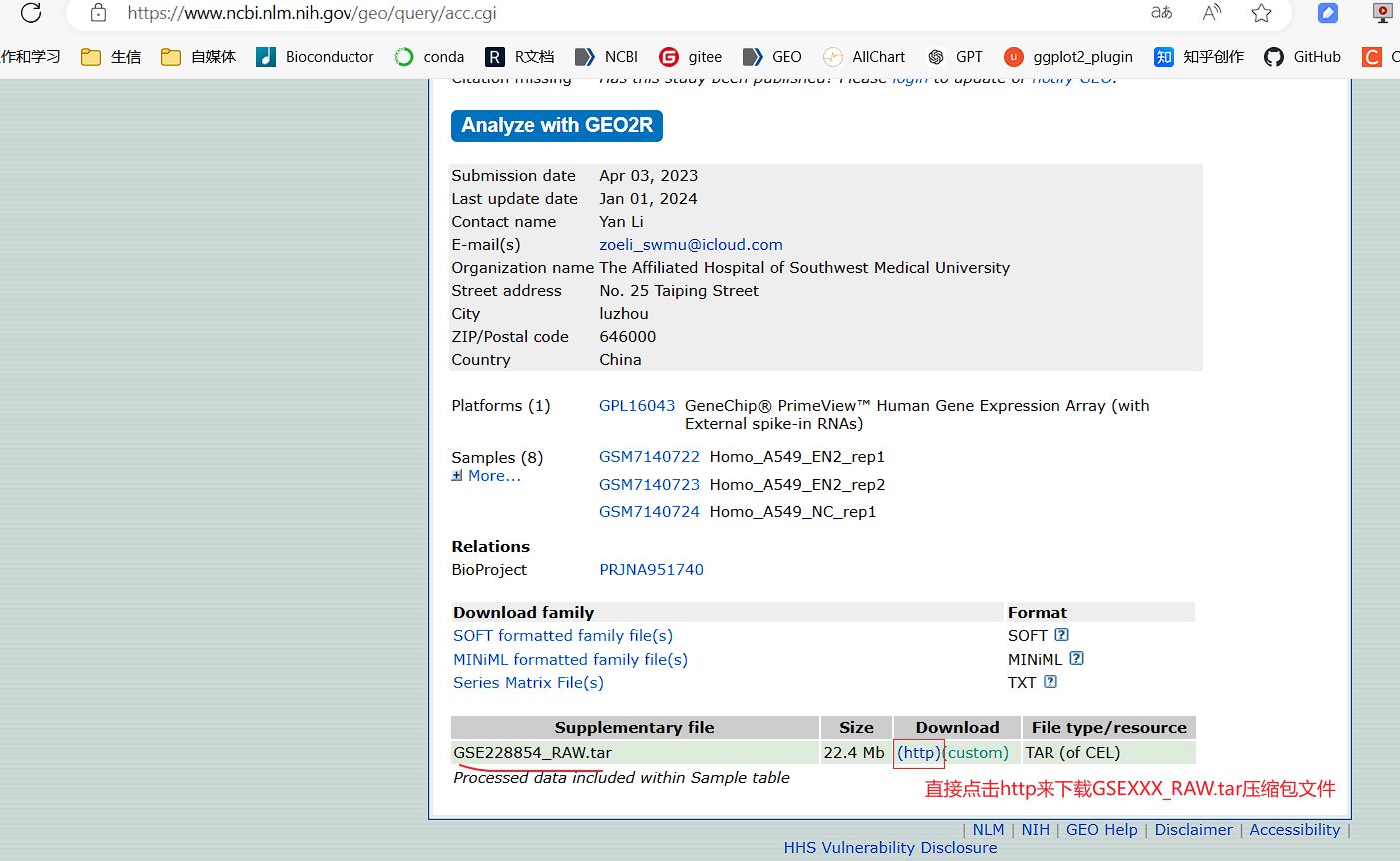
GEO中很多时候需要下载一个GSEXXX_RAW.tar压缩包,并对该压缩包进行解压和合并每个GSM样本的表达数据成一个表达矩阵这种更复杂的情况。
这里的详细教程请查看我这里的文章教程:

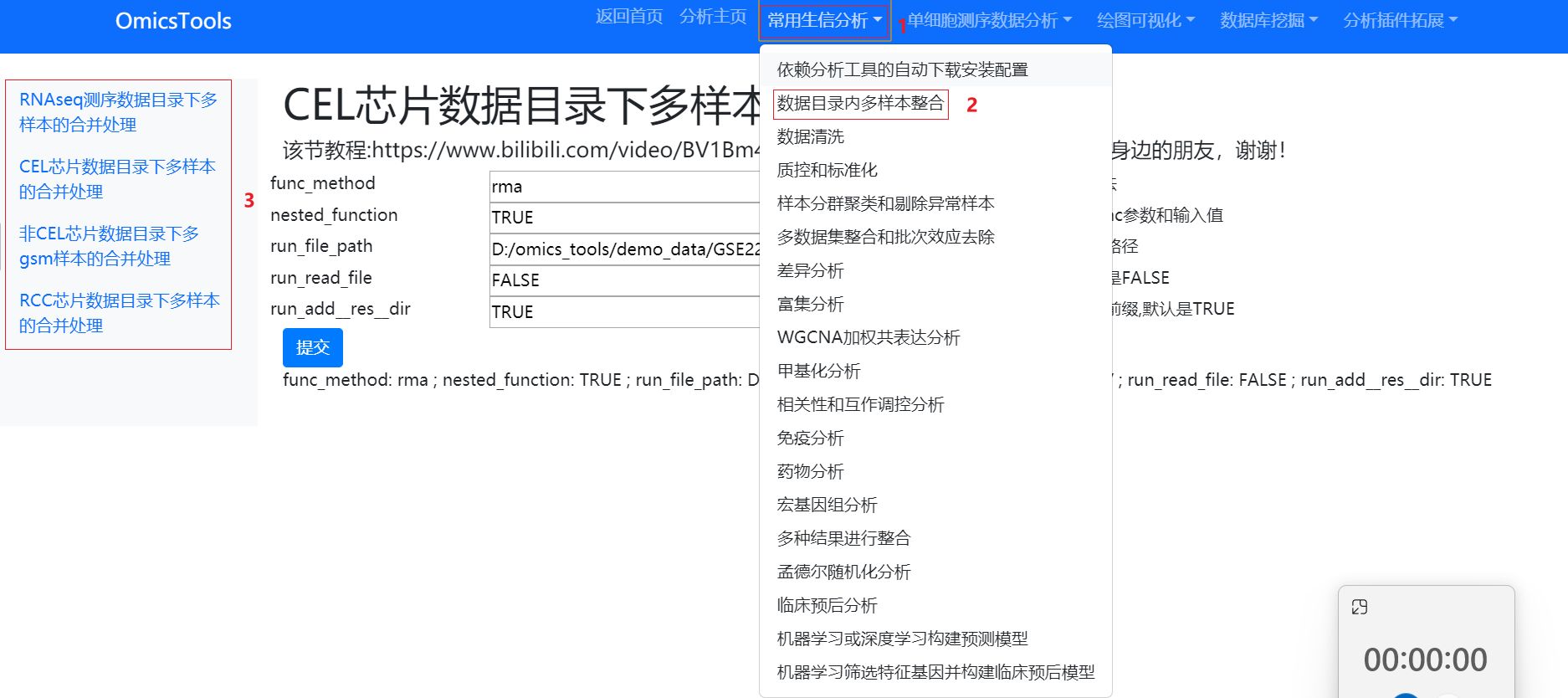
这里有四个模块可以对各种基因芯片GSEXXX_RAW.tar压缩包和普通的bulk RNAseq转录组的测序数据的GSEXXX_RAW.tar压缩包进行读取,合并多样本和处理,在让软件读取和处理GSEXXX_RAW.tar压缩包的时候,需要先对下载的该压缩包解压成GSEXXX_RAW这样的目录。
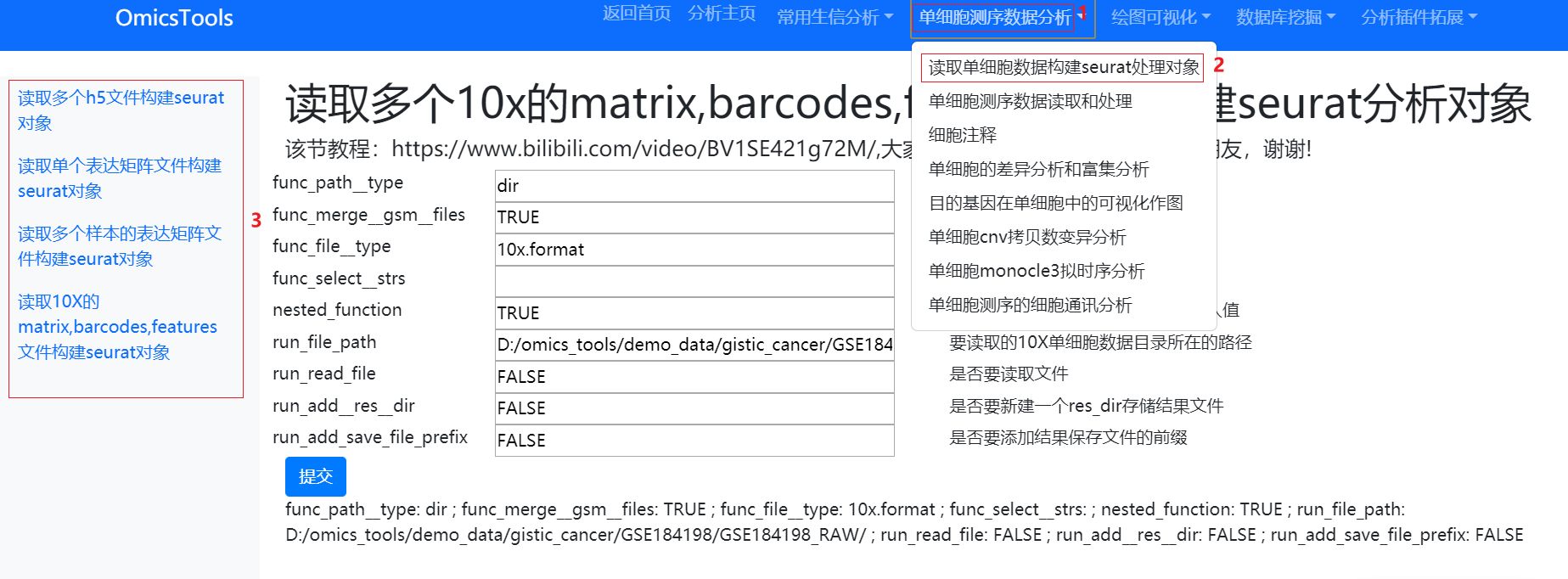
这里也有四个模块可以对各种单细胞的RNAseq测序数据进行读取并构建成seurat分析对象,在让软件读取和处理GSEXXX_RAW.tar压缩包的时候,也是需要先对下载的该压缩包解压成GSEXXX_RAW这样的目录。
1.2 GEO网页中存在可以手动下载的表达矩阵等文件的下载处理教程
GEO网站中的数据集示例
如果在1.1步没有自动下载提取出基因表达矩阵,GEO网页中也没有GSEXXX_RAW.tar压缩包文件,但是存在可以手动下载的一些表达矩阵等数据文件,可以用这一步进行下载和处理。
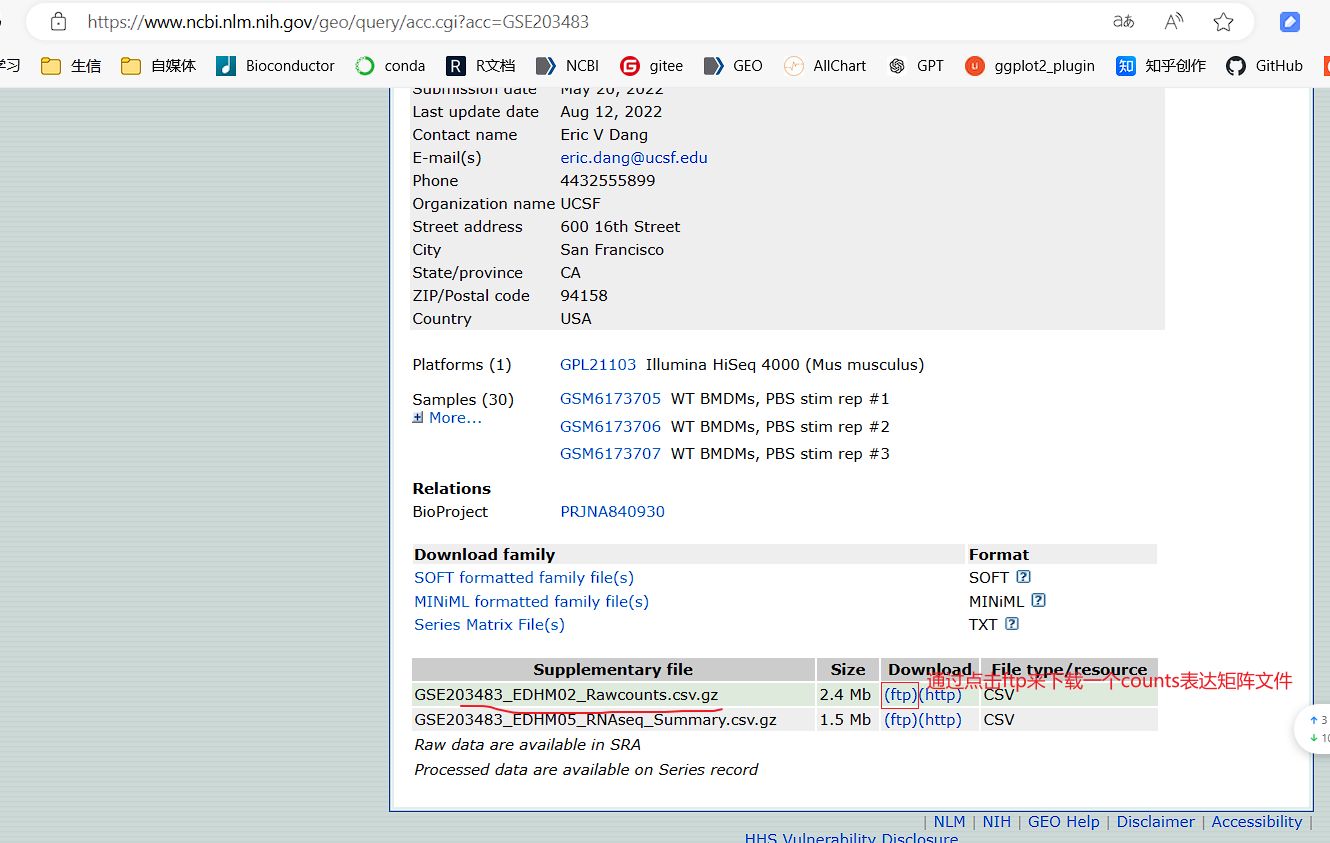
软件位置和窗口截图

输入文件的内容
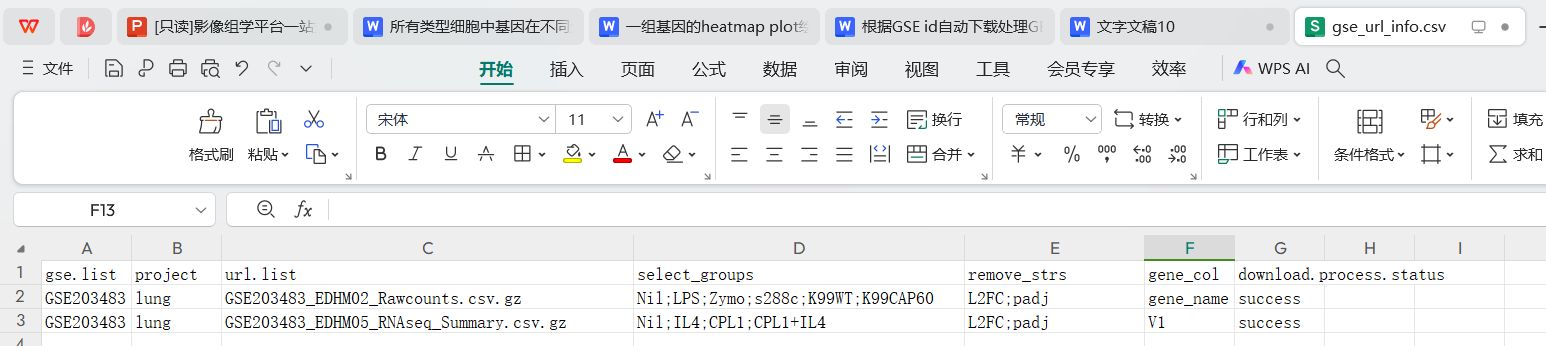
gse.list
project
url.list
select_groups
remove_strs
gene_col
download.process.status
GSE203483
lung
GSE203483_EDHM02_Rawcounts.csv.gz
Nil;LPS;Zymo;s288c;K99WT;K99CAP60
L2FC;padj
gene_name
success
GSE203483
lung
GSE203483_EDHM05_RNAseq_Summary.csv.gz
Nil;IL4;CPL1;CPL1+IL4
L2FC;padj
V1
success
运行中的显示信息
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data; 分析结果日志保存的路径为: D:/omics_tools/demo_data\gse_url_info_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data; 分析结果日志保存的路径为: D:/omics_tools/demo_data\gse_url_info_last_final_run_res_log.csv
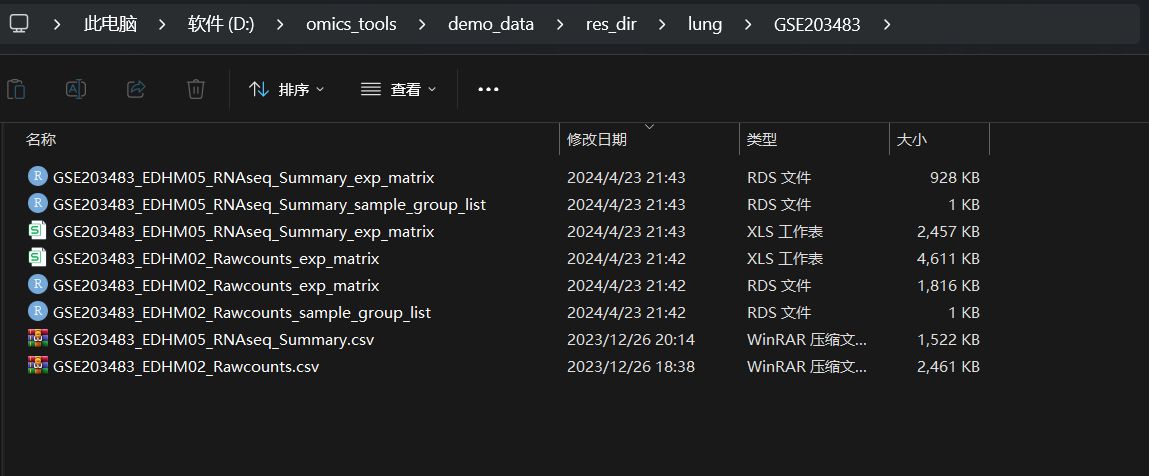
分析结果显示
结果文件列表
1.3 转录组和基因芯片GSE数据集RAW.tar压缩包下载和多样本整合处理教程 (GEO网页中有RAW.tar压缩包可以用这一步下载处理)
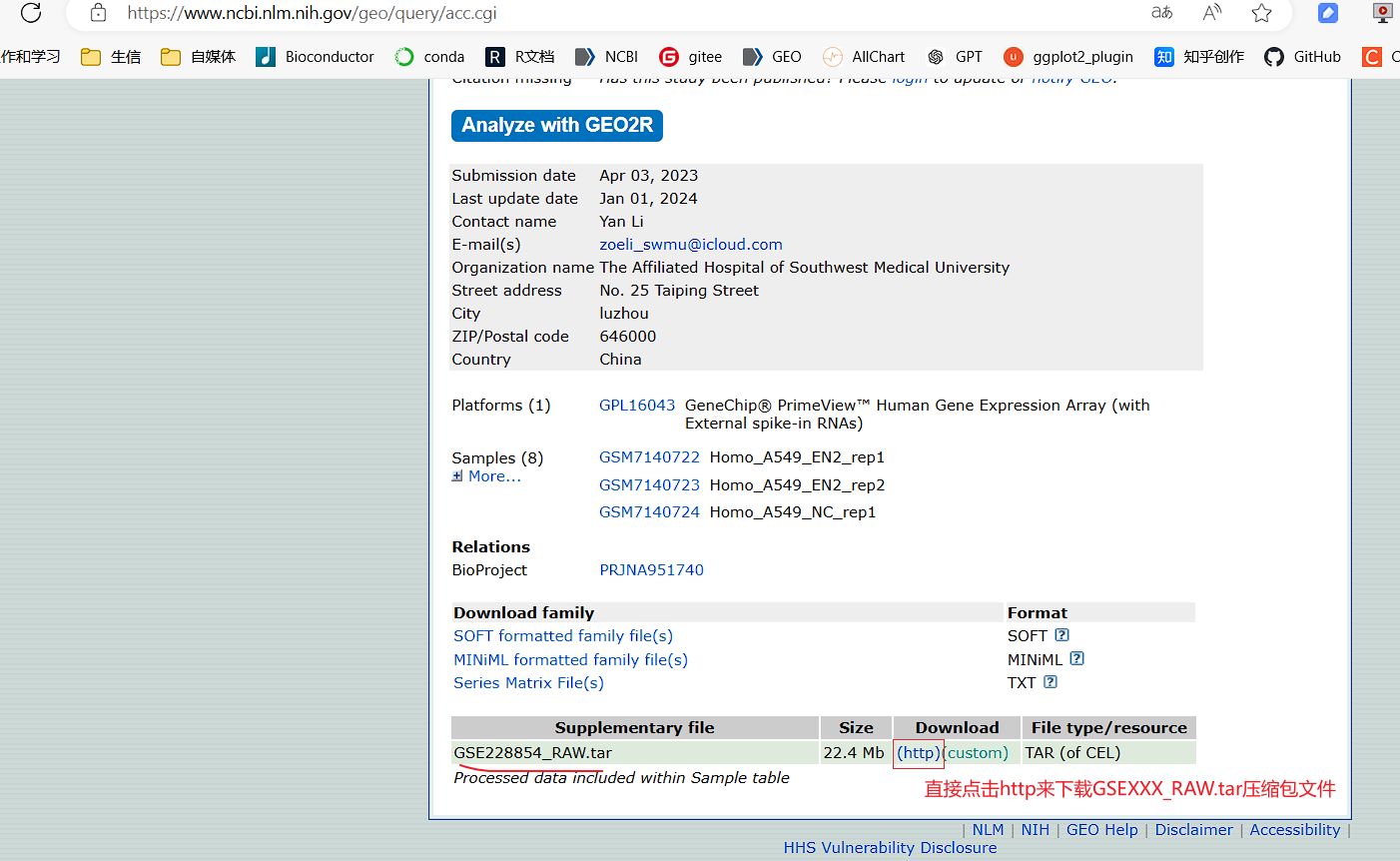
可以在
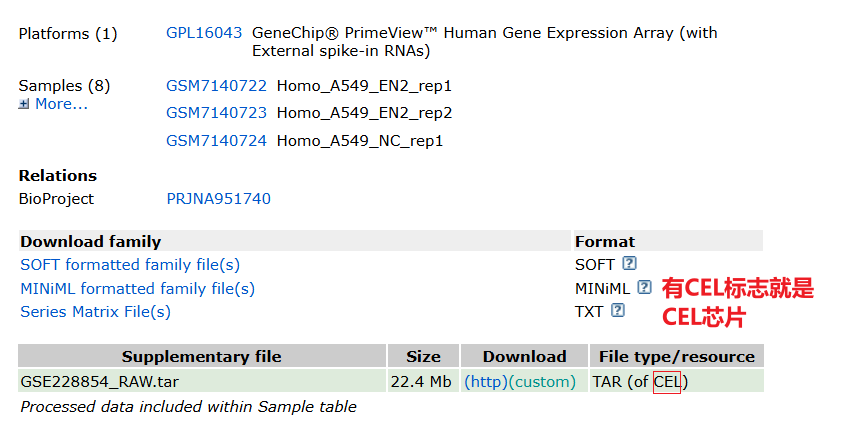
可以在File type/resource里看到数据文件对应的类型,例如有CEL标志就是CEL芯片,有RCC标志的就是RCC芯片。
GSEXXX_RAW.tar压缩包手动下载
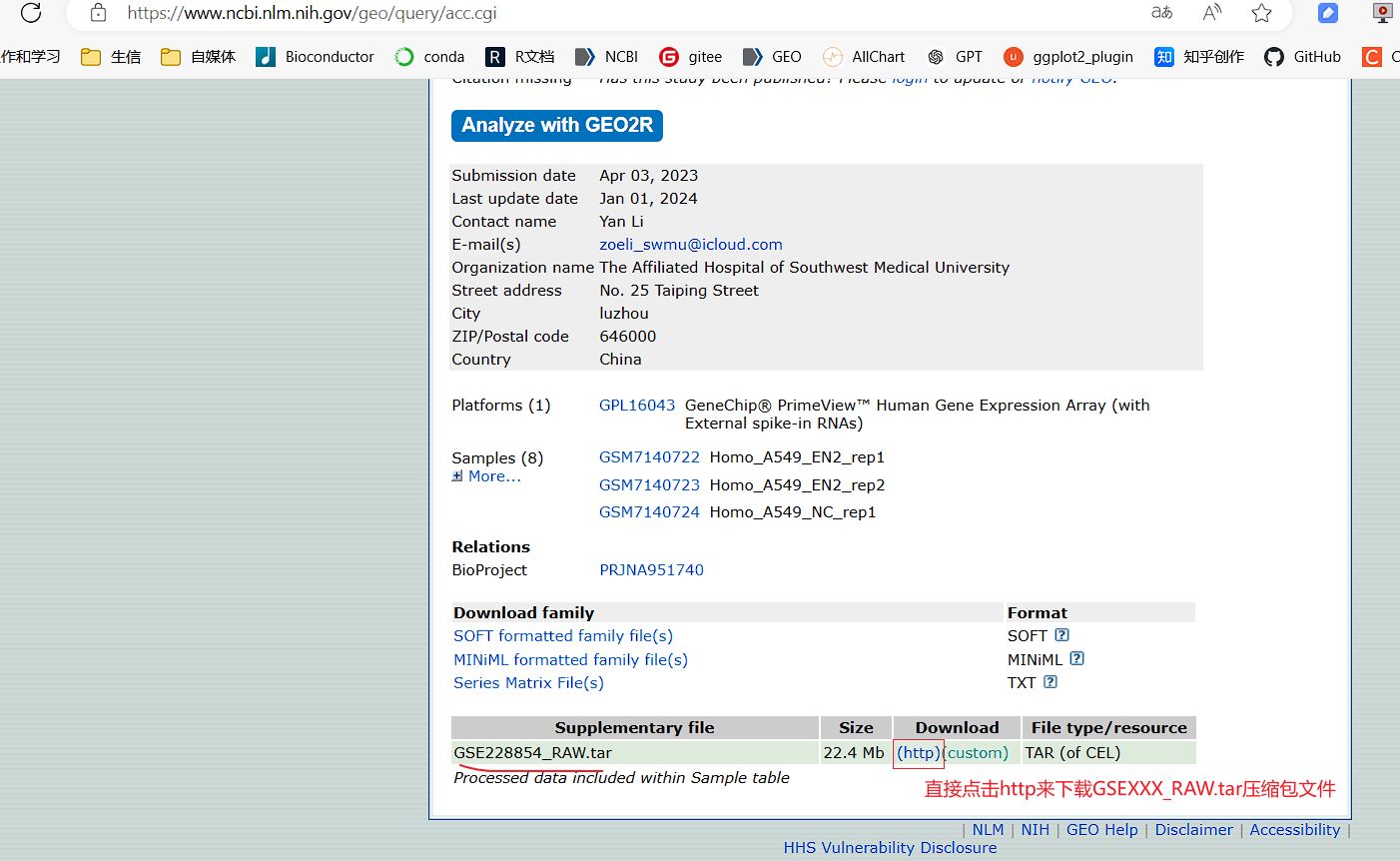
GSEXXX_RAW.tar压缩包的解压
以GSE228854数据集为例,我们下载到了GSE228854_RAW.tar压缩包后,需要把压缩包里面的子文件都解压到一个GSE228854_RAW目录下,目录内是每个GSM样本的表达数据的单个文件,注意,大家一定要这样进行解压操作,就是不要直接把GSE228854_RAW.tar中的文件都解压到当前目录,而是解压到GSE228854_RAW目录下,这样用OmicsTools进行分析的时候会更容易识别和读取和执行。
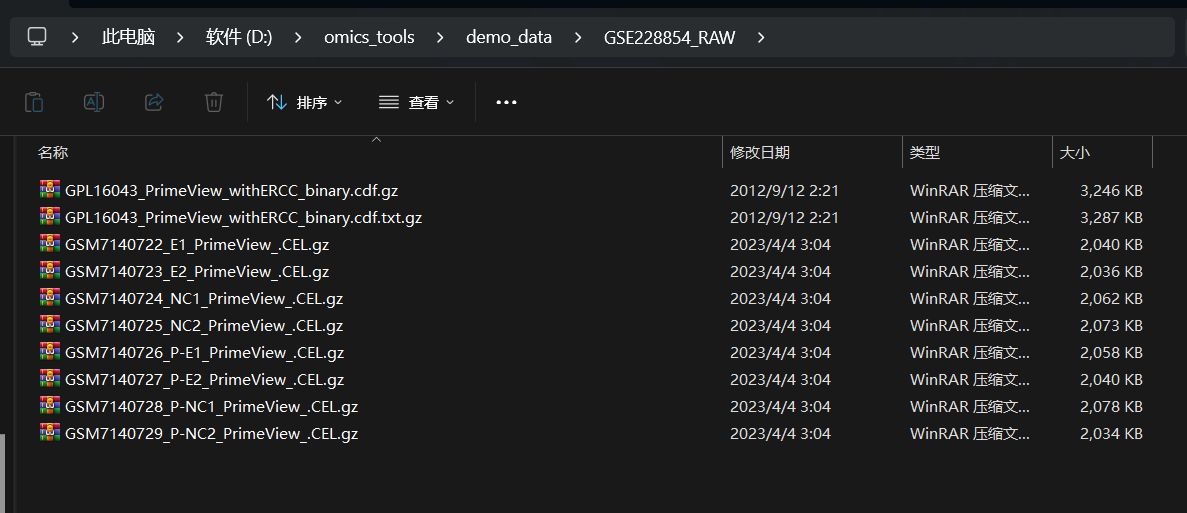
把GSE228854_RAW.tar压缩包正确解压好的文件示意图如下:

普通RNAseq转录组的GSEXXX_RAW.tar压缩包解压后的多样本整合处理
模块所在位置
窗口截图
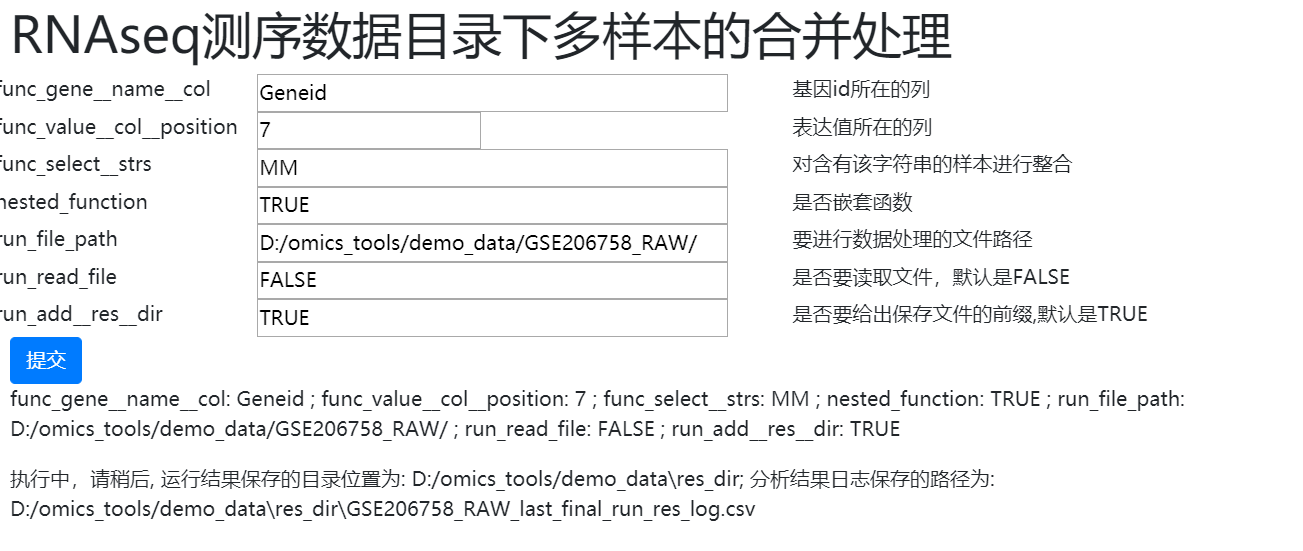
D:/omics_tools/demo_data/GSE206758_RAW/目录下的文件信息

运行状态显示
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE206758_RAW_last_final_run_res_log.csv
运行完成的显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE206758_RAW_last_final_run_res_log.csv
运行完的结果展示
运行完产生的文件

合并的表达矩阵结果
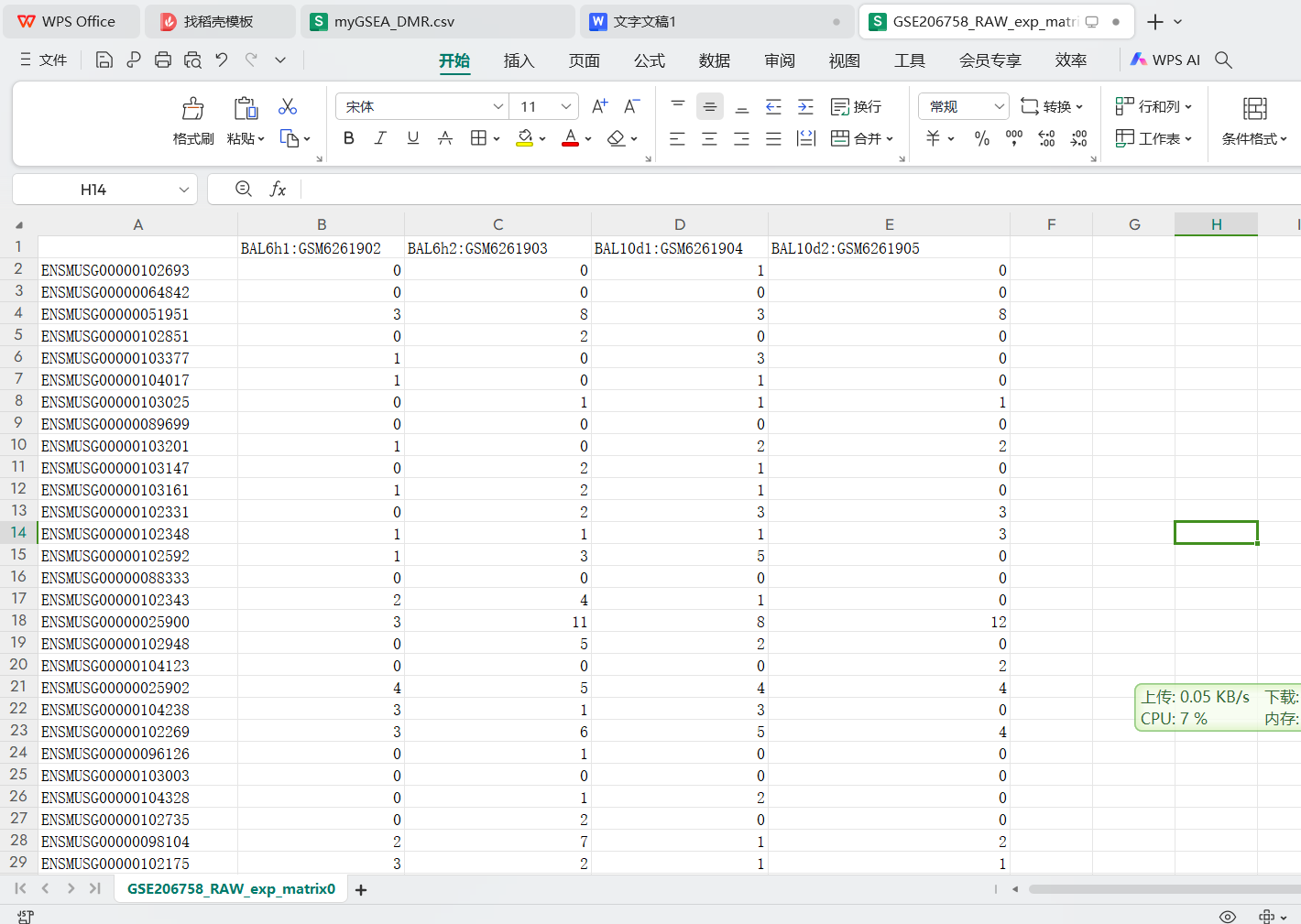
该模块运行后把每个样本的表达数据整合成了一个表达矩阵,对于该数据集里第一列是基因id,不是标准的基因名称,在后面的步骤中我会进一步讲到怎么把基因id转成标准的基因名称,变成有基因名称的表达矩阵。
各种不同格式的基因表达芯片的GSEXXX_RAW.tar压缩包解压后的多样本整合处理
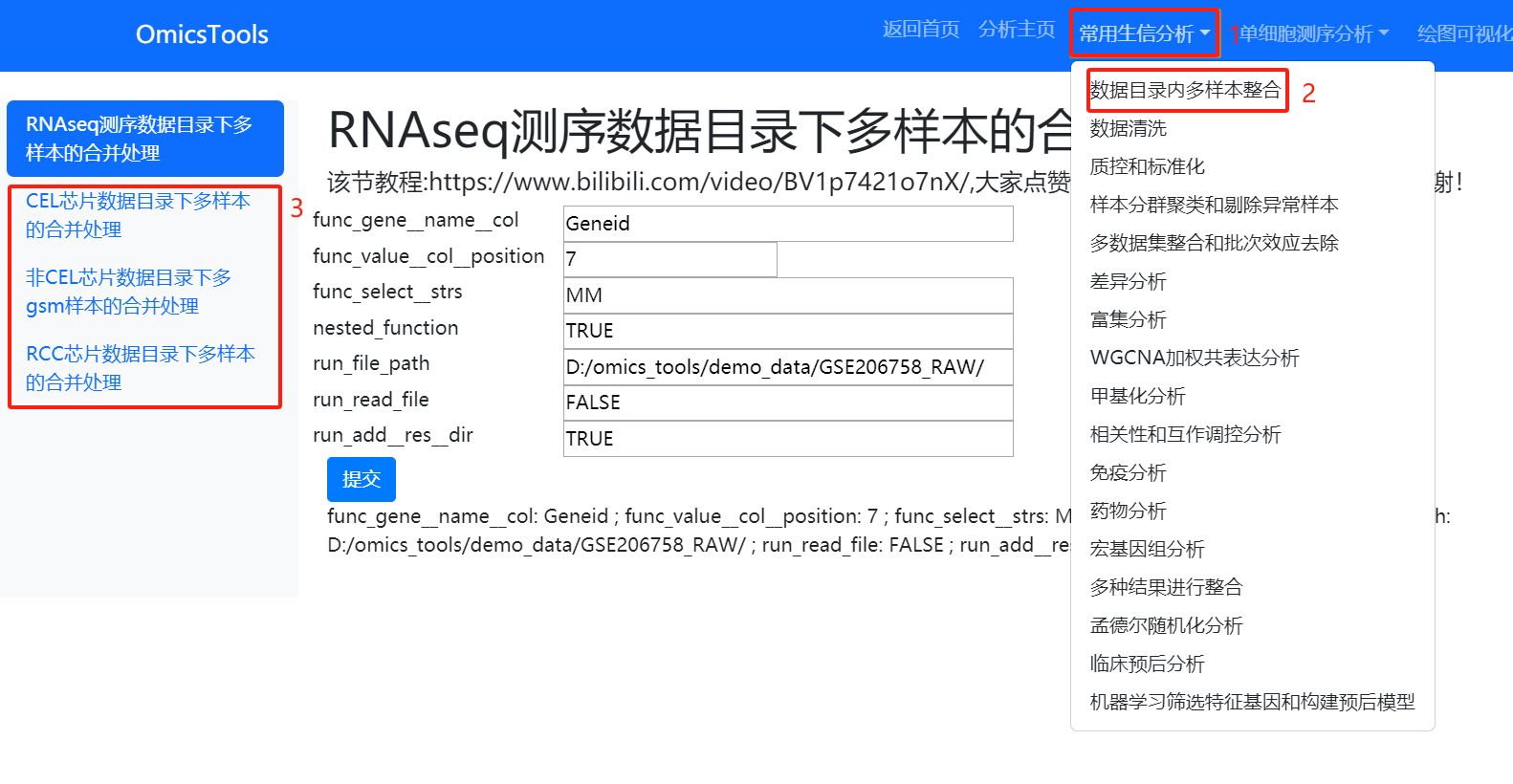
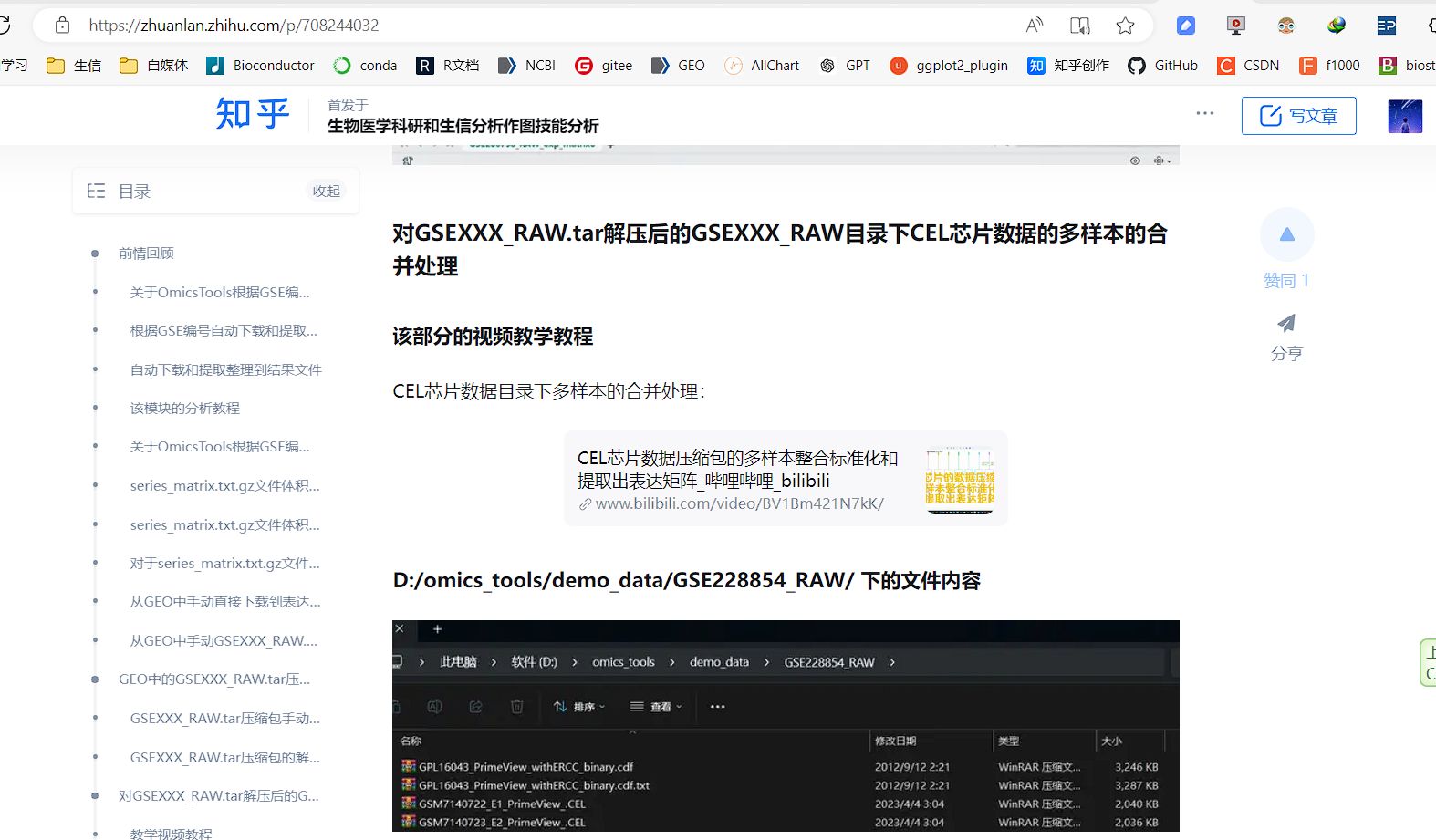
对GSEXXX_RAW.tar解压后的GSEXXX_RAW目录下CEL芯片数据的多样本的合并处理
D:/omics_tools/demo_data/GSE228854_RAW/ 下的文件内容
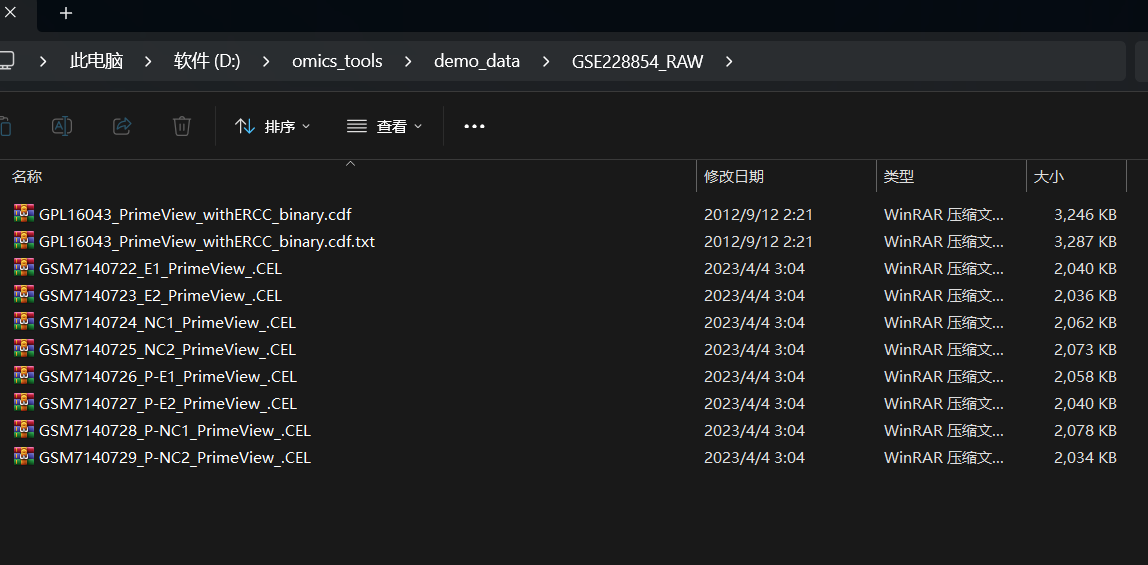
运行窗口展示
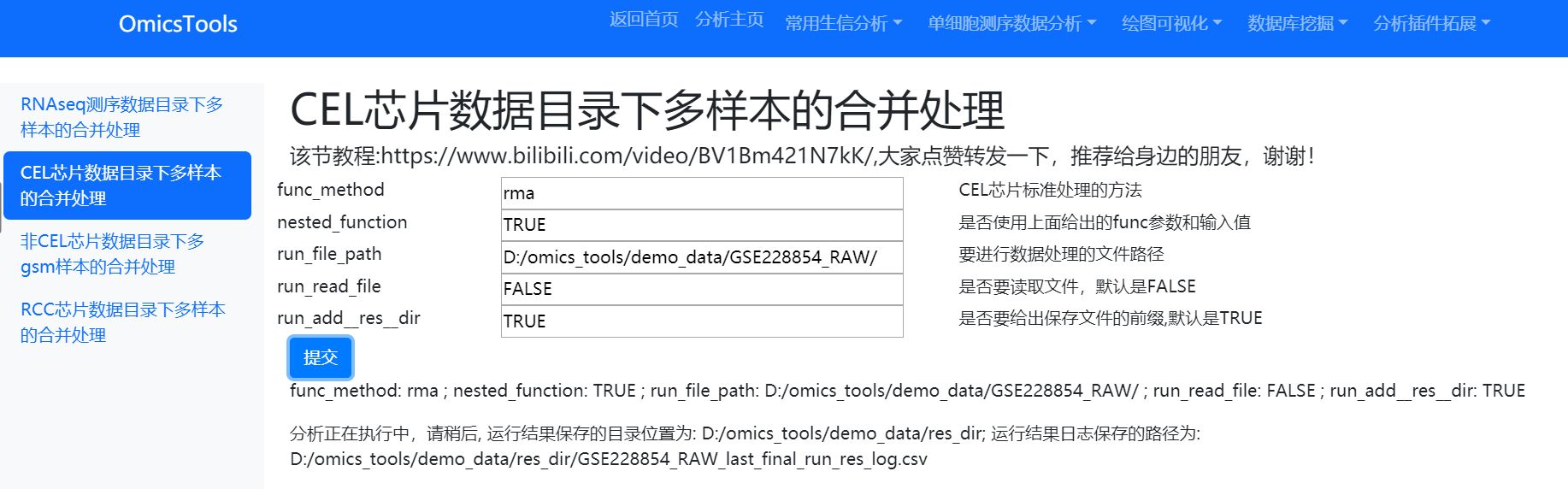
运行状态显示内容
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE228854_RAW_last_final_run_res_log.csv
运行完成状态显示内容:
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE228854_RAW_last_final_run_res_log.csv
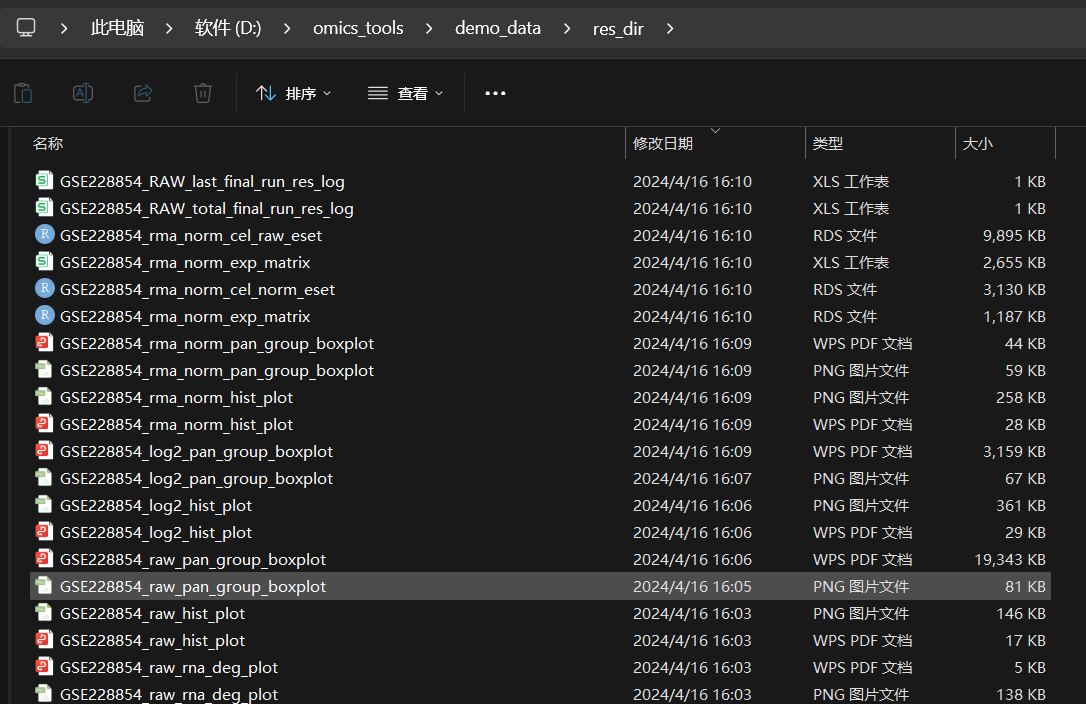
运行完成的结果文件
结果文件列表
查看RNA降解情况
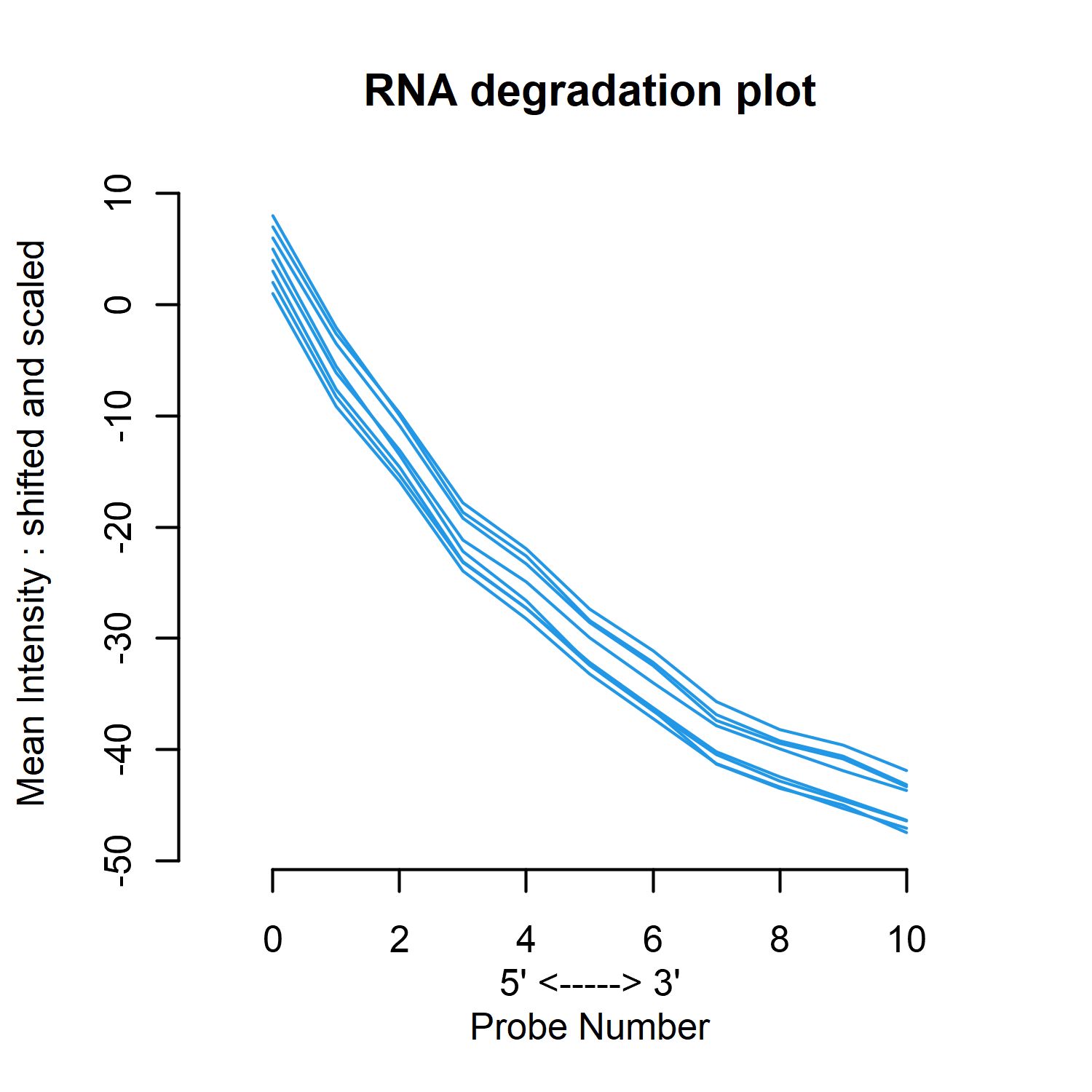
RNA降解曲线主要用于评估样本中的RNA质量,帮助确定其是否适合进一步的实验分析,特别是基因表达分析。你可以通过以下几点来解读该图:
1. 曲线趋势:该图展示了每个位点探针从5'端到3'端的平均信号强度变化。理想的RNA样本在降解曲线上通常会呈现一个逐渐下降的趋势。图中的曲线确实显示出一个平滑的下降趋势,这意味着RNA从5'端到3'端的降解是相对一致的,没有明显的异常波动。
2. 曲线斜率:如果曲线从5'端到3'端的降解非常陡峭,可能表明RNA降解较为严重。图中曲线的斜率不是特别陡峭,这可能表示RNA降解在一个可接受的范围内。
3. 曲线的一致性:多条探针曲线之间的距离也可以反映样本间的一致性。在这个图中,各条曲线彼此接近,说明样本之间的降解模式相对一致,RNA质量在各个样本间的变化不大。
4. RNA质量判断:一般来说,如果降解曲线比较平滑,没有剧烈波动,说明RNA质量较好,适合后续的实验。如果曲线在中途出现波动或不规则变化,可能需要考虑RNA样本是否有降解问题。
这个RNA降解曲线图中显示了较平滑且一致的下降趋势,说明RNA质量较为稳定,适合后续的基因表达实验。这表明RNA的降解较为一致,未出现严重的降解问题。
未标准化前原始数据的数据质量展示
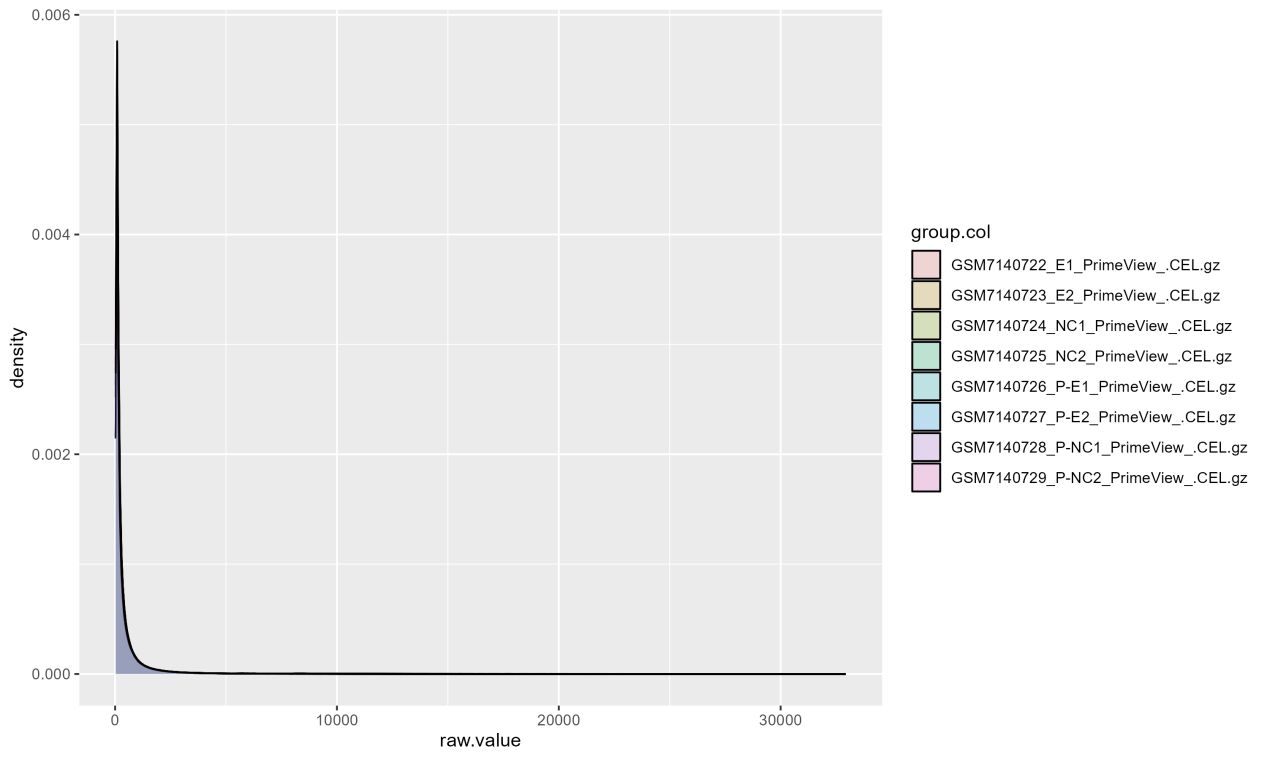
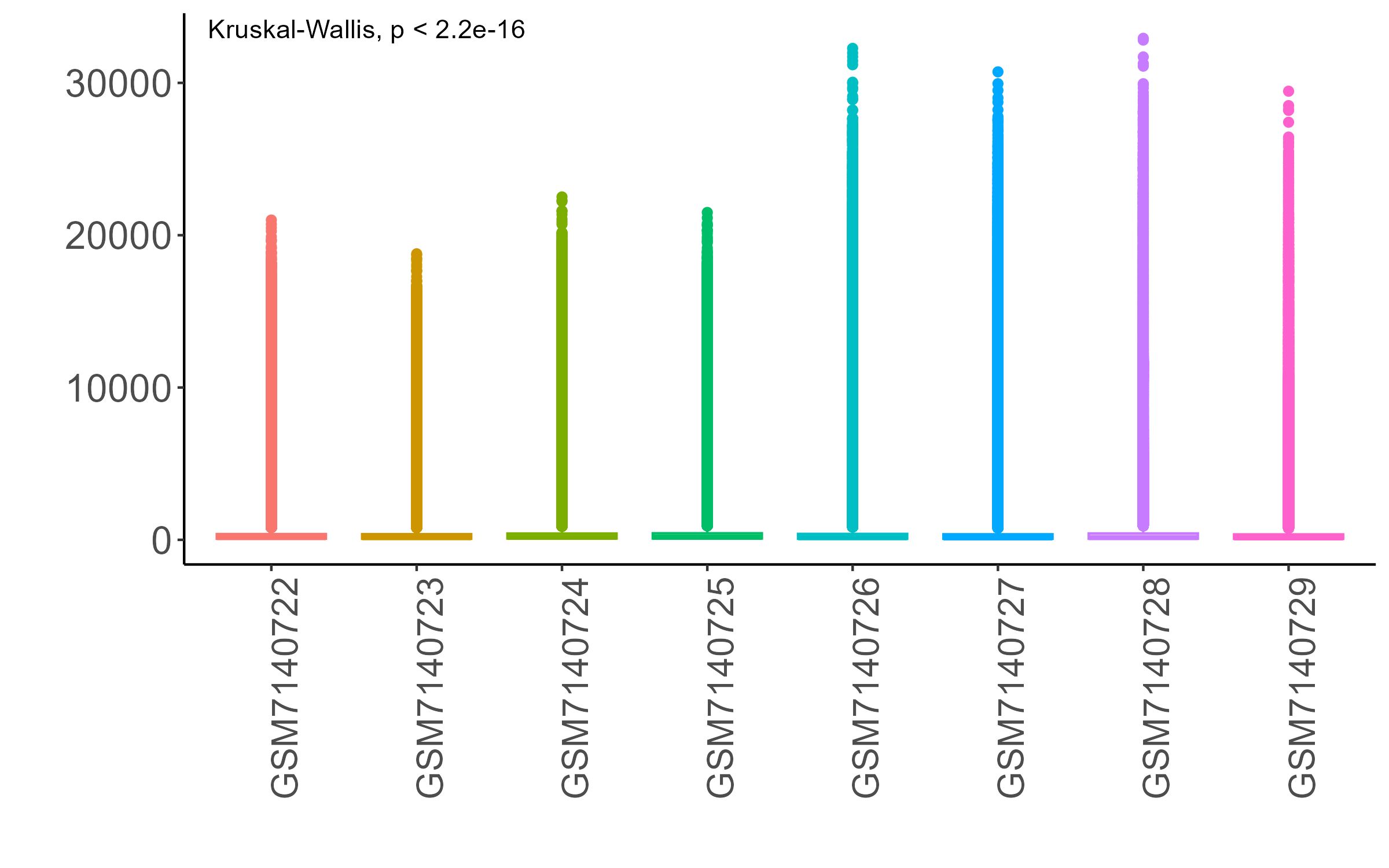
看不到中位数,数据最小值和最大值差别大,这样的数据是必须要经过标准化的。
取log2的结果
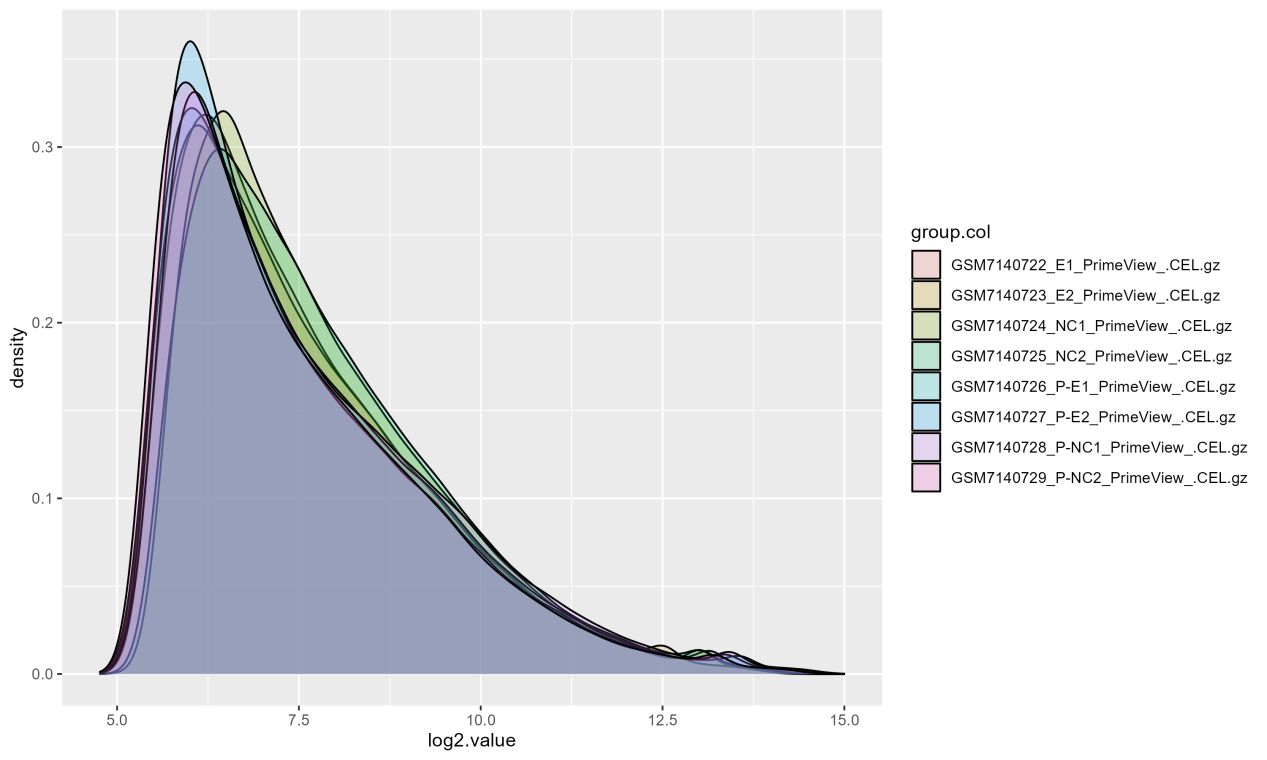

log2标准化之后样本中的表达值被缩放到了0-15之间,极差缩小了,表达值在一个数量级内了,但是中位数没有被矫正在同一水平线上,样本间的可比性还不太好。
RMA归一化处理后的结果
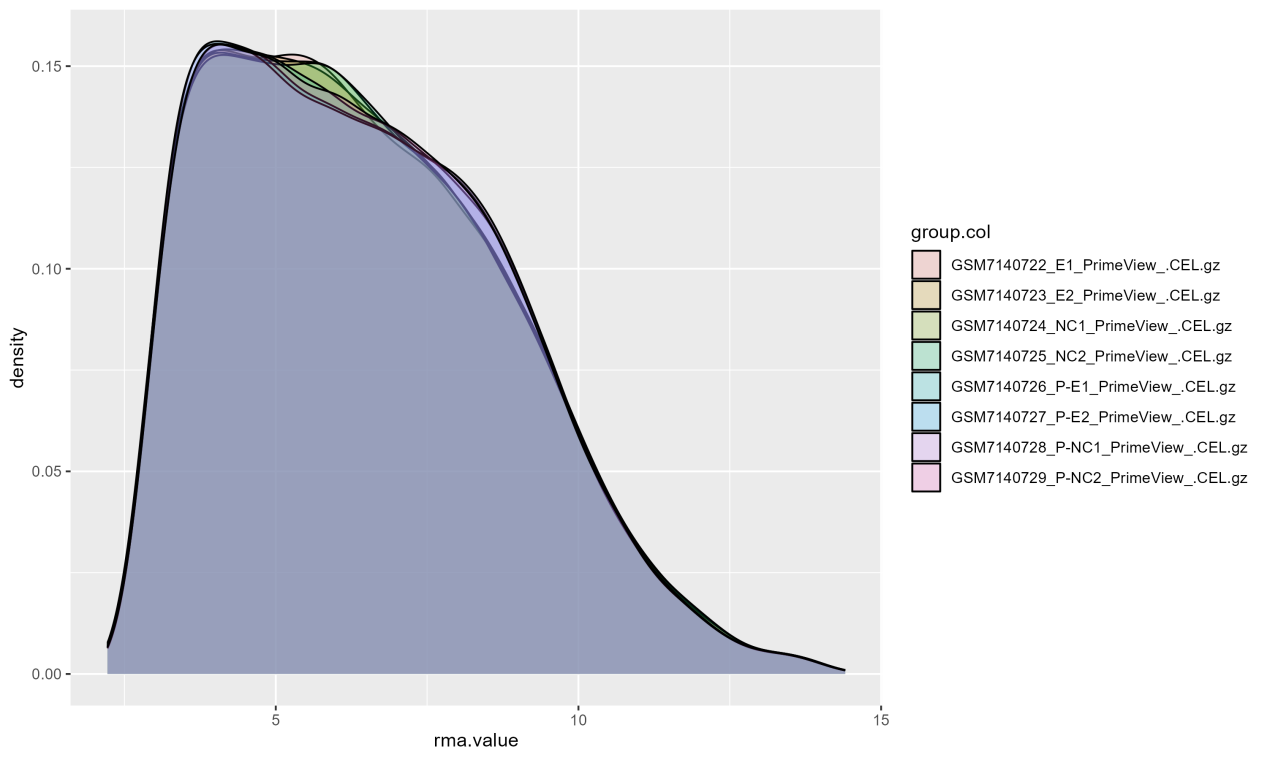
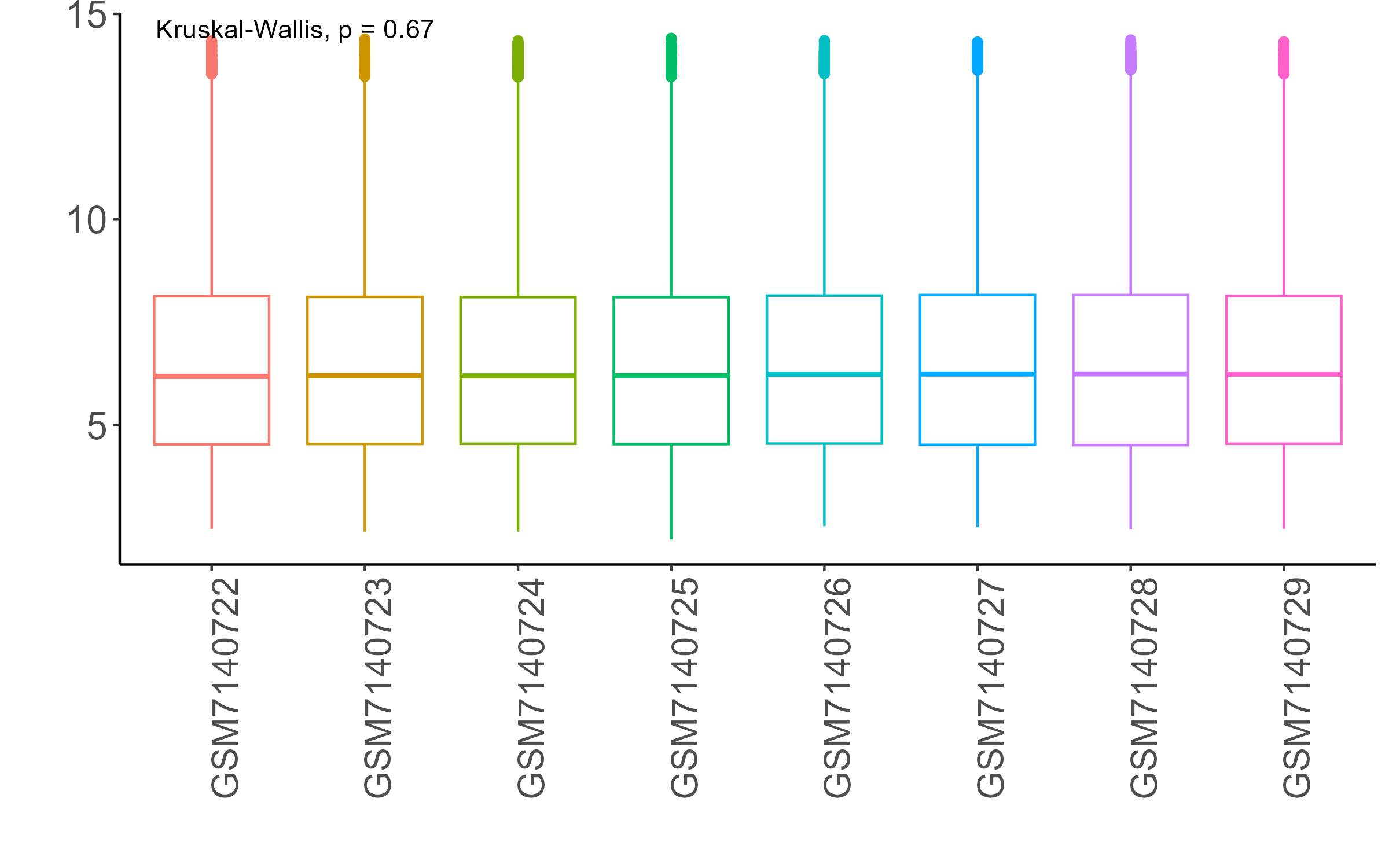
RMA标准化之后样本中的表达值被缩放到了0-12之间,极差缩小了,表达值在一个数量级内了,中位数被矫正在同一水平线上,比log2方法好很多,样本间的可比性变好了,适合用RMA标准化后的数据做后续的差异分析和可视化作图。
样本整合后的表达矩阵
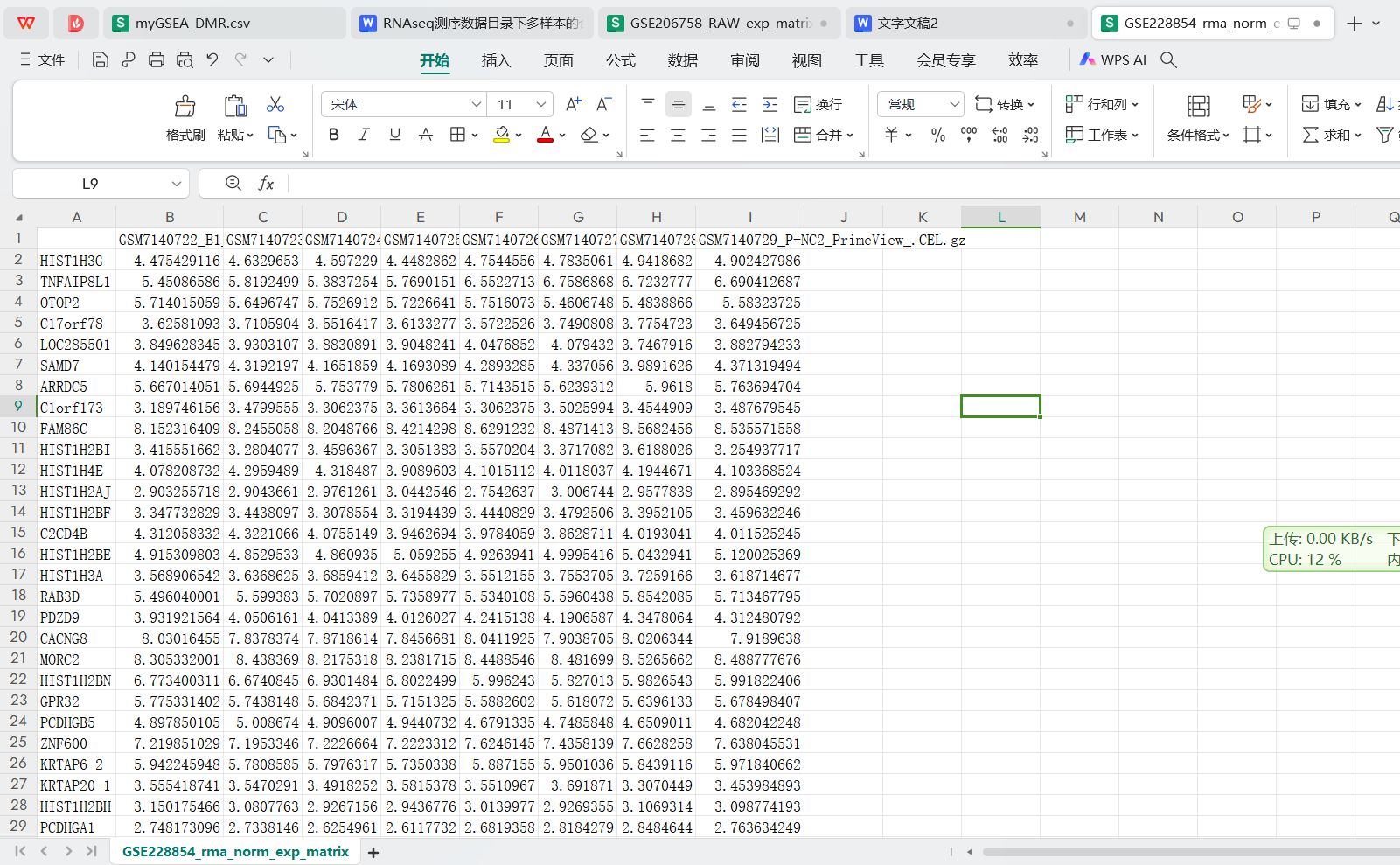
对GSEXXX_RAW.tar解压后的GSEXXX_RAW目录下非CEL芯片数据的多样本的合并处理
运行窗口
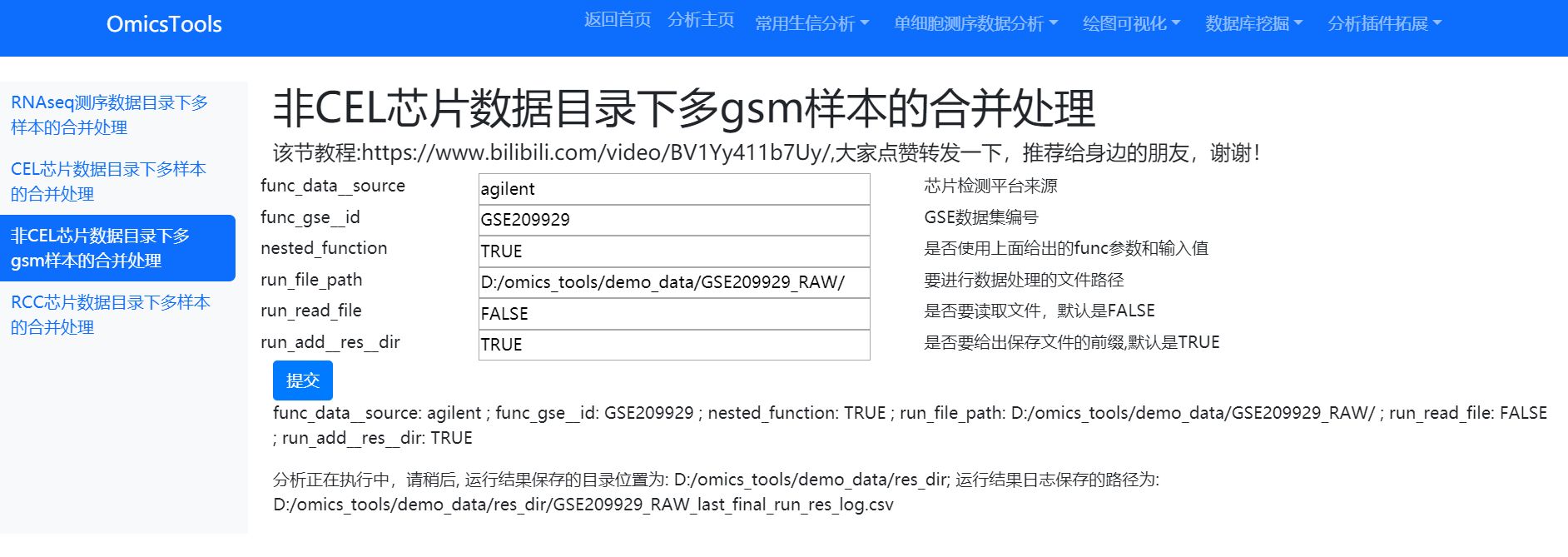
D:/omics_tools/demo_data/GSE209929_RAW/目录中的文件内容
运行中的状态信息
分析正在执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 运行结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE209929_RAW_last_final_run_res_log..csv
运行完成的状态信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\GSE209929_RAW_last_final_run_res_log.csv
运行完成的结果展示
结果文件列表
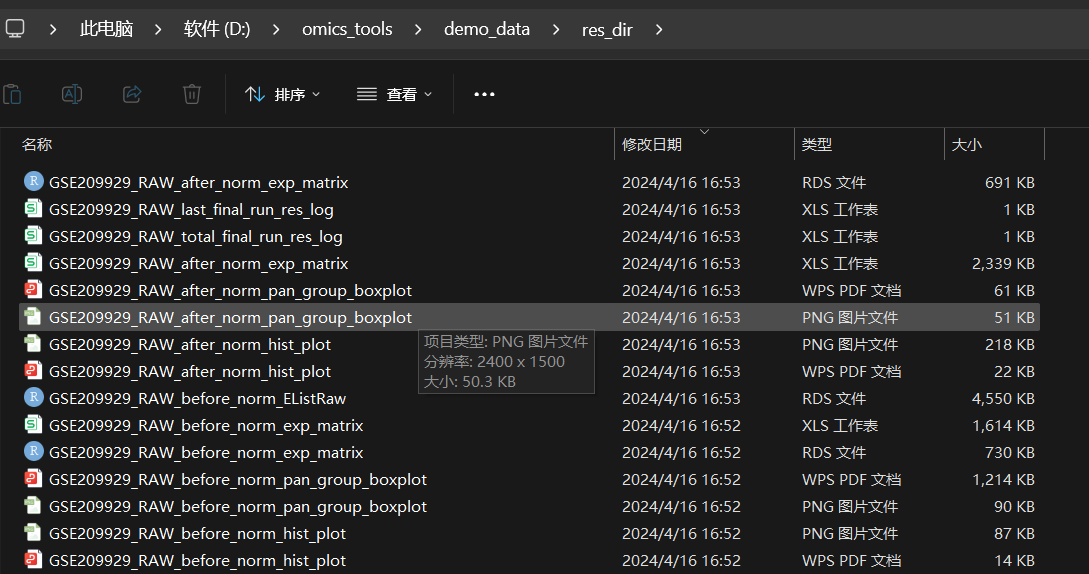
未标准化前的表达数据分布
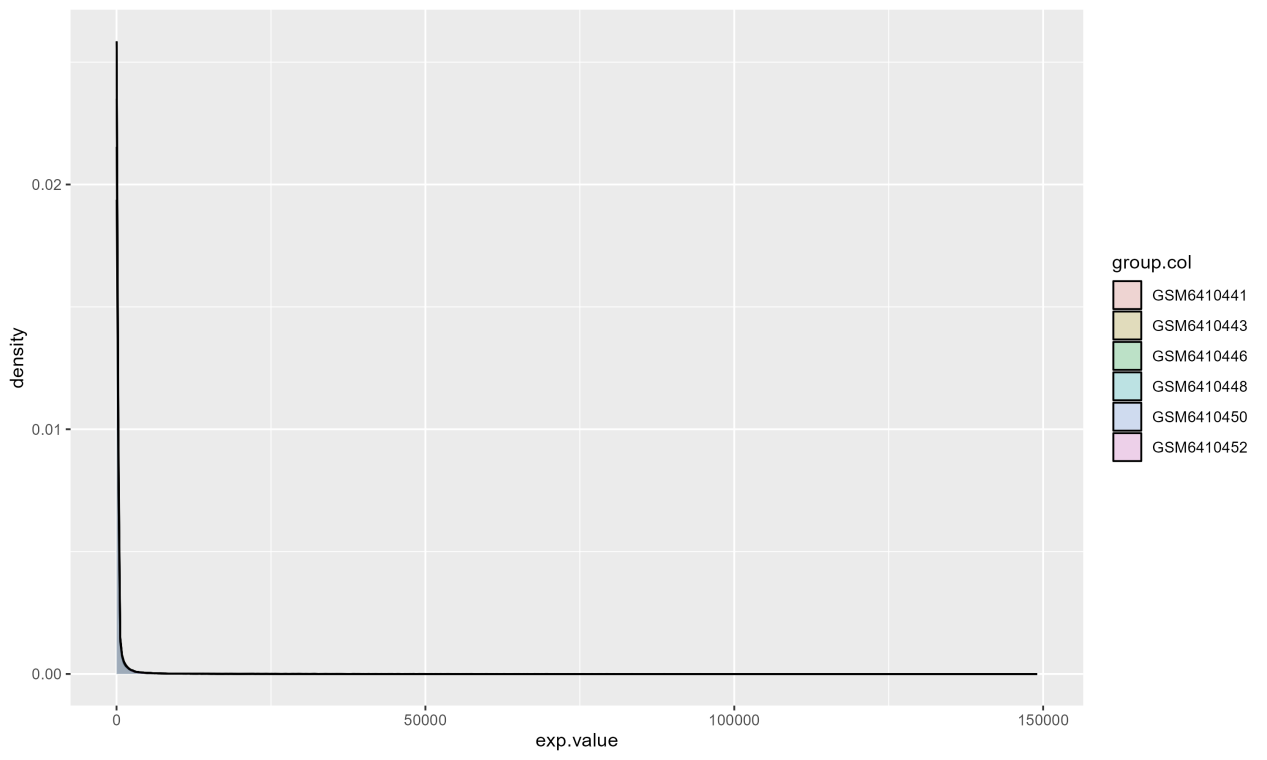
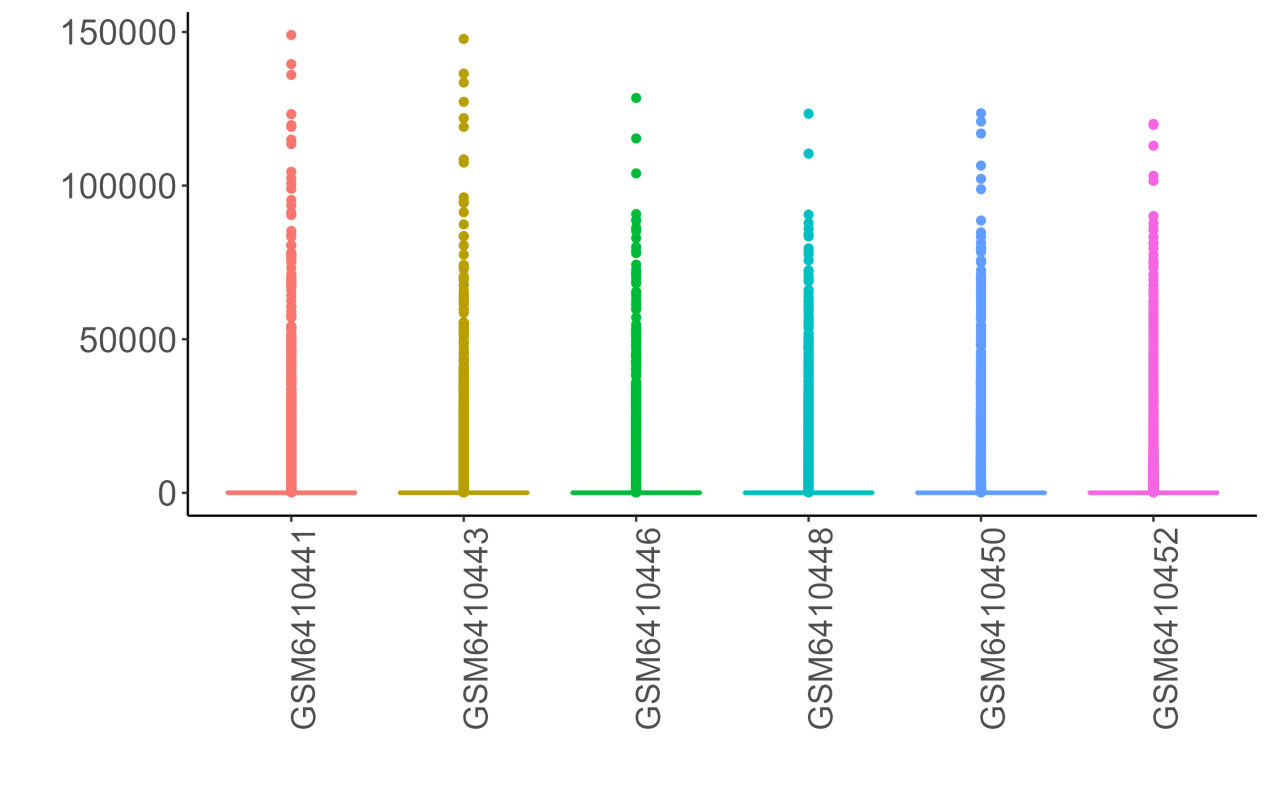
看不到中位数,数据最小值和最大值差别大,这样的数据是必须要经过标准化的。
标准化之后的数据分布
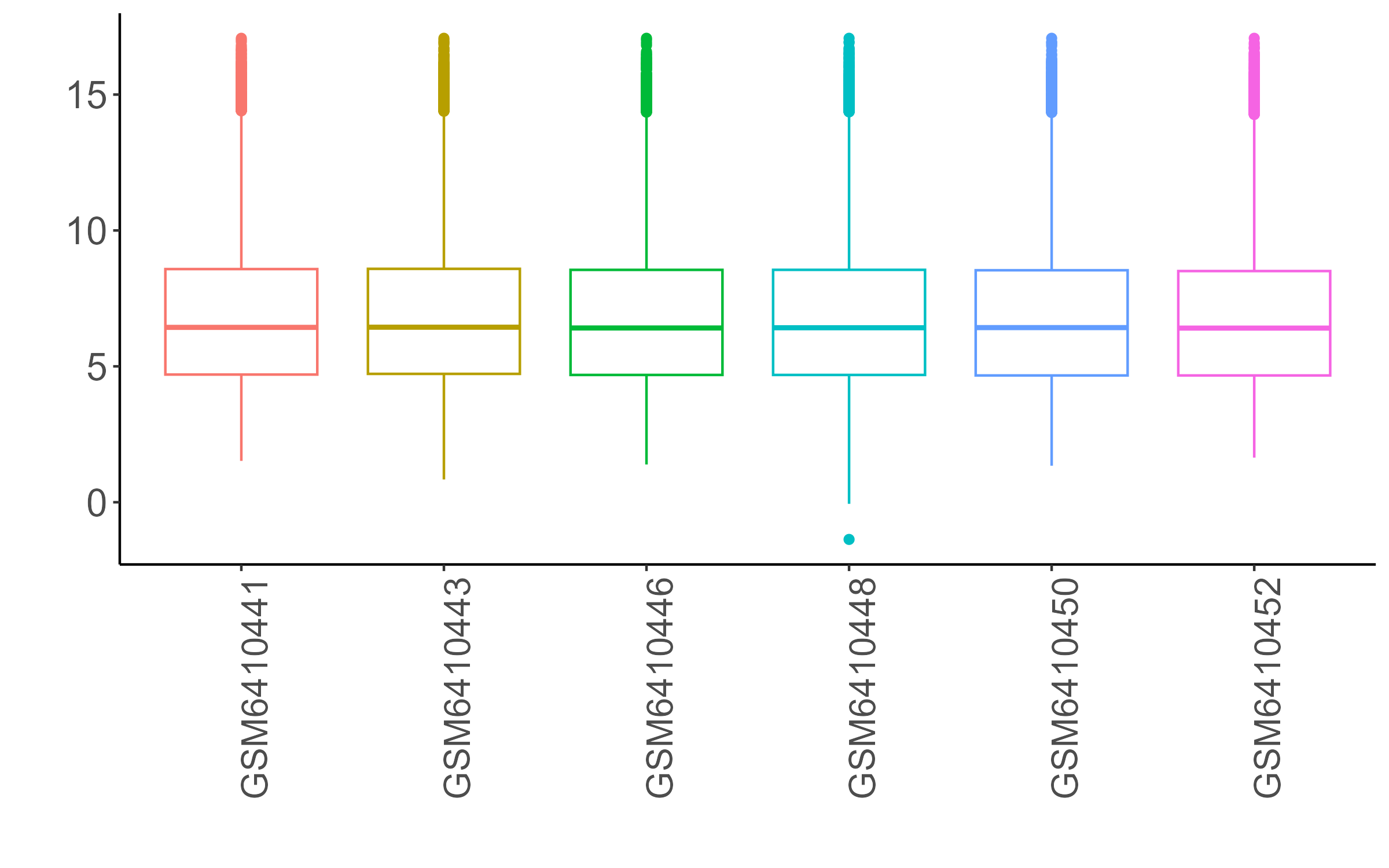

标准化之后样本中的表达值被缩放到了0-15之间,极差缩小了,表达值在一个数量级内了,但是中位数被矫正在同一水平线上,样本间的可比性变好了。
标准化后整合的表达矩阵
对GSEXXX_RAW.tar解压后的GSEXXX_RAW目录下RCC芯片数据的多样本的合并处理
运行窗口展示
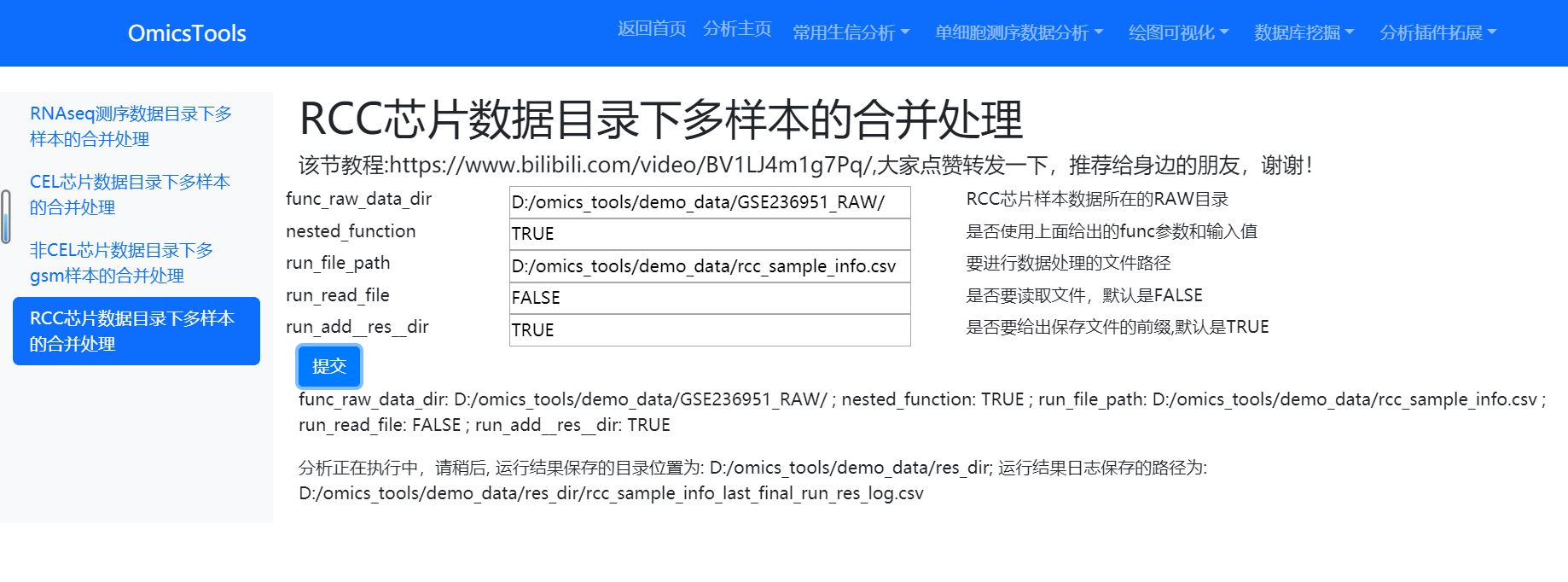
D:/omics_tools/demo_data/GSE236951_RAW/中的文件内容
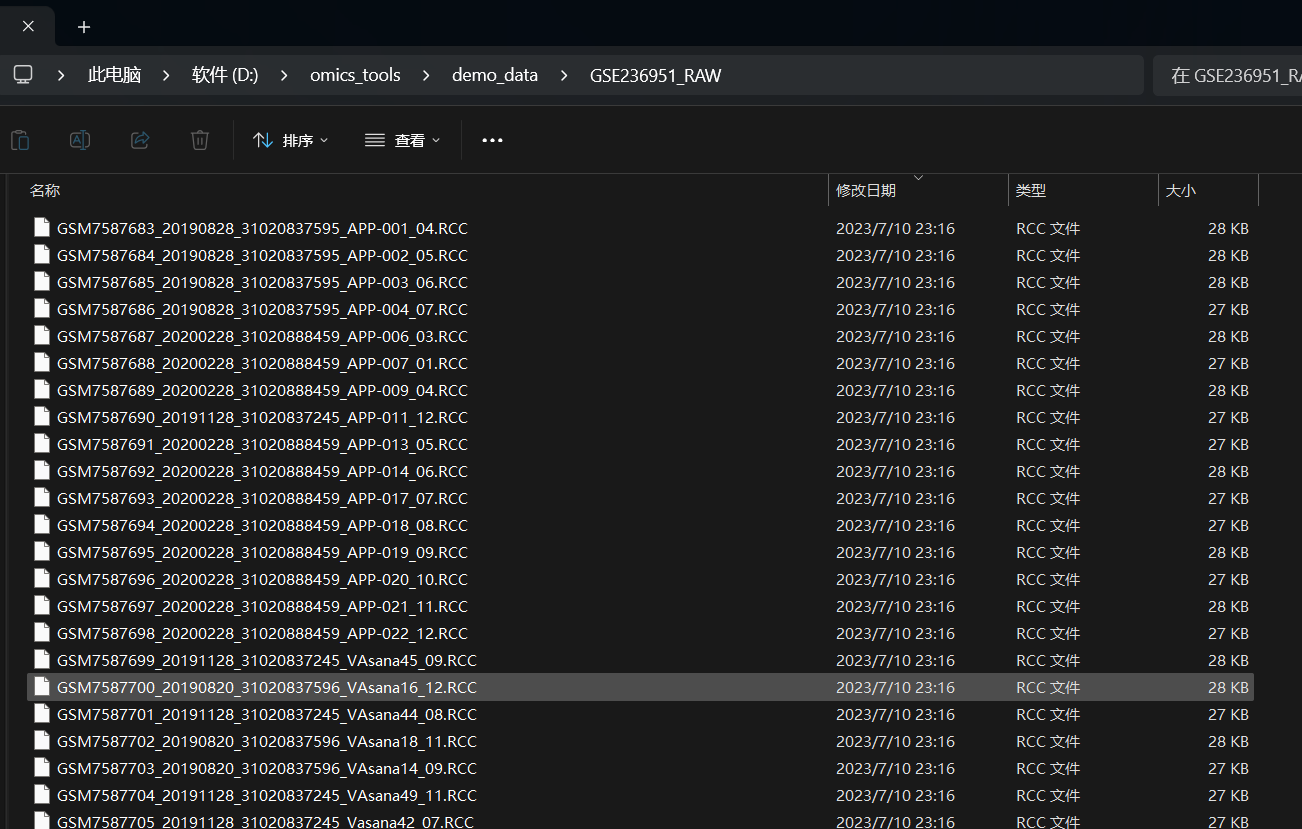
D:/omics_tools/demo_data/rcc_sample_info.csv中的文件内容
运行状态显示信息
执行中,请稍后, 运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\rcc_sample_info_last_final_run_res_log.csv
运行完成显示信息
执行已完成,运行结果保存的目录位置为: D:/omics_tools/demo_data\res_dir; 分析结果日志保存的路径为: D:/omics_tools/demo_data\res_dir\rcc_sample_info_last_final_run_res_log.csv
运行结果文件列表
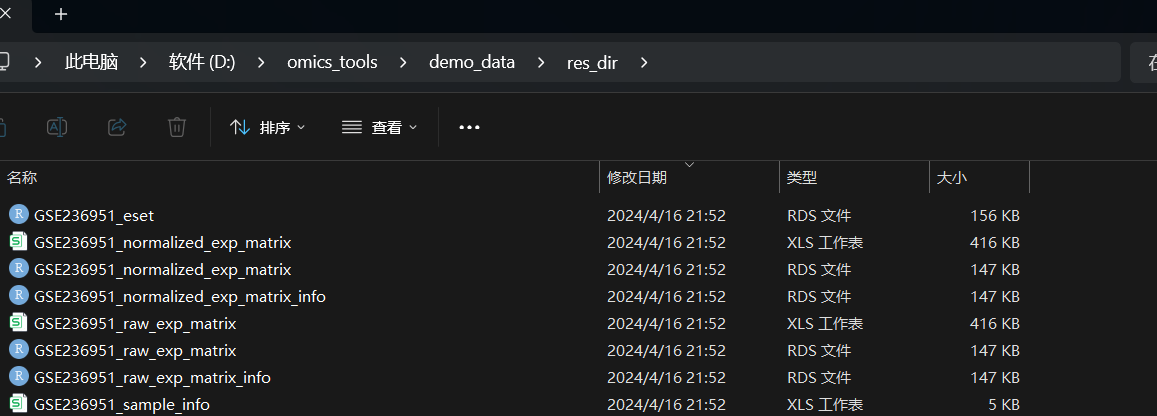
多样本整合标准化后的表达矩阵
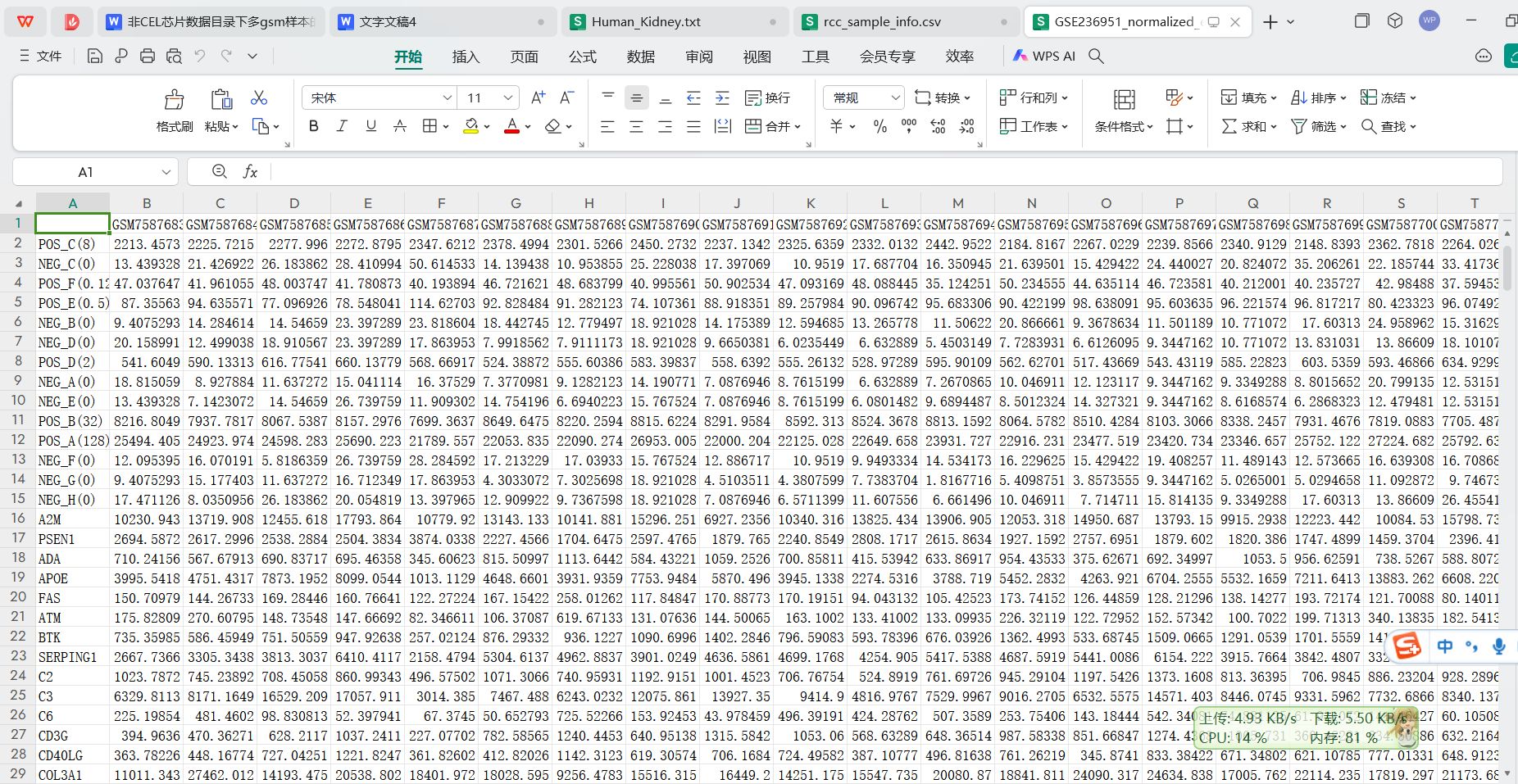




























 1214
1214

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










