很久很久以前,看到过文章中的KEGG富集可视化结果图。是对KEGG通路进行注释的。后来在一些测序公司的宣传页上也见到过类似的图:

image.png
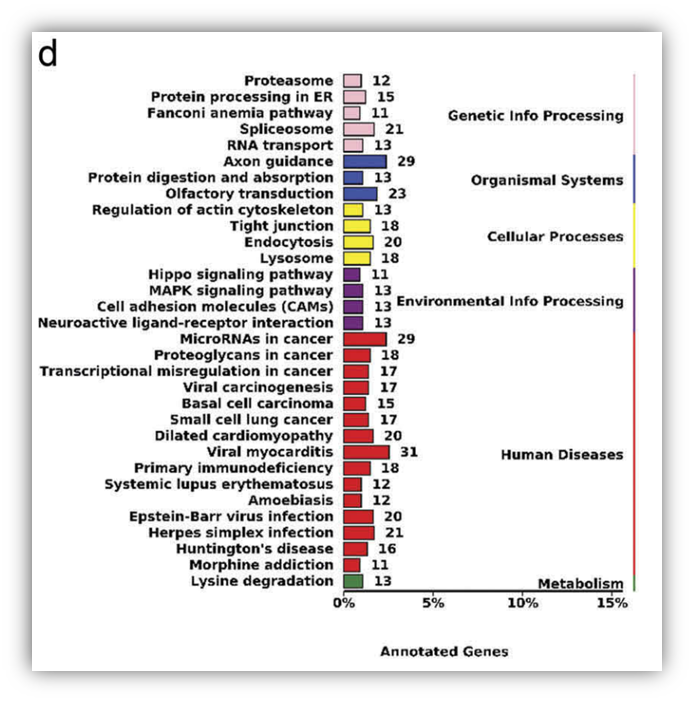
image.png
其实这个图就是多了一个KEGG通路注释,近期也有小伙伴寻求怎么做。网上很多在线工具可以完成这个图,只需要按照相应的示例提供数据就可以一键出图,很方便。用到的数据就是分析完成的KEGG富集通路,关键的两列一个是KEGG同类名称,一列是counts,当然这个counts也可以选择展示怕值。例如这里的这个网站:微生信-免费在线绘制pathway富集结果分类图
出图的效果如下:

image.png
当然我们还是希望自己动手做出来,毕竟也不是什么难事。需要的文件一个是我们分析的富集通路文件,另外一个就是KEGG注释分类,这个文件在KEGG官网可以整理,这里我们已上传分享到QQ群文件了,可自行下载!
首先加载包和数据,做一个普通的柱状图很简单:
# setwd('D:/KS项目/公众号文章/KEGG通路注释')
library(dplyr)
library(ggplot2)
library(forcats)
library(dittoSeq)
kegg <- read.csv('KEGG.csv', header = T)
#其实做一个柱状图没有什么难度
ggplot(kegg,aes(Description, Gene.Count))+
geom_bar(stat = "identity")+
geom_text(aes(label=Gene.Count, y=Gene.Count+2),size=3)+
coord_flip()+
labs(x='',y='Gene count')+
theme_bw()+
theme(panel.grid = element_blank(),
legend.position = 'none',
axis.ticks.y = element_blank(),
axis.text = element_text(colour = 'black', size = 10))
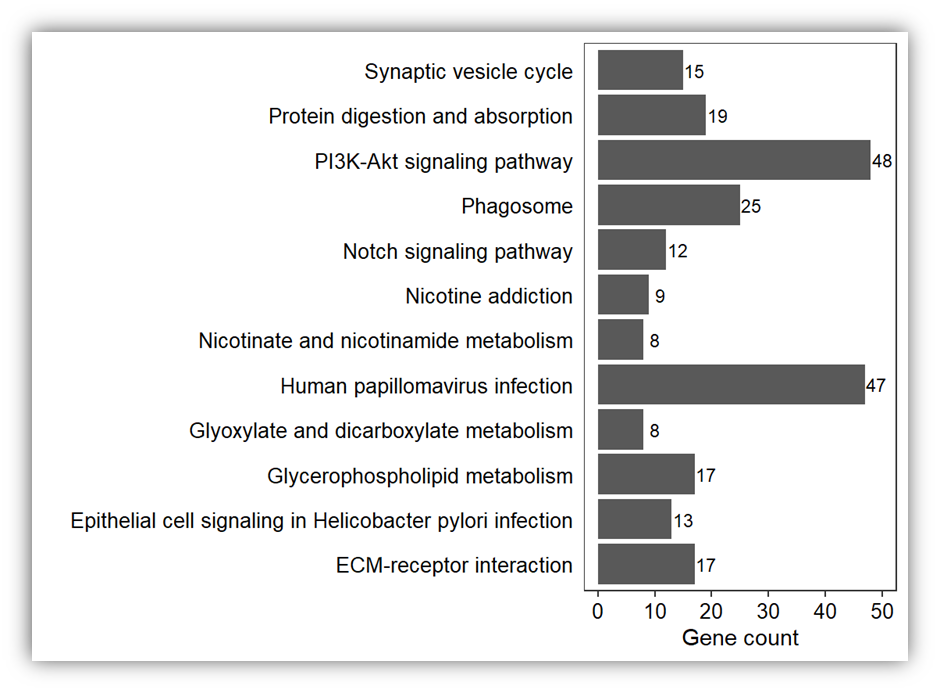
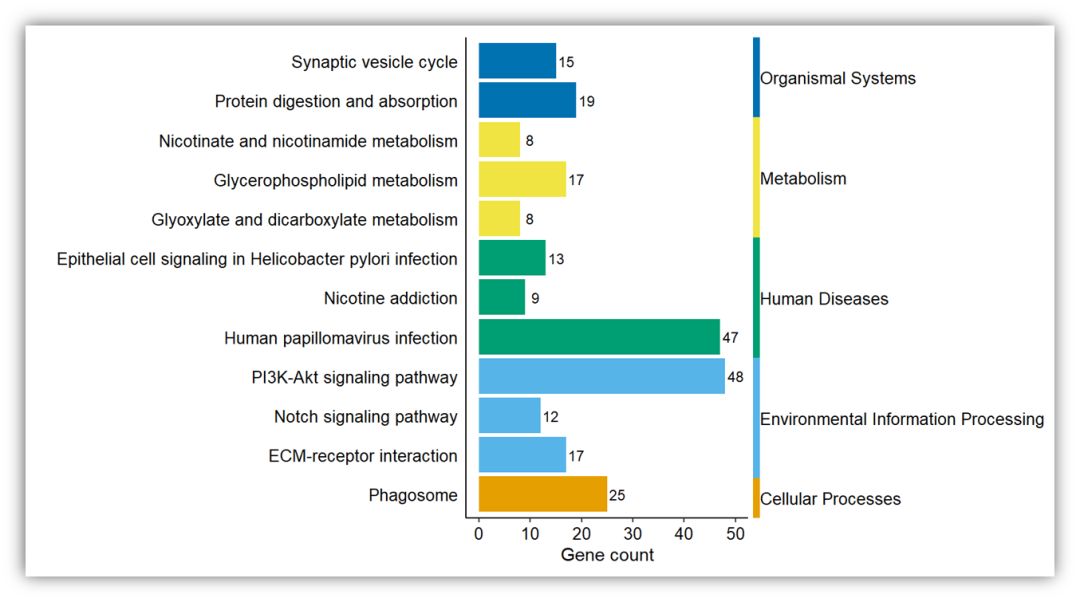
然后读入注释文件,将通路注释,这里使用的笨办法,直接筛选:
#注释文件
kegg_ann <- read.csv('kegg_annotation.csv')
rownames(kegg_ann) <- kegg_ann$directory3
df <- kegg_ann[kegg$Description,]
kegg1 <- cbind(kegg, df)
table(kegg1$directory1)
# Cellular Processes Environmental Information Processing
# 1 3
# Human Diseases Metabolism
# 3 3
# Organismal Systems
# 2
kegg1 <- kegg1[order(kegg1$directory1),]
kegg1$Description <- as.factor(kegg1$Description)
kegg1$Description <- fct_reorder(kegg1$Description)
p <- ggplot(kegg1,aes(Gene.Count,Description))+
geom_bar(stat = "identity", aes(fill=directory1))+
geom_text(aes(label=Gene.Count, x=Gene.Count+2),size=3)+
labs(y='',x='Gene count')+
theme_classic()+
theme(panel.grid = element_blank(),
legend.position = 'none',
axis.ticks.y = element_blank(),
axis.text = element_text(colour = 'black', size = 10),
plot.margin = margin(0,0,0,-0.05, "cm"))+
scale_fill_manual(values = dittoColors())
最后添加上右侧的注释就完成了:
#构建注释
df1<-data.frame(x="A", y=kegg1$Description, group=kegg1$directory1)
df1$group <- factor(df1$group, levels = c("Organismal Systems",
"Metabolism",
"Human Diseases",
"Environmental Information Processing",
"Cellular Processes"))
p1 <- ggplot(df1,aes(x,y, fill=group))+
geom_tile(show.legend = F)+
facet_grid(group~.,scales = 'free',space = 'free')+
labs(x=NULL,y=NULL)+
scale_x_discrete(expand = c(0,0))+
scale_y_discrete(expand = c(0,0))+
theme(panel.background = element_blank(),
axis.ticks = element_blank(),
axis.text = element_blank(),
plot.margin = margin(0,-0.05,0,0, "cm"),
strip.text.y = element_text(angle=0,size=10,color = "black",
hjust = 0,margin = margin(b = 3,t=3)),
strip.background = element_rect(colour=NULL,fill = 'white'),
panel.spacing=unit(0, "mm"))+
scale_fill_manual(values = c("#0072B2","#F0E442","#009E73","#56B4E9","#E69F00"))
#拼图
library(deeptime)
ggarrange2(p, p1,nrow = 1,widths =c(2,0.05))
当然了,网上也有别的形式的注释,这里不再演示,大概样子就是在柱状图y轴添加了分类的名称,有了注释文件就可以构造了。具体的做法可以参考我们之前的帖子:柱状图|GO、KEGG|标签与柱状图颜色对应。
那么最后还有一个问题,很多人肯定会想到,就是GO的结果可以注释嘛?我们知道GO分为BP、MF、CC三个,但是在BP中能不能继续注释,我是没有看到官方的文件,但是需要自己注释和作图可以参考我们这个帖子。希望分享对你有帮助,点个赞、分享下再走呗!





















 6042
6042











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








