CoGAPS 的全称是 Coordinated Gene Activity in Pattern Sets。它实现了一种贝叶斯马尔可夫链蒙特卡罗(MCMC)矩阵分解算法,称为 GAPS(Gene Activity Pattern Sets),并将其与基因集统计方法连接起来,以推断生物过程的活性。CoGAPS 可以对任何数据进行稀疏矩阵分解,当这些数据代表生物分子时,它还可以进行基因集分析。
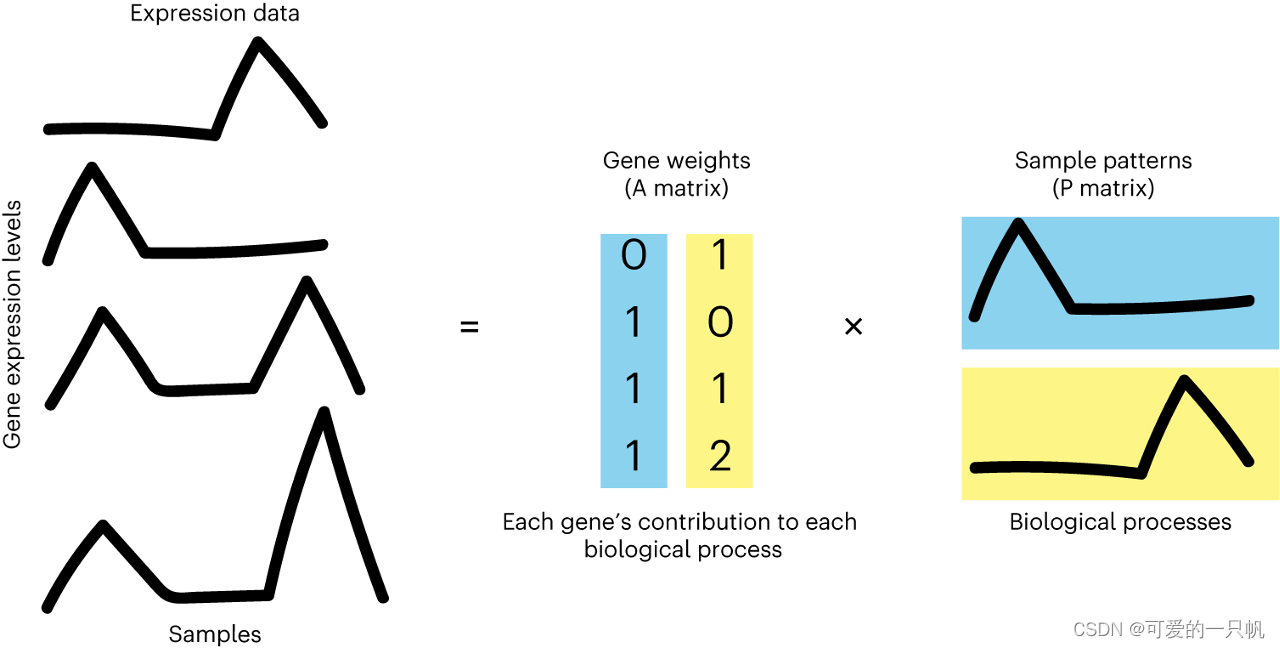
使用方法:
1.创建conda环境安装pycogaps
conda create -n pycogaps
git clone <a href="https://github.com/FertigLab/pycogaps.git" target="_blank">https://github.com/FertigLab/pycogaps.git</a> --recursive
cd pycogaps
pip install -r requirements.txt
python3 setup.py install安装成功后会显示:
Finished processing dependencies for pycogaps==0.0.12.数据集准备
我的数据集是seurat文件,写个函数把seurat文件转换为h5ad文件。
subset_and_convert_seurat <- function(seurat_obj, output_h5ad_path) {
seurat_obj_subset <- subset(seurat_obj, features = features)
h5seurat_file <- tempfile(fileext = ".h5Seurat")
SaveH5Seurat(seurat_obj_subset, filename = h5seurat_file,overwrite = TRUE)
Convert(h5seurat_file, dest = output_h5ad_path, overwrite = TRUE, assay="RNA")
unlink(h5seurat_file)
}
subset_and_convert_seurat(myscdata, "./myscdata.h5ad")
3.进入虚拟环境运行python
conda activate pycogaps
cd pycogaps
pythona.导入模块
from PyCoGAPS.parameters import *
from PyCoGAPS.pycogaps_main import CoGAPS
import scanpy as scb.导入数据集
scpath = "./myscdata.h5ad"
adata = sc.read_h5ad(scpath)c.修改数据输入格式,官方建议运行pycogaps之前进行log-normalization
sc.pp.log1p(adata)
adata.X = adata.X.todense()
sc.pp.log1p(adata)
adata = adata.T
adatad.设置运行参数,nIterations迭代次数,seed随机数,nPatterns设置分成多少pattern,建议先使用cNMF包确定最佳pattern值
params = CoParams(adata=adata)
setParams(params, {
'nIterations': 10000,
'seed': 42,
'nPatterns': 8,
'useSparseOptimization': True,
'distributed': "genome-wide"
})
# 设置并行参数
params.setDistributedParams(nSets=12)e.运行并保存
start = time.time()
result = CoGAPS(adata, params)
end = time.time()
print("TIME:", end - start)
result.X = np.asarray(result.X)
result.write("./sc_result.h5ad")f.分析及可视化
建议分析及可视化在GenePattern进行
from PyCoGAPS.analysis_functions import *
plotPatternUMAP(result)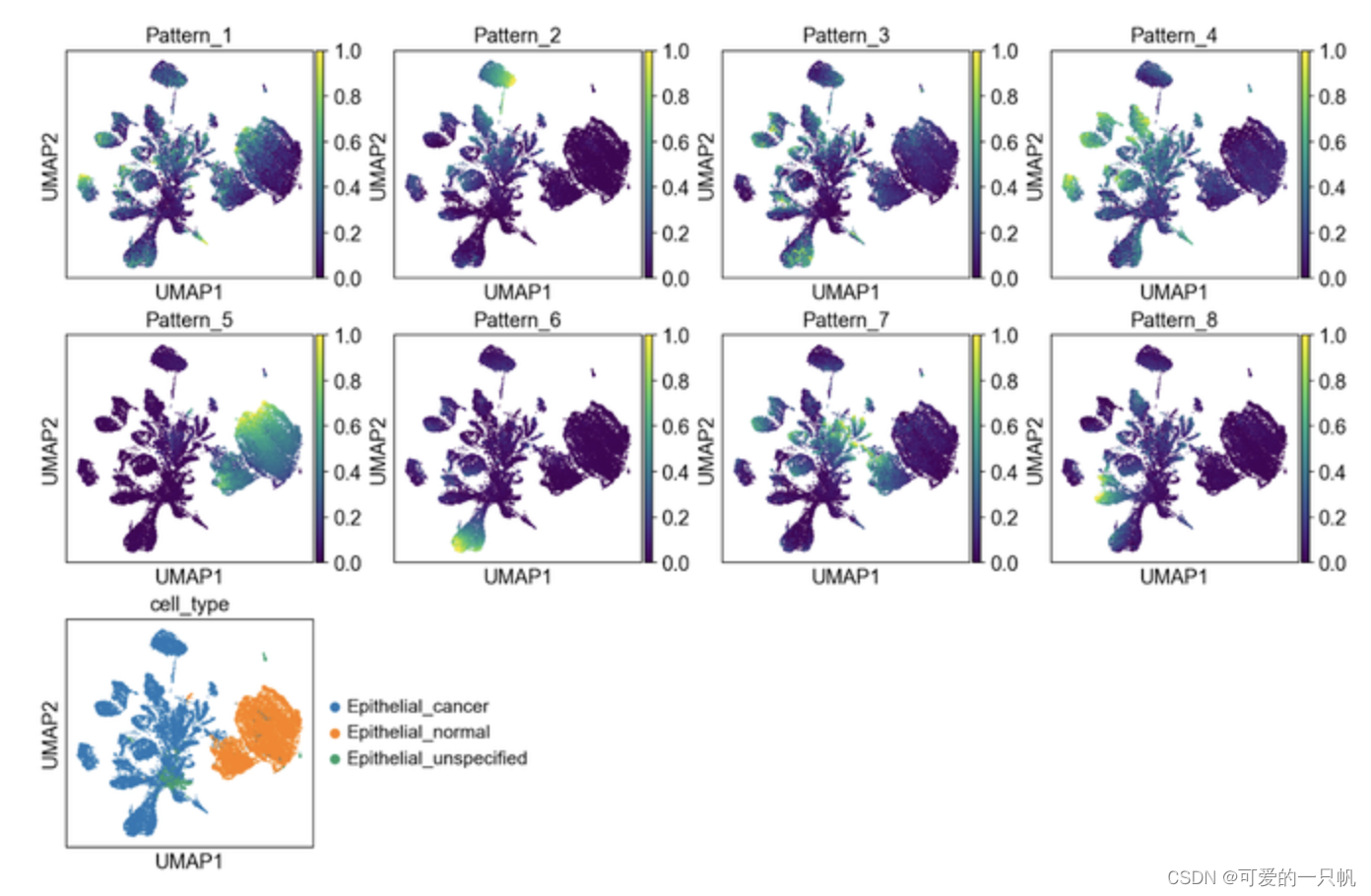
可提取pattern特异性基因做富集分析:
pm = patternMarkers(cogapsresult, threshold="cut")
pm["PatternMarkers"]["Pattern7"]参考:Nature Protocols, 2023, 18, 3690–3731.
https://fertiglab.github.io/CoGAPSGuide/





















 1104
1104

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








