菲沙基因的拳头产品之一-三维基因组广受大家欢迎,其中有很多老师在菲沙基因进行了ATAC-seq和CUT&Tag,这两个技术的重要结果文件之一是可用于可视化浏览的peak文件(bw、narrowPeak文件),为了让更多人知道如何使用此文件进行可视化,今天小编就给大家详细演示下如何利用IGV软件对我们的peak文件进行全基因组“点哪看哪”的可视化步骤。
IGV是什么?
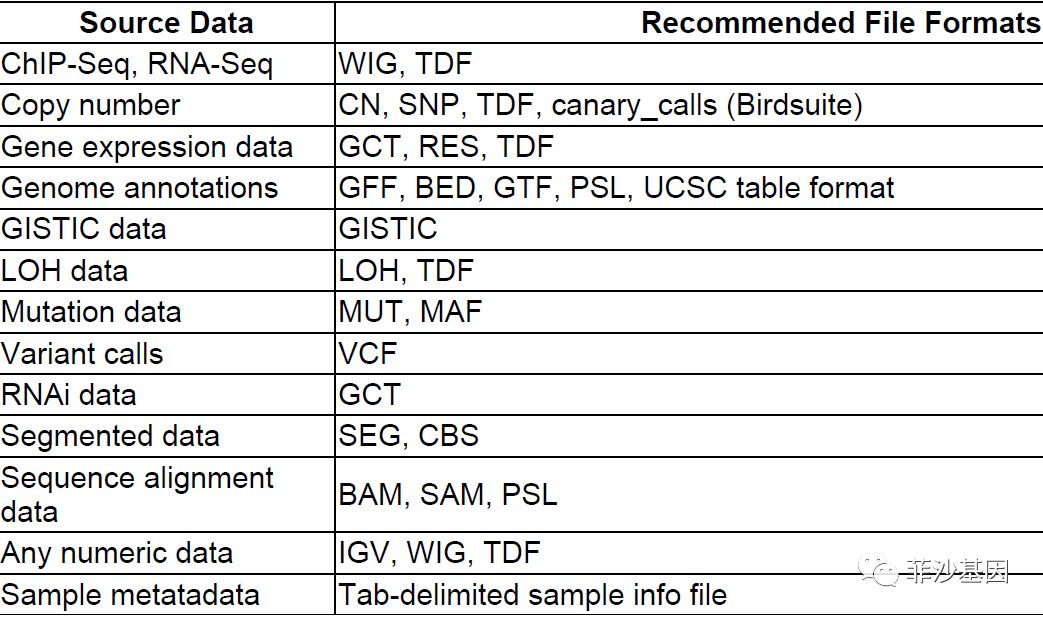
IGV(Integrative Genomics Viewer)是一款本地的探索基因组数据的可视化浏览器,它拥有多个系统版本,支持多种不同类型的输入格式,包括芯片测序、二代测序、基因组注释文件等,如下表所示文件均可使用IGV查看。相关网址如下:
IGV官网:
http://www.igv.org
IGV下载网址:
http://software.broadinstitute.org/software/igv/download
JAVA 下载:
https://www.java.com/en/download/mac_download.jsp
完整的官方说明:
http://software.broadinstitute.org/software/igv/book/export/html/6
IGV实操演示
下面为大家展示IGV的操作步骤。
1. 打开IGV
先按提示安装IGV,在短暂的小窗口黑屏后会进入导入基因组的进度条,请耐心等候IGV界面。
2. 进入IGV界面
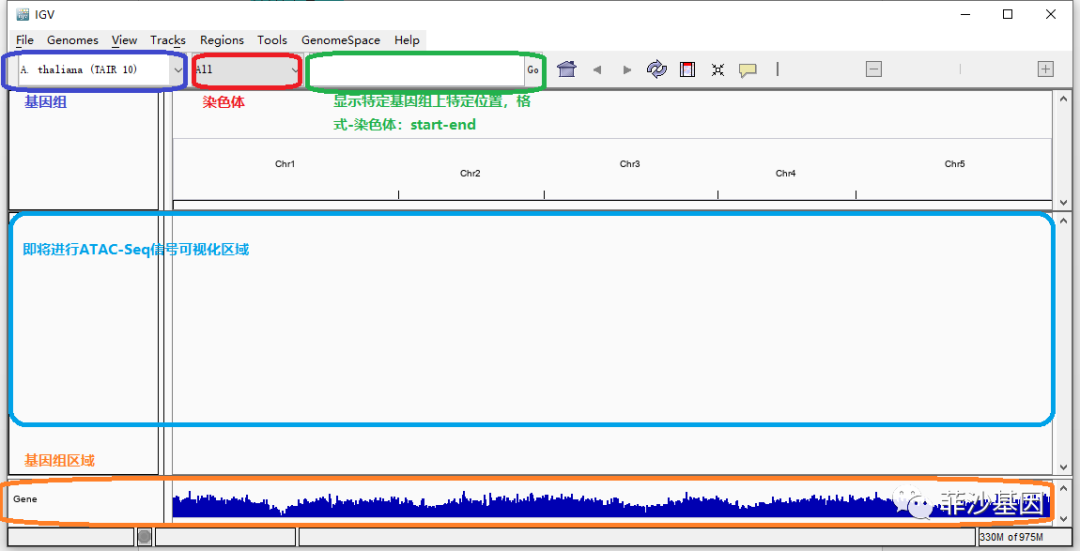
从整个界面来看,主要有5个区域,分别如下图所示:选择基因组区域、选择染色体区域、输入特定的基因组位置区域、信号可视化区域、基因注释区域。
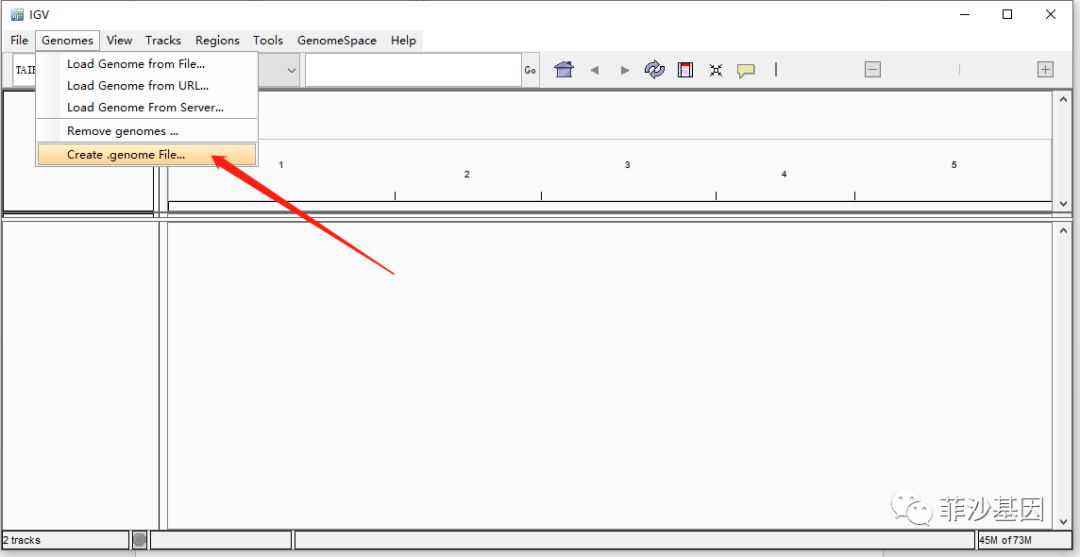
3. 导入基因组
导入基因组分为自动导入和手动导入。自动导入则是导入IGV内置的物种基因组,如果待分析的基因组不在内置中,则需要手动导入。
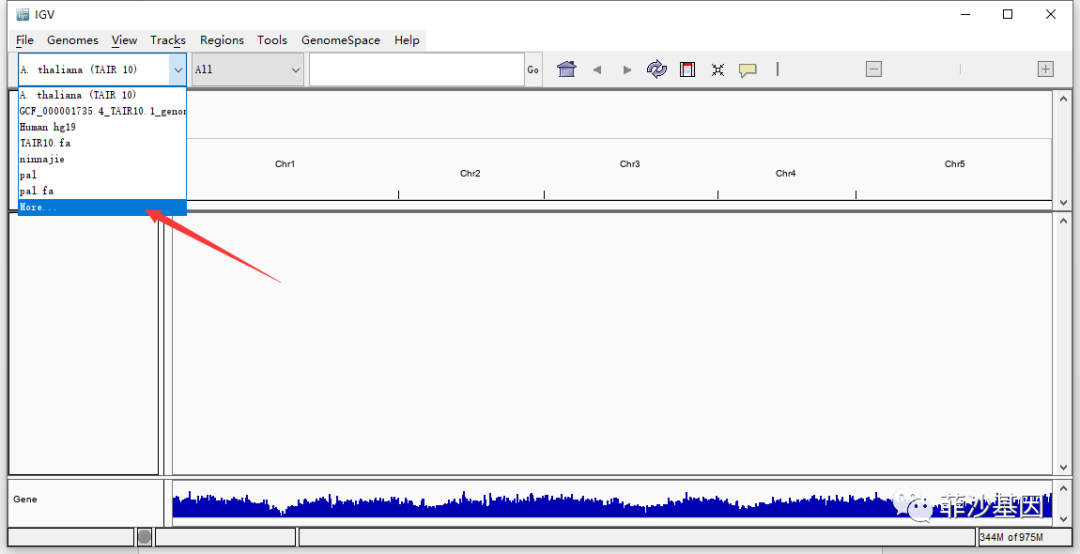
自动导入基因组:点击More,寻找对应的物种基因组。目前IGV内置基因组共包含156个物种,详情请点击查看:
http://software.broadinstitute.org/software/igv/Genomes
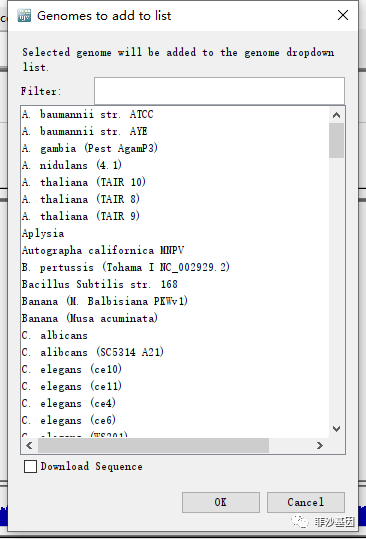
手动导入基因组:需要准备的文件有基因组序列FASTA文件(必需)、基因组注释GTF文件(非必需,格式可为BED、GFF或genePred table所有变形格式)、基因组名转化文件(非必需)。步骤如下:

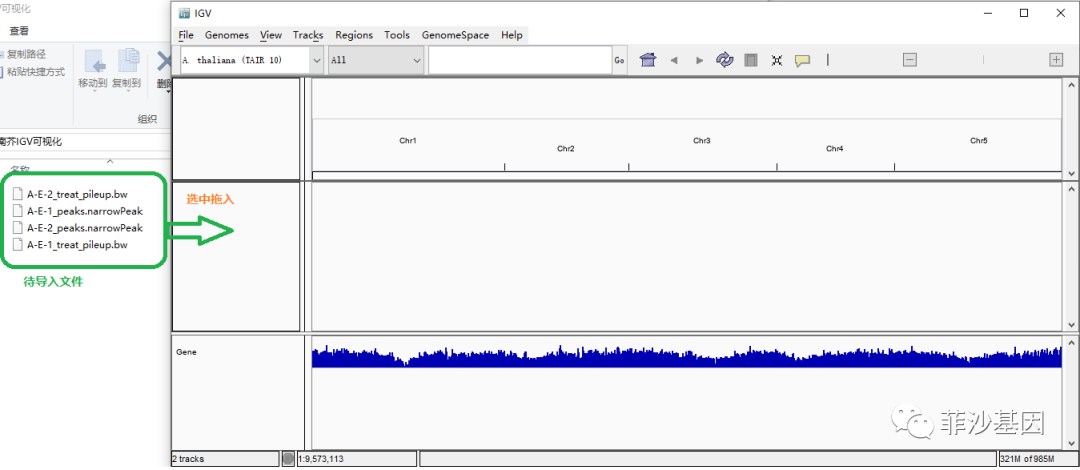
4. 导入目标文件(bw、narrowPeak)
我们交付的文件中含有①peaks.xls;②_pileup.bw;③peaks.narrowPeak。其中①可以用Excel打开查看peak信息,②③则可以选中后直接拖入IGV界面进行可视化浏览。
5. 选择目标位置进行可视化浏览
(1)选择指定染色体、输入目标位置或直接在位置区域输入基因名。点击右上角的分辨率按钮就可以对目标区域进行放大和缩小处理,也可以左右拖动信号峰。

(2)右键bw文件,可以调整一些参数,包括样品名称、信号峰的颜色、信号值的范围等。

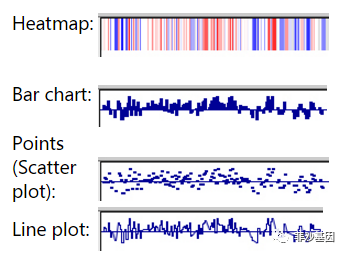
可视化的类型包含有热图、柱状图、点图、折线图,图例如下:
(3)右键Gene,可以对基因注释文件的展现形式进行调整。
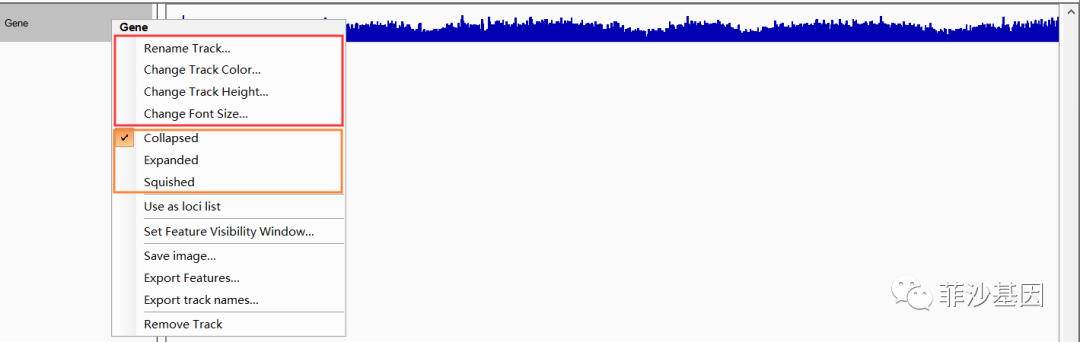
基因注释文件包含三种可视化类型,图例如下:

(4)将多个样本的信号值放在一起进行比较
如下图所示,将一个样本的信号峰设成粉红色,另一个样本的信号峰设成蓝色,信号值高度均设置为100,形式为柱状图,范围保持一致(Group Autoscale)即可直观的进行比较。
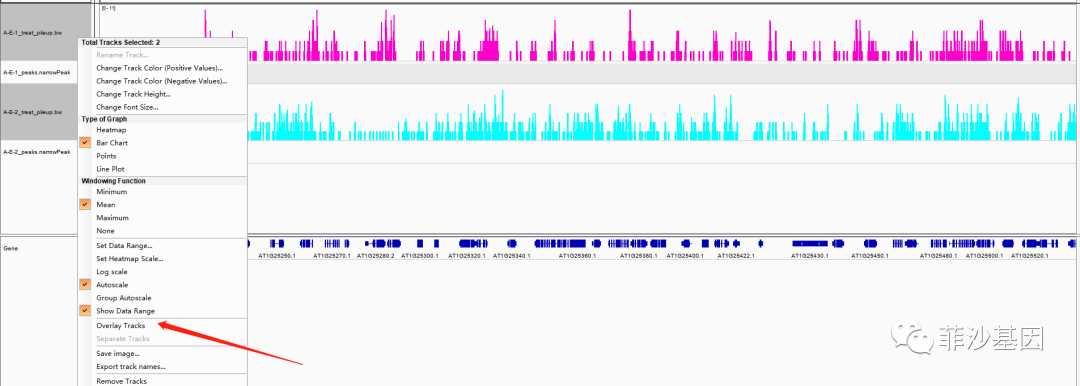
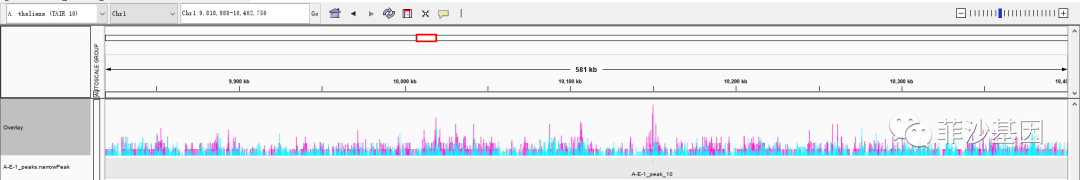
如果要将信号值合并展示,可以选中两个bw文件后右键Overlay Tracks即可实现。
6. 保存图片
点击File/Save image即可保存当前视野中的图片。
以上就是使用IGV查看ATAC-seq或CUT&Tag peaks的基本操作步骤,是不是很简单呀?此外,如果是Hi-C数据,我们还有基于WashU的 Hi-C Studio可视化浏览器,复杂的数据,可以简单看哦!赶快联系菲沙基因开展三维基因组研究吧。
你可能错过的精彩:
官宣|清华大学张奇伟团队与菲沙基因正式签署合作协议
科研合作丨新年伊始,菲沙基因携手清华大学颉伟团队,开启三维低起始量研究新篇章
No.1增强子丨增强子链中更重要的增强子距离启动子更远
Communications Biology丨ATAC-seq探索全球气候变暖破坏珊瑚生态系统的机制
为什么一次性要做这么多ATAC-seq?
宏基因组Hi-C实战分析“避坑”操作指南























 1413
1413











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








