
这期分享一篇 2024年1月发表于 Clin Transl Med (IF 10.6)的文章,作者基于人工智能衍生的预后指数确定角鲨烯环氧化酶在高危骨肉瘤中的代谢易感性。

该文章使用桓峰基因公众号里面生信分享教程即可实现,有需要类似思路的老师可以联系我们!
摘 要
背景: 骨肉瘤(OSA)是一个临床难题,其5年生存率较低。目前,缺乏先进的分层模型使得个性化治疗变得困难。本研究旨在寻找新的生物标志物对OSA高危患者进行分层并指导治疗。
方法: 我们将10种机器学习算法组合成101种组合,从中建立了基于254个样本转录组谱预测总生存的最佳模型。并评估了转录组、基因组和表观基因组景观的改变,以阐明导致预后不良的机制。单细胞RNA测序(scRNA-seq)揭示了OSA细胞中过表达的基因作为潜在的治疗靶点,其中一个基因通过组织染色、敲低和药物抑制得到了验证。描述了多种表型的变化,包括增殖、集落形成、迁移、侵袭、凋亡、化学敏感性和体内致瘤性。RNA-seq和Western blotting分析了角鲨烯环氧化酶(SQLE)抑制对信号通路的影响。
结果: 由模型生成的人工智能衍生预后指数(AIDPI)是一种独立的预后生物标志物,优于临床病理因素和先前发表的特征。将AIDPI与临床因素合并到图中可提高预测准确性。为了方便用户,模型和图都可以在线访问。高AIDPI组患者表现出化疗耐药,伴有MYC和SQLE的过表达,mTORC1信号增加,PI3K-Akt信号中断,免疫浸润减少。ScRNA-seq结果显示,OSA细胞中MYC和SQLE高表达。在OSA患者中,SQLE表达升高与化疗耐药和较差的预后相关。在治疗上,通过降低胆固醇和抑制FAK/PI3K/Akt/mTOR通路,沉默SQLE可抑制OSA恶性肿瘤并增强化疗敏感性。此外,SQLE特异性抑制剂FR194738在体内表现出抗OSA作用,并与化疗药物表现出协同作用。
结论: 人工智能预测指数(AIDPI)是识别OSA患者高危亚群的可靠生物标志物。在这些患者中,SQLE蛋白作为代谢易感性出现,提供了具有翻译潜力的靶标。
生信分析流程
AIDPI是对以胆固醇稳态失调为特征的高危OSA患者进行分层的强有力的生物标志物,而SQLE是这些患者的代谢易感性。
沉默SQLE通过降低胆固醇和抑制FAK/PI3K/Akt/mTOR通路来阻碍OSA。
SQLE抑制剂在体内抑制OSA进展,提高化疗效果。
相关数据集选择
多组学TARGET-OSA (n = 85)下载:
RNA-SEQ,
gene-level CNV,
DNA methylome,clinical data)
GEO数据集下载:
GSE21257(n = 53),
GSE33382 (n = 82),
GSE16091 (n = 34),
GSE14827 (n = 27),
GSE87437 (n = 21),
GSE42352 ,
GSE16089 ,
GSE9967 (RNA-seq 18 patients),
GSE238110 (RNA-seq 186 samples)
细胞系多组学:CCLE-23Q2
单细胞数据集:PRJNA681896
药物基因组数据集:
GDSC_2020(v1-8.2)
GDSC_2020 (v2-8.2)
基因集选择
MYC and SQLE
生信分析方法
根据文章的分析流程提取所有的分析内容,整理出来就27个分析条目,每个条目都包括分析的内容,这些分析构成了整个文章,本文属于机器学习的临床预测分析类文章,下面我们就看看哪些分析可以利用桓峰基因公众号的教程来实现,点击分析条码就会跳转到对应公众号的教程,跟着教程做,您也能发轻松发高分,如下:
8. 单细胞差异表达基因(Seurat-FindAllmarkers);
9. 细胞系表达量与剂量反应曲线(PharmacoGx-DrugSensitivitySig);
10. 人工智能预测指数(AIDPI)十种机器学习算法:
1).selection operator (LASSO),
4).partial least squares regression for Cox (plsRcox),
6).supervised principal components (SuperPC),
8).survival support vector machine(Survival-SVM),
AIDPI的生成涉及以下步骤:

11. 单/多因素COX回归分析(ezcox, survival);
18. 列线表校正曲线(Calibration curve);
22. KEEG富集分析(clusterProfiler);
27. 基于甲基化数据计算浸润免疫细胞和基质细胞的比例(ESTIMATE);
研究结果
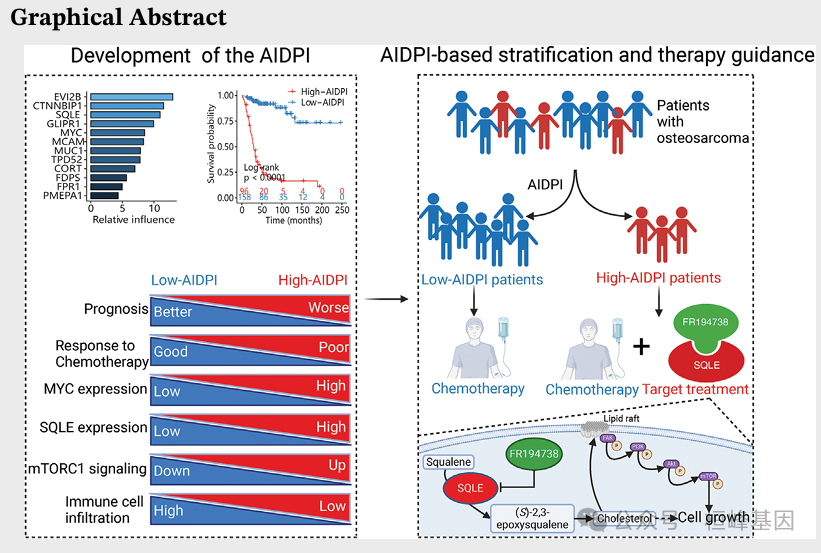
1. 人工智能衍生预后指数(AIDPI)的开发和验证
(A)三个队列中不同机器学习算法组合得到的多个模型的c-index。
(B-D和H-J) AIDPI的多队列随时间变化的ROC曲线分析。
(E-G和K-M)多队列的AIDPI Kaplan-Meier生存分析。
(N)对AIDPI和先前发表的预后特征进行单变量Cox回归分析的结果。
(O)AIDPI的C-index与Xu等人建立的模型的比较。
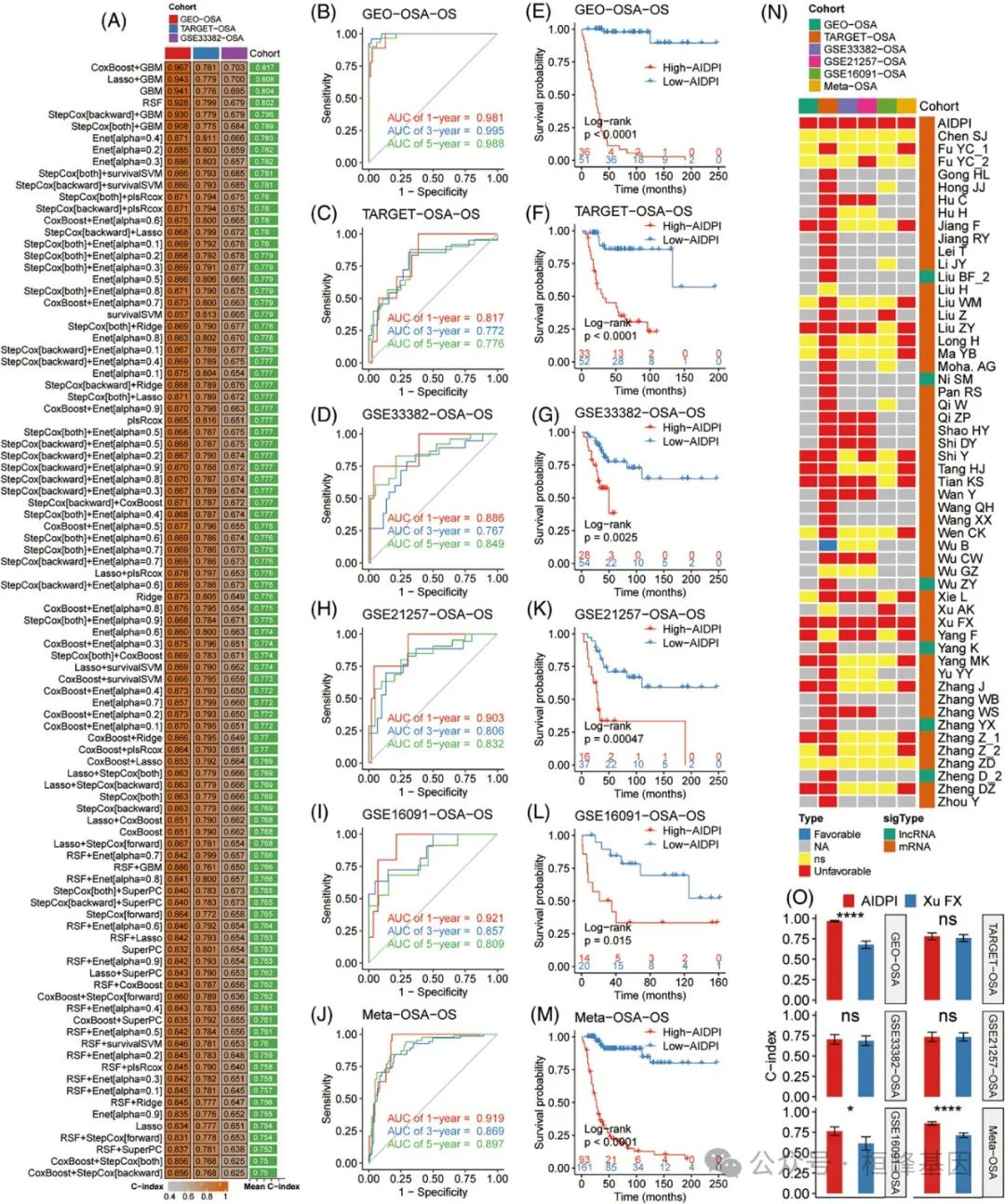
2. 基于人工智能预后指数(AIDPI)和临床特征的生存预测
(A) AIDPI与临床病理特征预后价值关系的单因素Cox回归分析,
(B-D)显示AIDPI在不同Huvos分级中的分布的箱形图,
(C-E)评估AIDPI预测新辅助化疗反应能力的ROC曲线,
(F-G)有或没有Huvos分级整合的多因素Cox回归分析,
(H)来自meta-骨肉瘤(OSA)的nomogram,
(I)模态图的标定曲线,
(J)建立的模态图的时间相关ROC曲线分析,
(K)各因素之间的预测性能比较,
(L)决策曲线分析强调了模态图相对于其他指标的优越净效益。
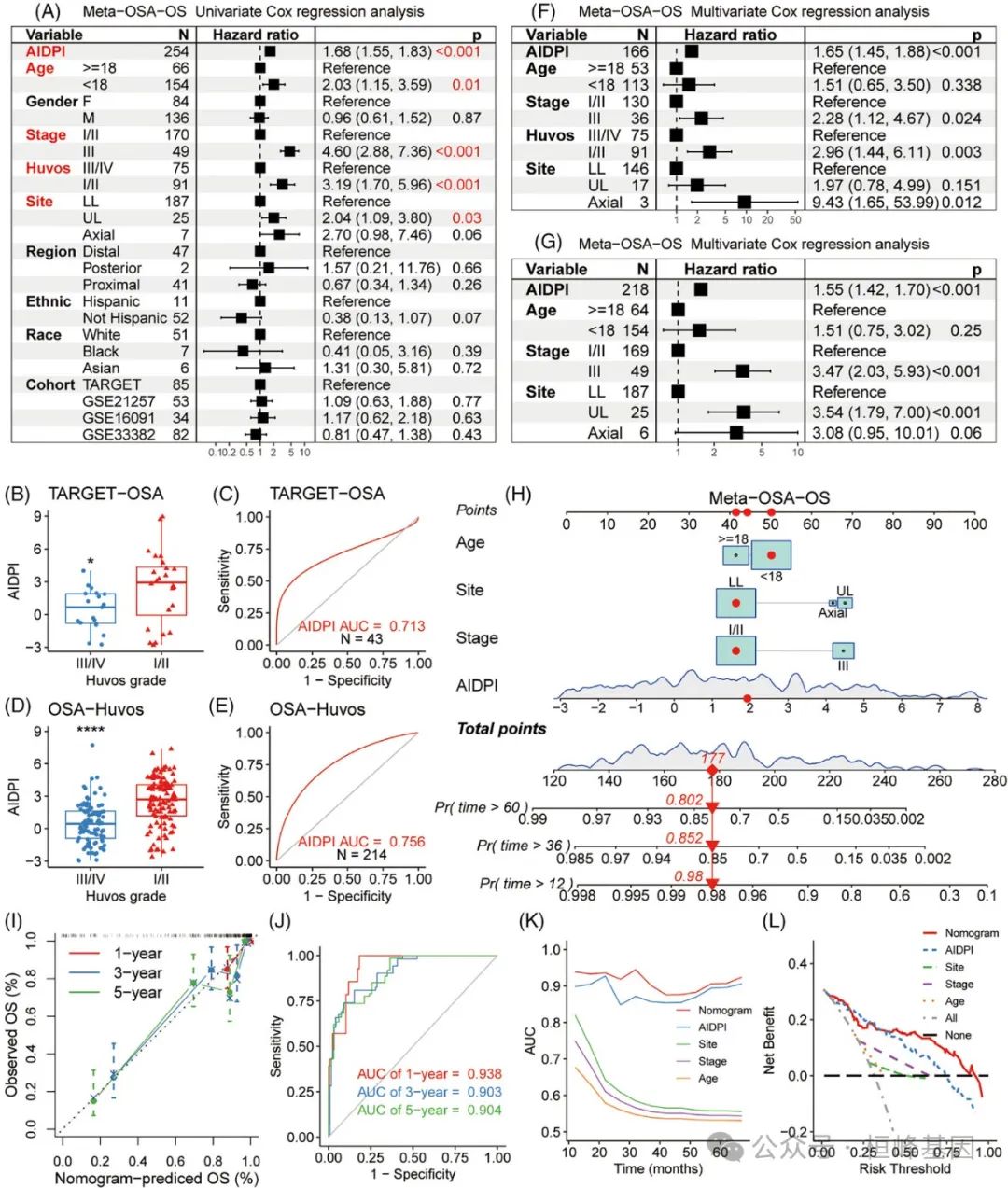
3. 识别高人工智能预后指数(AIDPI)患者中的失调通路
(A)热图阐明了AIDPI基因的表达及其与免疫评分的相关性。
(B)基因集富集分析(GSEA)结果聚焦与高(红色)或低(蓝色)AIDPI显著相关的生物过程。
(C)气泡图显示了基于差异表达基因(DEGs)的KEGG富集分析结果。
(D)富集图将富集的term排列成一个网络,将具有共享基因的term连接起来。
(E)箱线图对比了两个AIDPI组的平均DNA甲基化水平。
(F)柱状图显示了基于表观基因组数据的经验BayesGSEA结果。
4. 确定高人工智能预后指数(AIDPI)患者的治疗靶点
(A)热图显示了预测骨肉瘤(OSA)细胞中推断的CNA,
(B)九种注释细胞类型的均匀聚类图和UMAP图,
(C)气泡图显示了九个已鉴定细胞簇中的标记基因表达,
(D)在曼哈顿图中,特定细胞簇中高表达的基因被精确定位,
(E)环状图显示了两个AIDPI组之间差异表达基因(DEGs)的细胞来源。
(F)维恩图表示指示基因组之间的共享实体,
(G)特征图显示单个细胞中指示基因的规范化表达。
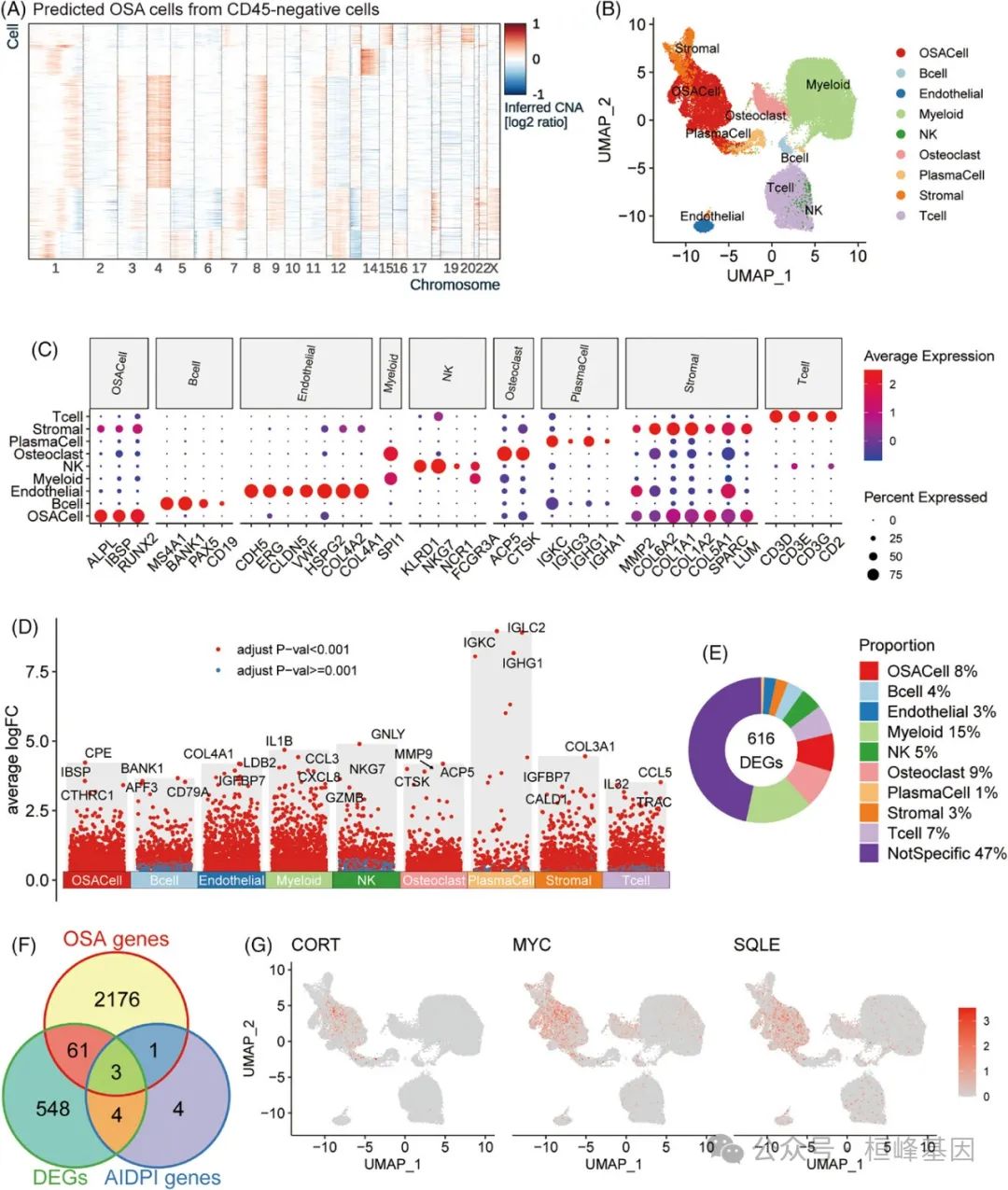
5. 骨肉瘤(OSA)中角鲨烯环氧化酶(SQLE)的过表达与肿瘤进展相
(A-C)箱形图显示SQLE mRNA水平,对比OSA组织与邻近正常组织(A),OSA细胞系(OSACell)与成骨细胞(OB)和间充质干细胞(MSC)。
(B)不同Huvos分级的样品和甲氨蝶呤耐药Saos2 (Saos2/MTX)与其亲本细胞系(C)。
(D-E)散点图显示OSA样品和OSA细胞系中SQLE拷贝数与mRNA水平之间的关系。
(F-G)通过OSA样本和OSA细胞系的散点图描绘了MYC和SQLE基因水平拷贝数之间的相关性。
(H)散点图显示了数据集中MYC和SQLE mRNA表达的相关性。
(I-P) Kaplan-Meier生存分析SQLE mRNA水平在多个人类和OSA队列中进行。
(Q)TMA中SQLE免疫组化(IHC)染色的代表性图像。
(S)森林图为多变量Cox回归分析结果。
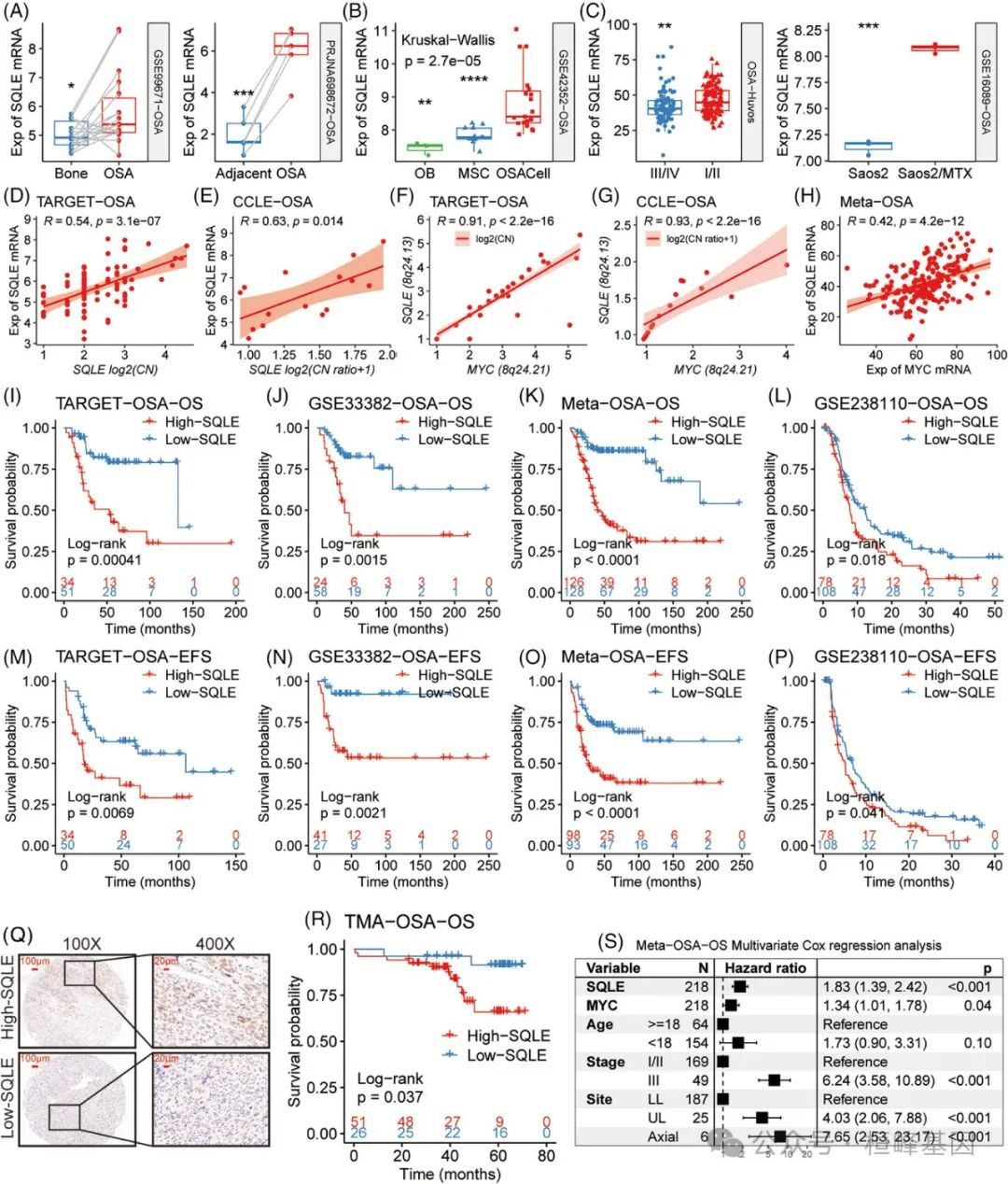
6. 角鲨烯环氧化酶(SQLE)敲低抑制骨肉瘤(OSA)在体内和体外的进展
(A)转染shSQLE或shControl后的SQLE蛋白丰度通过Western blot在标记的细胞组中显示。
(B-E)标记细胞组细胞增殖(B)、克隆生长(C)、迁移(D)和侵袭(E)的测定。
(F-G)流式细胞术分析,采用AnnexinV-APC/7-AAD染色,定量标记细胞组中的凋亡细胞,无论是未经处理(F)还是阿霉素(ADM)暴露(G)。
(H-J)条形图对比了指定细胞组对指定药物的敏感性。
(K)使用含有shControl或shSQLE的U2OS细胞建立的皮下异种移植物模型的分离肿瘤图像。
(L)指示组肿瘤生长曲线。
(M)肿瘤肿块的体重和胆固醇水平以条形图表示。
(N)异种移植模型肿瘤中SQLE和Ki-67染色的免疫组化代表性图像。
(O)条形图对比了Ki-67阳性细胞的SQLE丰度和百分比。
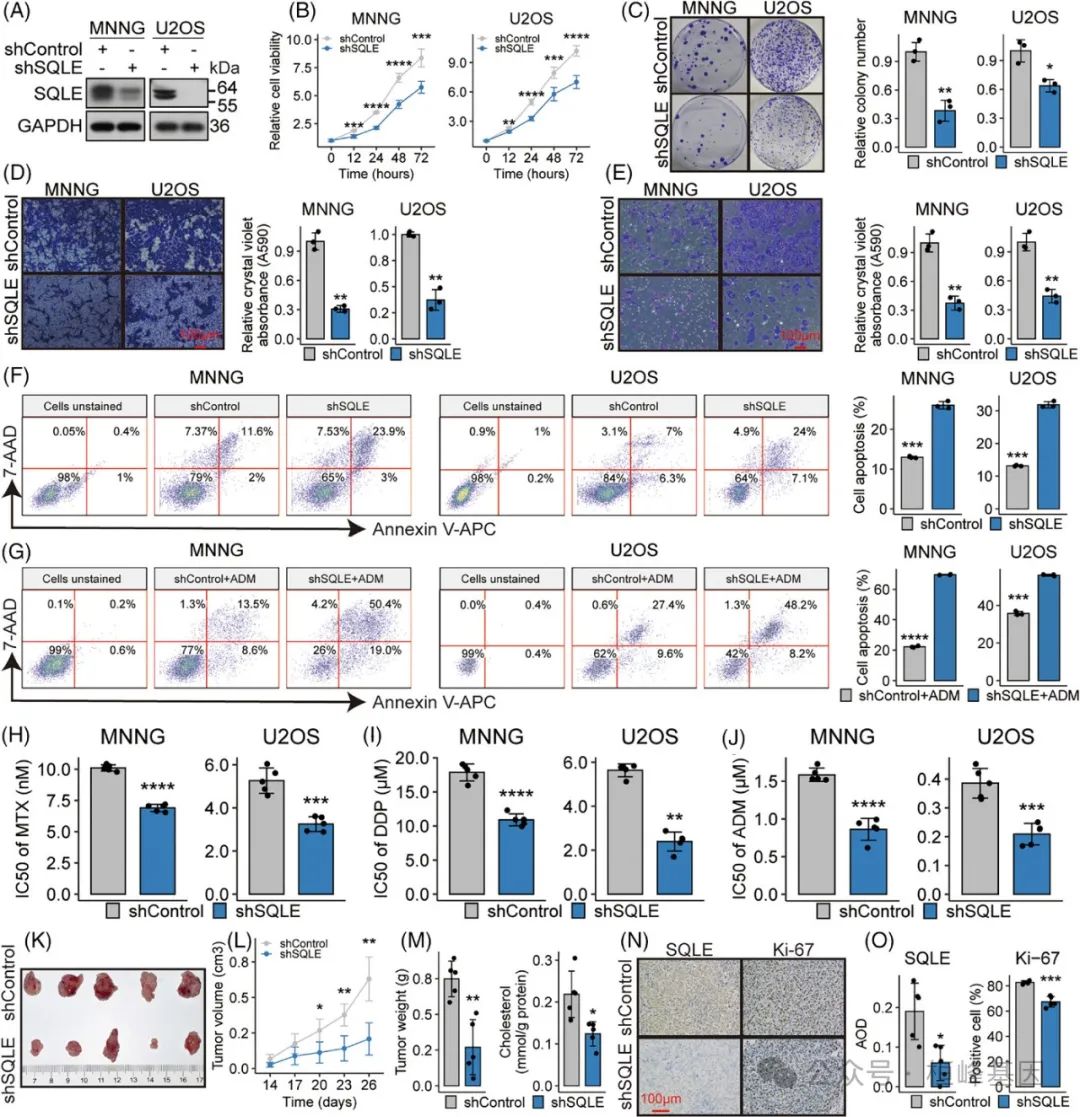
7. 角鲨烯环氧化酶(SQLE)沉默通过降低胆固醇和抑制FAK/PI3K/Akt/mTOR通路来抑制骨肉瘤(OSA)
(A-D)基因集富集分析(GSEA)图显示了指定基因集的富集趋势。
(B)柱状图显示标记细胞组的细胞内胆固醇水平。
(C)差异表达基因(DEGs)的KEGG富集结果以气泡图表示。
(E)散点图显示了针对PI3K/mTOR信号级联的三种特异性抑制剂的SQLE mRNA水平与剂量-反应曲线(AAC)值上方面积之间的相关性。
(F-G)显示标记细胞组中的蛋白质表达谱和随后的定量。
(H-K)显示指定细胞组的实验结果,包括细胞增殖(H)、克隆潜能(I)、迁移(J)和侵袭能力(K)。
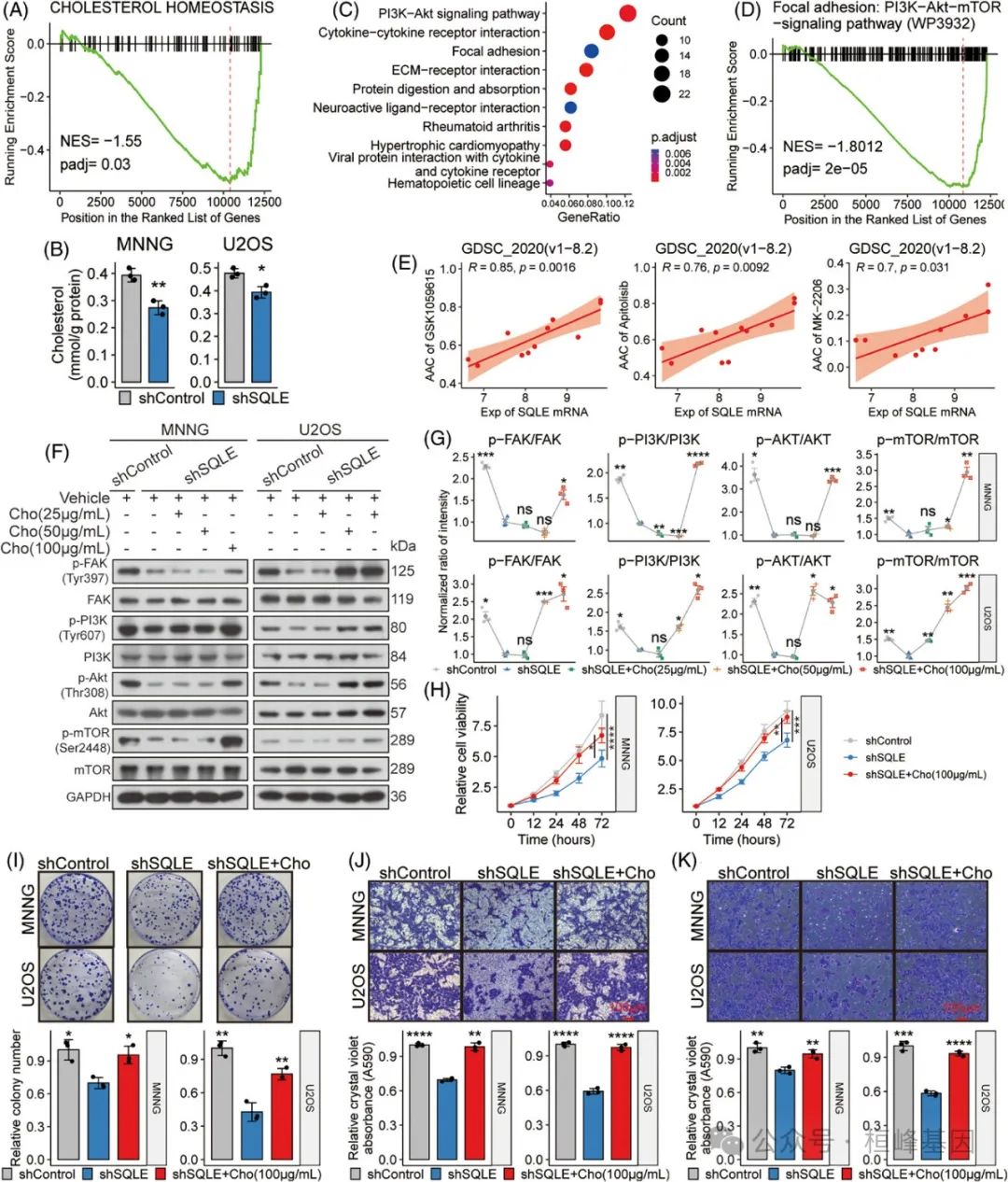
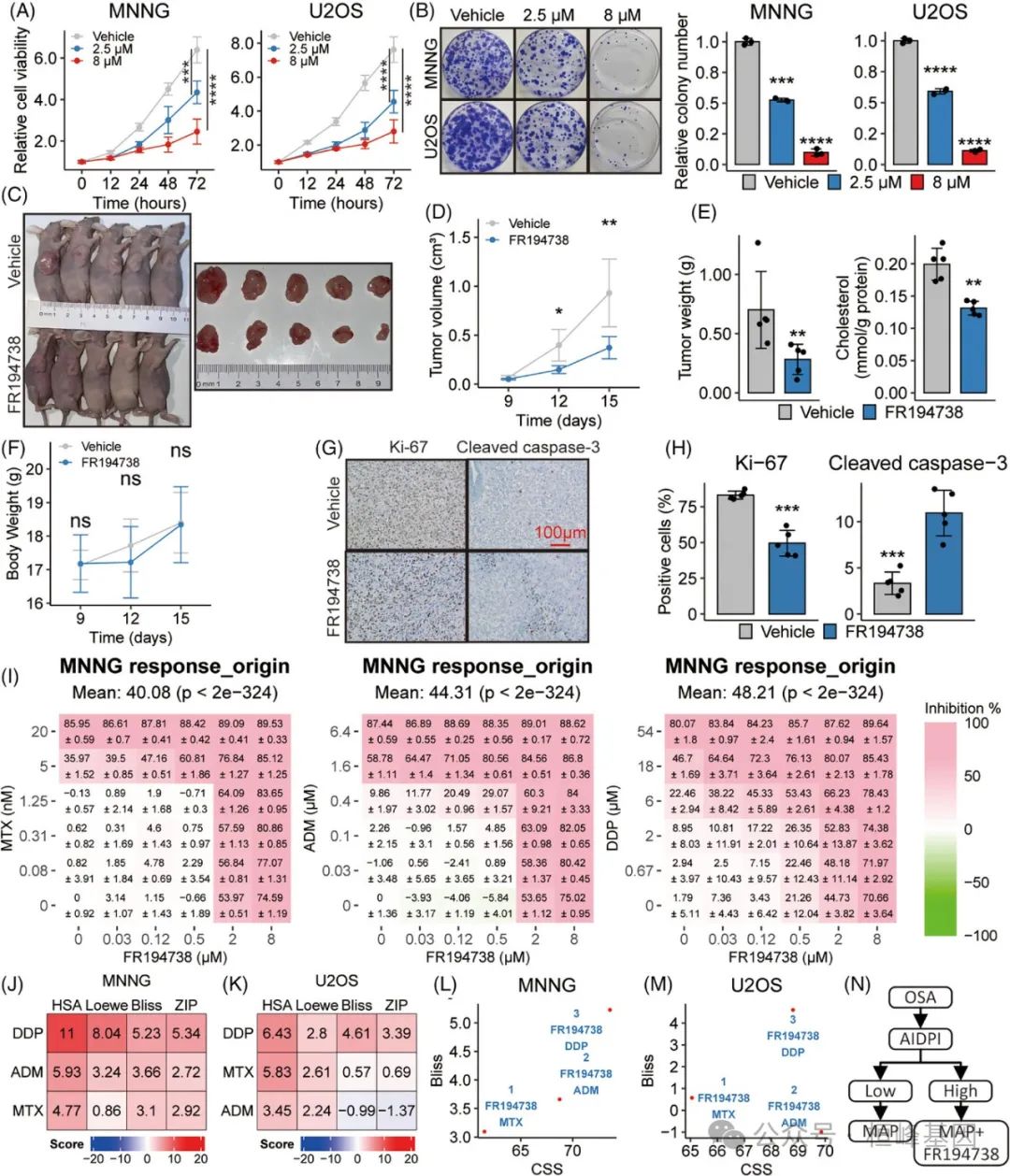
8. 药理角鲨烯环氧化酶(SQLE)抑制骨肉瘤(OSA)进展并提高化疗疗效
(A-B)实验表明,与对照剂相比,两种浓度的FR194738对细胞增殖(A)和集落形成能力(B)的影响。
(C)对照剂或FR194738处理的皮下异种移植模型分离肿瘤图像。
(D)指示组肿瘤生长曲线。
(E)肿瘤肿块的体重和胆固醇水平在柱状图中表示。
(F)指示组体重曲线。
(G)肿瘤切片中Ki-67和裂解caspase-3染色的免疫组化代表性图像。
(H)条形图对比Ki-67和裂解caspase-3阳性细胞的百分比。
(I)热图提供了MNNG对一系列药物组合的剂量反应可视化。
(J-K)热图显示了联合使用FR194738和指定药物对抗指定OSA细胞系时的协同评分。
(L-M)散点图显示了OSA细胞系的敏感性指标和协同指标。
(N)示意图展示了人工智能衍生预后指数(AIDPI)在临床应用的可能途径。
Reference
Wang Y, Ma X, Xu E, Huang Z, Yang C, Zhu K, Dong Y, Zhang C. Identifying squalene epoxidase as a metabolic vulnerability in high-risk osteosarcoma using an artificial intelligence-derived prognostic index. Clin Transl Med. 2024 Feb;14(2):e1586.
基于机器学习构建临床预测模型
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
MachineLearning 14. 机器学习之集成分类器(AdaBoost)
MachineLearning 15. 机器学习之集成分类器(LogitBoost)
MachineLearning 16. 机器学习之梯度提升机(GBM)
MachineLearning 17. 机器学习之围绕中心点划分算法(PAM)
MachineLearning 18. 机器学习之贝叶斯分析类器(Naive Bayes)
MachineLearning 19. 机器学习之神经网络分类器(NNET)
MachineLearning 20. 机器学习之袋装分类回归树(Bagged CART)
MachineLearning 21. 机器学习之临床医学上的生存分析(xgboost)
MachineLearning 22. 机器学习之有监督主成分分析筛选基因(SuperPC)
MachineLearning 23. 机器学习之岭回归预测基因型和表型(Ridge)
MachineLearning 24. 机器学习之似然增强Cox比例风险模型筛选变量及预后估计(CoxBoost)
MachineLearning 25. 机器学习之支持向量机应用于生存分析(survivalsvm)
MachineLearning 26. 机器学习之弹性网络算法应用于生存分析(Enet)
MachineLearning 27. 机器学习之逐步Cox回归筛选变量(StepCox)
MachineLearning 28. 机器学习之偏最小二乘回归应用于生存分析(plsRcox)
MachineLearning 29. 机器学习之嵌套交叉验证(Nested CV)
MachineLearning 30. 机器学习之特征选择森林之神(Boruta)
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








