欢迎关注”生信修炼手册”!
MetaGenyo是一个GWAS meta分析的在线网站,通过该网站,只需上传指定格式的文件,然后鼠标点击就可以轻松实现meta分析,网址如下
http://bioinfo.genyo.es/metagenyo/
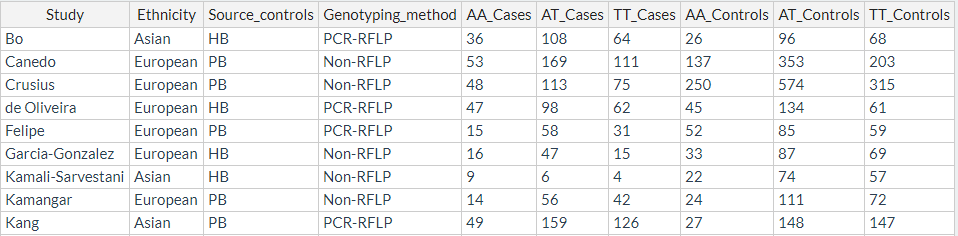
对应的数据格式图示如下

每个文件表示的是一个SNP位点在多个study中的研究结果,每一行为一个study。其中有6列是必须的,即case和control两组中不同基因型的频数,上图中SNP位点有3种基因型AA, AT, TT,表头的格式是固定,基因型+cases/controls, 其他列是自定义的。
用法简便,包含以下几步
1. 上传数据
通过Data input菜单上传数据,示例数据如下
上传完成后,可以在Your data菜单查看文件内容,确认没问题之后,点击Next就可以了。
2. Hardy-Weinberg table
对于上传的数据,会进行哈温平衡的检验,结果如下所示

3. Association values
对于原始的频数分布,先针对每个study的结果进行关联分析,然后对多个study的结果进行meta分析,在进行关联分析时,会有以下模型
Allele contrast (A vs. T)
Recessive model (AA vs. AT+TT)
Dominant model (AA+AT vs. TT)
Overdominant model (AT vs. AA+TT)
AA vs. TT
AA vs. AT
AT vs. TT
每种模型的结果都会给出,以第一种模型为例,结果如下所示

4. Forest plots
对于每个模型的meta分析结果,提供了如下所示的森林图

利用该网站,可以方便的对多个case/control关联分析的结果进行meta分析,当然该网站也有一定的限制,很多时候我们收集到的结果已经是pvalue值了,并不会有原始的频数分布,这个时候就无法使用该网站进行分析。
·end·
—如果喜欢,快分享给你的朋友们吧—
往期精彩
基因型填充
CNV分析








 MetaGenyo是一个在线平台,提供简单的GWAS meta分析服务。用户只需上传特定格式的数据,经过几步操作,如哈温平衡检验、关联分析和森林图展示,即可完成分析。虽然该网站适用于有原始频数分布的数据,但不支持只有pvalue值的情况。
MetaGenyo是一个在线平台,提供简单的GWAS meta分析服务。用户只需上传特定格式的数据,经过几步操作,如哈温平衡检验、关联分析和森林图展示,即可完成分析。虽然该网站适用于有原始频数分布的数据,但不支持只有pvalue值的情况。
 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 3180
3180

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








