
Model weights: https://huggingface.co/InstructPLM/MPNN-ProGen2-xlarge-CATH42
Code: https://github.com/Eikor/InstructPLM
Abstract
大语言模型在捕获共同进化信息和潜在的蛋白质语言方面的有效性。然而,目前方法不能说明基因组差补、重复和插入/缺失(indels)的出现,这些现象约占人类致病突变的14%。鉴于蛋白质结构决定其功能,具有相似结构的突变蛋白更有可能在整个生物进化过程中持续存在。受此启发,利用跨模态对齐并指导受大型语言模型启发的微调技术,以将生成蛋白质语言模型与蛋白质结构指令对齐。
Introduction
突变,包括单核苷酸变化引起的点突变,以及多个碱基的缺失、重复和插入,是进化的驱动力,促进生命快速适应不断变化的环境条件,这是遗传多样性的根本原因 。 这些遗传变异为蛋白质工程中的参考和选择提供了丰富的蛋白质库。蛋白质工程的一大挑战是蛋白质序列设计(蛋白质逆折叠),识别能够折叠成特定蛋白质主链结构的氨基酸序列。高度准确的蛋白质序列设计能够生成更有效的酶、改进的基于蛋白质的疗法、用于生物燃料生产和环境修复等工业目的的工程蛋白。最近基于深度学习的方法极大的推动了蛋白质序列设计领域的发展(ProteinMPNN和ESM-IF)通过编码器-解码器的训练将蛋白质主链结构转化为相应的序列。
深度学习的快速发展和蛋白质数据库(PDB)和AlphaFoldDB的高质量蛋白质结构数据的可用性,促使该领域的进步。例如,ProteinMPNN 设计的肌红蛋白和烟草蚀纹病毒 (TEV) 蛋白酶已表现出优异的表达、稳定性和功能性; ProteinMPNN 设计的从头结合剂对靶点表现出高亲和力和特异性。
多模态学习虽然有效,但是缺乏高质量的跨模态数据而面临重大障碍。在蛋白质设计中,ProteinMPNN的训练,依赖于蛋白质数据库(PDB)。为了解决数据有限的问题,大型视觉语言模型领域的一个新兴策略是跨模态对齐。预训练的单模态模型通过进行广泛的训练来学习特定领域的表示,但它们通常缺乏跨模态的理解和推理能力。
跨模态对齐通过仔细地将预训练的视觉模型与预训练的语言模型对齐来弥补这一差距,从而实现有效的跨模态生成、理解和推理。 尽管跨模态比对技术在视觉语言理解方面取得了成功,但由于蛋白质科学和视觉语言任务之间固有的差异,它们在蛋白质序列设计中的潜力仍未得到探索。 因此,解决这一挑战需要开发专门针对蛋白质的强大的单模态模型和比对技术。
幸运的是,蛋白质语言模型(pLM),例如 ESM、ProtGPT 和 ProGen,已经成为生物信息学和计算生物学领域的关键创新。在这项工作中,我们展示了最初为大型语言模型开发的跨模态比对和指令微调技术成功适应蛋白质序列设计领域。 我们提出的模型 InstructPLM 采用轻量级交叉注意力层将蛋白质主链编码器与蛋白质语言模型解码器对齐,旨在教导蛋白质语言模型按照蛋白质结构指令设计序列。
InstructPLM的整体架构包括——蛋白质语言模型解码器、蛋白质主干编码器、蛋白质序列适配器。
蛋白质语言模型解码器:InstructPLM采用ProGen2作为其蛋白质语言模型。 ProGen2 模型系列包括对来自 Uniref90 和 BFD30 数据库混合的约 2.3 亿个蛋白质序列进行预训练的自回归语言模型,其模型大小范围从 151M 到 6.4B。 文中选择最大的一个,ProGen2-xlarge,在 InstructPLM 中具有 6.4B 参数。
蛋白质主干编码器:InstructPLM从其他现有的蛋白质序列设计模型初始化其蛋白质主干编码器,例如ProteinMPNN和ESM-IF的主干编码器。
蛋白质结构-序列适配器:蛋白质结构-序列适配器不仅负责将结构和序列对齐到同一语义空间,还包含InstructPLM的所有可训练参数。 该适配器包含随机初始化的单层交叉注意模块。该模块使用多个可训练的嵌入作为查询向量,使用蛋白质主干编码器的输出作为交叉注意力计算的键/值。该交叉注意力模块将蛋白质主干嵌入压缩为固定长度的结构指令。 查询向量(即结构指令的长度)为512。此外,我们遵循 Qwen-VL将一维绝对位置编码添加到交叉注意力中,以在压缩过程中保留蛋白质一级结构信息。 压缩的蛋白质主干结构特征序列随后作为软提示输入蛋白质语言模型。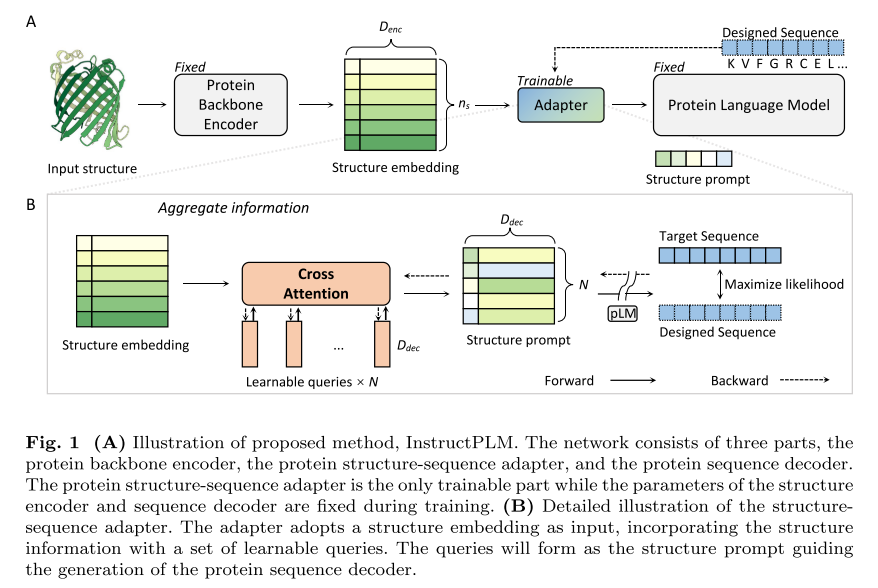
Method
Model Architecture
给定一个蛋白质骨架,目标是识别合适的蛋白质序列
S
S
S,其中
S
=
(
s
1
,
s
2
,
.
.
.
.
,
)
S=(s_1,s_2,....,)
S=(s1,s2,....,)折叠成给定结构。
Protein Backbone Encoder
首先使用一个预训练的结构编码器获得结构嵌入
E
∈
R
n
×
D
e
n
c
E \in R^{n \times D_{enc}}
E∈Rn×Denc,
n
n
n是蛋白质残基的说量。
D
e
n
c
D_{enc}
Denc是结构编码器的嵌入维度。
Protein Structure-Sequence Adapter
包括四个组件:用于将结构嵌入与潜在的相互作用空间对齐的线性投影层、用于捕获蛋白质残基水平的连接的动态位置编码层、具有一组可学习查询 L = R N × D d e c L=R^{N \times D_{dec}} L=RN×Ddec的交叉注意层,用于知识迁移,其中其中 N N N 是可调超参数, D d e c D_{dec} Ddec 由预训练序列解码器的隐藏维度确定。最后,部署一个映射到语言嵌入的输出投影层。 为了确保模块输出的稳定性,我们进一步合并了三个归一化层。
对于输入的结构嵌入
E
E
E,适配器会将
E
E
E填充0到固定的长度
n
s
=
512
n_s=512
ns=512。首先将位置信息合并到结构嵌入
E
E
E和可学习的查询(Q)
L
L
L。对于最大长度为
n
s
n_s
ns的蛋白质序列,位置编码ProtPE为编码的每个维度
i
=
0
,
.
.
.
,
D
d
e
c
/
2
−
1
i=0,...,D_{dec}/2-1
i=0,...,Ddec/2−1被定义在位置
p
o
s
=
0
,
.
.
.
,
n
s
−
1
pos=0,...,n_s-1
pos=0,...,ns−1为:
ProtPE
[
p
o
s
,
2
i
]
=
sin
(
p
o
s
1000
0
2
i
D
)
,
ProtPE
[
p
o
s
,
2
i
+
1
]
=
cos
(
p
o
s
1000
0
2
i
D
)
.
\begin{aligned} \text { ProtPE }[p o s, 2 i] & =\sin \left(\frac{p o s}{10000^{\frac{2 i}{D}}}\right), \\ \operatorname{ProtPE}[p o s, 2 i+1] & =\cos \left(\frac{p o s}{10000^{\frac{2 i}{D}}}\right) . \end{aligned}
ProtPE [pos,2i]ProtPE[pos,2i+1]=sin(10000D2ipos),=cos(10000D2ipos).
在与位置嵌入耦合之前,结构嵌入
E
E
E 被投影到潜在交互空间:
E
′
=
L
a
t
e
r
N
o
r
m
(
E
W
)
+
P
r
o
t
P
E
E^{'}=LaterNorm(EW)+ProtPE
E′=LaterNorm(EW)+ProtPE
其中
W
∈
R
D
e
n
c
×
D
d
e
c
W \in R^{D_{enc} \times D_{dec}}
W∈RDenc×Ddec,由于可学习的Q的长度与输入结构长度
n
s
n_s
ns不一致,因此通过添加具有均匀分布步长的位置特征嵌入来将位置信息注入到查询中:
L
′
=
L
a
t
e
r
N
o
r
m
(
L
)
+
P
r
o
t
P
E
[
:
:
s
t
r
i
d
e
]
L^{'}=LaterNorm(L)+ProtPE[::stride]
L′=LaterNorm(L)+ProtPE[::stride]
s
t
r
i
d
e
=
n
s
/
/
N
stride=n_s//N
stride=ns//N
交叉注意力层被表述为将Q、K和V映射到输出的函数。 我们根据结构嵌入定义键和值,并使用定义的可学习查询来查询它们。为了增强模型关注来自不同子空间的信息的能力,我们采用了多头注意力策略,其中注意力功能在不同的注意力头中并行执行。 对于每个注意力头 k ∈ {1, 2, · · · , A},结构嵌入和查询通过线性投影投影到相同的维度:
Q
k
=
L
′
W
Q
k
,
K
k
=
E
′
W
K
k
,
V
k
=
E
′
W
V
k
,
\boldsymbol{Q}^{k}=\boldsymbol{L}^{\prime} \boldsymbol{W}_{Q}^{k}, \quad \boldsymbol{K}^{k}=\boldsymbol{E}^{\prime} \boldsymbol{W}_{K}^{k}, \quad \boldsymbol{V}^{k}=\boldsymbol{E}^{\prime} \boldsymbol{W}_{V}^{k},
Qk=L′WQk,Kk=E′WKk,Vk=E′WVk,
其中
W
Q
k
,
W
K
k
,
W
V
k
∈
R
D
d
e
c
×
D
k
\boldsymbol{W}_{Q}^{k},\boldsymbol{W}_{K}^{k},\boldsymbol{W}_{V}^{k} \in R^{D_{dec} \times D_k}
WQk,WKk,WVk∈RDdec×Dk,
D
k
=
D
d
e
c
/
A
D_k=D_{dec}/A
Dk=Ddec/A代表注意力头的维度。
计算
H
k
H^k
Hk
H
k
=
softmax
(
Q
k
K
k
⊤
D
k
)
V
k
\boldsymbol{H}^k=\operatorname{softmax}\left(\frac{\boldsymbol{Q}^k \boldsymbol{K}^{k \top}}{\sqrt{D_k}}\right) \boldsymbol{V}^k
Hk=softmax(DkQkKk⊤)Vk






















 1186
1186











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








