写在前面
【这图怎么画】系列的图都来自VIP群里同学的提问。推文只是对图片的复现,不代表作者对图片展现形式的认同。欢迎同学们在群里分享有意思的图片。
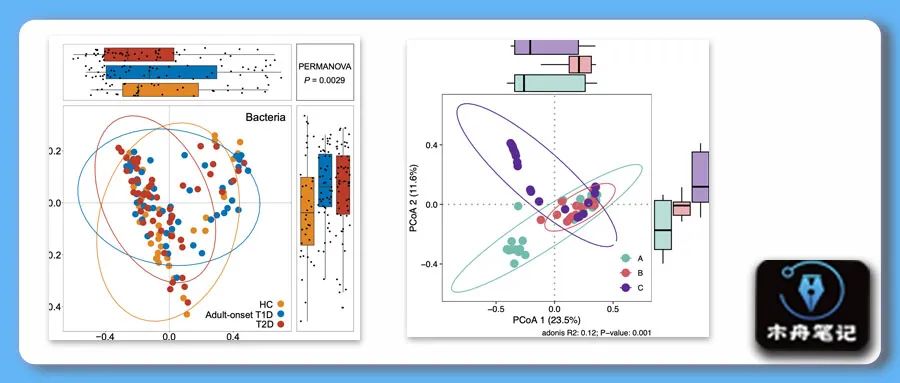
本期图片

❝Doi:https://doi.org/10.1016%2Fj.eclinm.2023.102132
❞
既往相关推文:R实战 | 置换多元方差分析(以PCoA的PERMANOVA分析为例)
复现结果

❝可在AI上微调。使用代码时请按需修改,切勿全部套用。
❞
示例数据和代码领取
木舟笔记永久VIP企划
「权益:」
「木舟笔记所有推文示例数据及代码(「在VIP群里」实时更新」)。

data+code 木舟笔记「科研交流群」。
「收费:」
「169¥/人」。可添加微信:mzbj0002 转账(或扫描下方二维码),或直接在文末打赏。木舟笔记「2022VIP」可直接支付「70¥」升级。
❝❞
点赞、在看本文,分享至朋友圈集赞30个并保留30分钟,可优惠20¥。

绘图
#install.packages('ggExtra')
library(ggplot2)
library(ggExtra)
#### PCoA ####
#install.packages('vegan')
# Load package
library(vegan)
library(ggthemes)
# Load example data
otu <- read.table('otu.txt',row.names = 1,header = T)
group <- read.table('group.txt',header = T)
#pcoa
# vegdist函数,计算距离;method参数,选择距离类型
distance <- vegdist(otu, method = 'bray')
# 对加权距离进行PCoA分析
pcoa <- cmdscale(distance, k = (nrow(otu) - 1), eig = TRUE)
## plot data
# 提取样本点坐标
plot_data <- data.frame({pcoa$point})[1:2]
# 提取列名,便于后面操作。
plot_data$ID <- rownames(plot_data)
names(plot_data)[1:2] <- c('PCoA1', 'PCoA2')
# eig记录了PCoA排序结果中,主要排序轴的特征值(再除以特征值总和就是各轴的解释量)
eig = pcoa$eig
#为样本点坐标添加分组信息
plot_data <- merge(plot_data, group, by = 'ID', all.x = TRUE)
head(plot_data)
# 计算加权bray-curtis距离
dune_dist <- vegdist(otu, method="bray", binary=F)
dune_pcoa <- cmdscale(dune_dist, k=(nrow(otu) - 1), eig=T)
dune_pcoa_points <- as.data.frame(dune_pcoa$points)
sum_eig <- sum(dune_pcoa$eig)
eig_percent <- round(dune_pcoa$eig/sum_eig*100,1)
colnames(dune_pcoa_points) <- paste0("PCoA", 1:3)
dune_pcoa_result <- cbind(dune_pcoa_points, group)
head(dune_pcoa_result)
#### 计算p值 ####
# 基于bray-curtis距离进行
dune.div <- adonis2(otu ~ group, data = group, permutations = 999, method="bray")
dune.div
#install.packages('ggalt')
library(ggalt)
dune_adonis <- paste0("adonis R2: ",round(dune.div$R2,2), "; P-value: ", dune.div$`Pr(>F)`)
dune_adonis
#### 绘制pcoa图 ####
p = ggplot(dune_pcoa_result, aes(x=PCoA1, y=PCoA2, color=group)) +
geom_point(size=4) +
stat_ellipse(aes(color = group),level=0.95)+
scale_color_manual(values = c('#73bbaf','#d15b64','#592c93'))+
labs(x=paste("PCoA 1 (", eig_percent[1], "%)", sep=""),
y=paste("PCoA 2 (", eig_percent[2], "%)", sep=""),
caption = dune_adonis,) +
theme_bw()+
geom_hline(yintercept = 0, linetype = 'dotted', color = "grey60",linewidth=0.8)+
geom_vline(xintercept = 0, linetype = 'dotted', color = "grey60",linewidth=0.8)+
theme(legend.position = c(0.9,0.15),
legend.title = element_blank(),
panel.grid = element_blank(),
axis.text = element_text(color = "black",size=10))
p
#### 绘制边缘分布图 ####
pdf('marginal.pdf',width = 5,height = 5)
ggMarginal(
p,
type =c('boxplot'),
margins = 'both',
size = 3.5,
groupColour = F,
groupFill = T
)
dev.off()往期内容
























 199
199

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








