大家都知道,孟德尔随机化很大程度依赖于国外的服务器。
最近我们发现孟德尔随机化常用的TwoSampleMR包的clump函数经常报错,这是由于服务器访问人群超时造成的现象,当线上版本失效。
很多人做孟德尔随机化,就卡在clump上。
于是我们就找了本地方法来进行连锁不平衡分析。
今天教一下大家如何操作:
第一步:下载两个东西
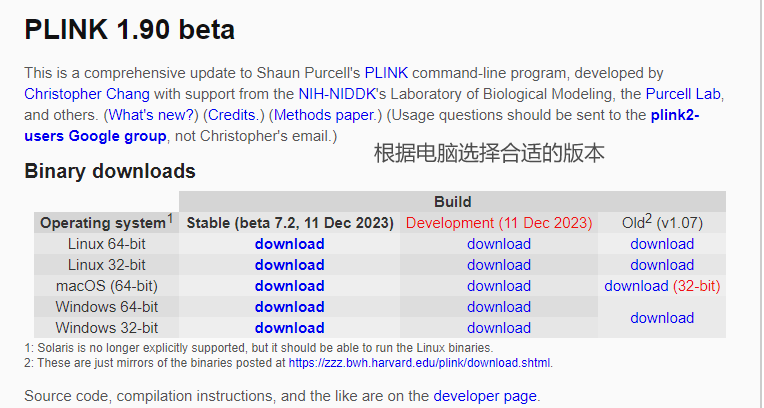
pink link软件和data_maf0.01_rs_ref参考文件下载
pink link软件:https://www.cog-genomics.org/plink/1.9/
data_maf0.01_rs_ref参考文件:https://github.com/MRCIEU/gwasvcf
1)下载pink link软件
2)下载data_maf0.01_rs_ref参考文件

下载后将文件解压后放在工作空间即可

第二步:在R中安装相应的包

第三步:读取数据后将数据框中的ID列和P值列进行保留

将列名重新命名成rsid和pval,否则接下来的操作会报错
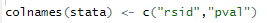
第四步:通过下面函数进行连锁不平衡操作

注意:bfile上写的是之前下载的data_maf0.01_rs_ref的工作路径,设置完后再加一个data_maf0.01_rs_ref(代表文件夹里的三个文件)。同理,plink_bin上写的之前下载的plink工作路径,再在后面加上plink
最后,通过以下函数保存文件

最后得到的文件就可以进行后续的孟德尔随机化分析啦~
一个专门做公共数据库的公众号,关注我们
























 1642
1642

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








