原创 普美瑞生物科技 普美瑞生物 2024年11月26日 15:27 江苏
前言
在药物研发领域,化学空间的广阔性意味着无数潜在化合物尚待探索和发现。然而,这些化合物中只有极少数具备治疗疾病的潜力。面对如此庞大的化合物库,传统的实验筛选方法显得力不从心,不仅耗时耗力,而且效率低下。如何快速、高效地筛选出具有潜在应用价值的药物分子,始终是该领域的一大挑战。虚拟筛选技术的出现,标志着药物研发领域的一次革命性进步。该技术通过先进的计算机模拟和算法,能够对庞大的化合物库进行高效筛选,从中识别出具有潜在药物活性的分子。这不仅显著缩小了实验筛选的范围,缩短了新药发现的周期,还降低了研发成本,并提升了成功率。因此,虚拟筛选技术是应对化学空间复杂性挑战的有效手段,对于推动药物研发进程具有深远意义。
01 虚拟筛选原理
虚拟筛选是指在进行生物活性实验筛选之前,根据预先设定的条件,在计算机上对化合物库中的分子进行预筛选,以识别出最有可能与靶标结合的小分子。这一过程大大减少了实际筛选的化合物数目,同时提高先导化合物的发现效率。从原理上来讲,虚拟筛选可以分为两类,即基于配体的虚拟筛选(包括结构相似度、QSAR、药效团模型)和基于受体/结构的虚拟筛选(如分子对接、分子动力学模拟)。
基于配体的虚拟筛选依赖于已知配体的化学和结构特性,通常认为具有相似结构的化合物往往具有类似的药理活性。在靶标未知或蛋白结构尚未解析的情况下,基于配体的虚拟筛选是一种有效的药物发现策略。该方法利用已知的活性配体作为模板或探针,从庞大的化合物库中筛选出与其具有相似结合特征或作用机制的潜在药物候选分子。近年来,随着AlphaFold的诞生与发展,越来越多的蛋白质三维结构被解析,这极大地推动了基于结构的虚拟筛选技术的进步。基于结构的虚拟筛选通过靶蛋白的三维结构来预测潜在的药物分子,主要通过分子对接技术将候选分子与靶蛋白的活性位点进行对接,评估分子间的相互作用能、结合模式及可能引发的构象变化,从而筛选出与靶蛋白具有高亲和力和特异性的候选药物分子。本文将重点讨论基于分子对接的虚拟筛选方法。
分子对接方法已广泛应用于药物研发领域,成为针对大规模药物分子进行虚拟筛选的重要手段。该方法通过计算模拟技术深入研究分子间的结合模式和亲和力,为药物研发提供了重要的理论支持。其理论基础源于1894年由Fisher提出的“锁-钥模型”概念,该理论将药物与受体之间的结合过程比作锁与钥匙的精确匹配,强调了药物分子与受体蛋白之间在形状和结构上的互补性[1]。随后,在1958年,Koshland进一步提出了诱导契合学说,该学说认为药物分子与受体蛋白在结合过程中会发生相互适应和诱导调整,使得药物分子更好地适应受体蛋白的活性口袋,从而达到更稳定、更高效的结合[2]。这两种理论为分子对接技术的发展提供了重要的理论基础,因此也催生了不同的分子对接方法。分子对接常用的方法主要包括刚性对接、半柔性对接和全柔性对接,这三种对接方法因对精度的要求不同,所需的计算量和耗时也呈现出明显的差异。在大规模虚拟筛选中,半柔性对接通常被认为是一种较优的选择。
02 基于分子对接的虚拟筛选一般流程
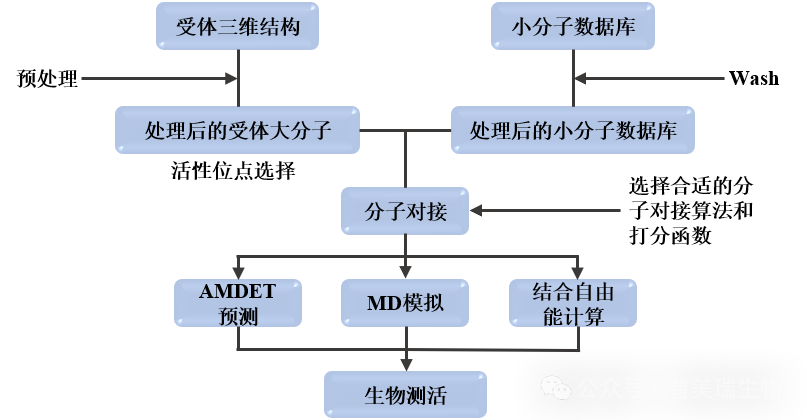
图1. 基于分子对接的虚拟筛选流程图
基于分子对接的虚拟筛选过程(图1)包括:
1)受体三维结构准备:对于已知晶体结构的蛋白靶点,可直接从PDB数据库中获取;对于未解析出晶体结构的受体,可采用同源建模技术构建其三维结构。获取受体结构后,需要进行一系列预处理操作,如去除水分子、添加氢原子和电荷、补全残基等;
2)小分子数据库准备:主流的小分子数据库包括ZINC、PubChem、ChEMBL、DrugBank、ChemDB、BindingDB等。下载的小分子数据库需要进行Wash操作,包括加氢、去盐、去除冗余分子等;
3)分子对接:选择合适的分子对接算法及打分函数,根据打分结果、小分子结合姿势以及配体-受体相互作用,筛选出最优的小分子用于后续研究;
4)ADMET预测:对初步筛选出的小分子进行药代动力学预测和毒性分析,为后续的药物优化和临床试验提供有力的数据支持,减少候选化合物因不良的药代动力学特征和毒性问题而导致的研发失败;
5)分子动力学模拟:对通过分子对接得到的配体-受体复合物结构进行分子动力学模拟,研究小分子与蛋白质之间的动态相互作用以及复合物体系在模拟期间的稳定性;
6)结合自由能计算:更全面地评估配体与受体之间的相互作用,精确预测配体与受体之间的结合亲和力;
7)生物测活:对最终筛选出来的化合物进行生物实验验证,以确认其活性。
03 虚拟筛选典型案例--靶向Mpro抑制剂的研究
Mpro作为SARS-CoV-2(新型冠状病毒)复制周期中的关键酶,负责切割病毒多聚蛋白为功能成熟的非结构蛋白,对病毒的生命周期至关重要。因此,Mpro成为抗COVID-19药物开发的重要靶点之一。通过抑制Mpro的活性,可以有效阻断病毒的复制过程,为治疗COVID-19提供新的策略。然而,鉴于病毒的高度变异性和药物开发的复杂性,需要更快、更有效的方式去加速药物研发进程,而虚拟筛选技术正是这样一种解决方案。
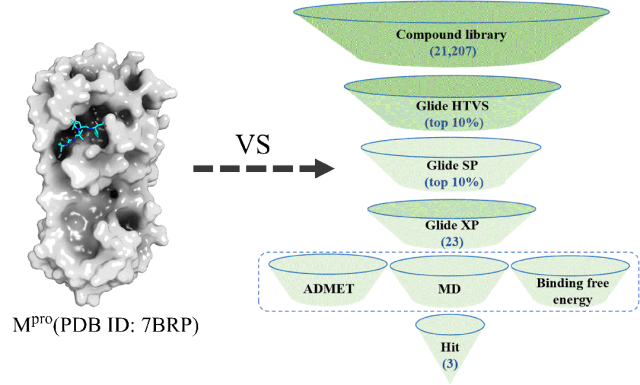
图2. 本案例的虚拟筛选流程图
该研究采用基于分子对接的虚拟筛选策略进行Mpro抑制剂的发现[3](图2)。首先从蛋白质数据库中选择具有高分辨率且在自然界中高度保守的Mpro晶体结构(PDB ID: 7BRP),使用Schrodinger的Protein Preparation Wizard模块对蛋白进行结构优化,包括质子化、去水和能量最小化等步骤,以确保蛋白质结构的合理性。此外,该研究人员收集了21,207个配体用于对Mpro的计算筛选,并在OPLS3e力场下优化生成其三维构象。随后,以共晶配体所在位置为对接位点,创建了一个尺寸为20 Å×20 Å×20 Å的对接盒子,以确保小分子能够在合理的空间范围内与蛋白质相互作用。最后,在Glide模块中,将优化后的配体对接到结合位点。
整个分子对接筛选过程分为三个阶段,第一阶段将对接精度设置为HTVS,对所有化合物进行高通量虚拟筛选,根据打分排序选择前10%的结果进行第二阶段的筛选。第二阶段将对接精度设置为SP,对初步筛选出来的小分子进行更加细致的筛选。Glide SP在对接精度上要优于Glide HTVS,但需要耗费较长的时间。第三阶段,使用对接精度更高的Glide XP对SP对接得到的前10%化合物进行筛选,最终获得23个initial hits。接着对这23个initial hits进行ADMET预测、MD模拟和结合自由能计算,发现3个final hits (F2679-0163、F6355-0442、Asinex-8250,如图3所示)展现出良好的药代动力学特性以及稳定的分子动态行为。结合模式分析表明这3个化合物与靶标蛋白之间形成了丰富的氢键网络,从而维持了化合物与蛋白之间的稳定性。
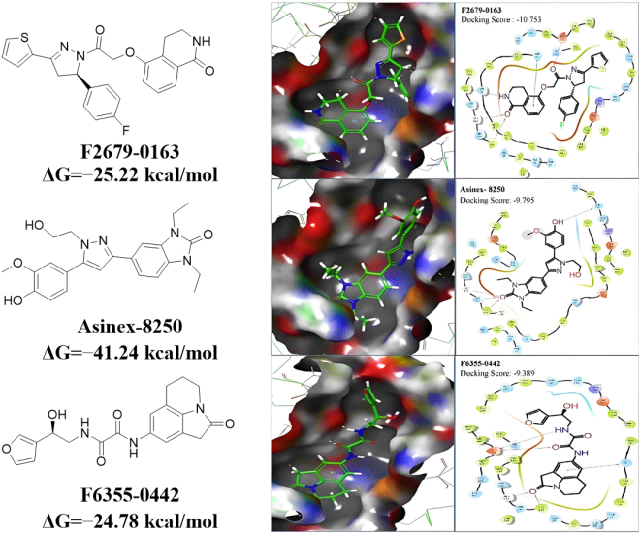
图3. 筛选出来的3个小分子以及与蛋白之间的结合模式
该研究运用合理的药物筛选策略,筛选出3个配体小分子,可能成为针对SARS-CoV-2主要蛋白酶的潜在药物分子。但是,这些通过计算模拟筛选得到的结果还有待通过体外实验进行验证。
04 普美瑞云平台进行虚拟筛选的操作流程
磷酸二酯酶Ⅱ型(PDE2)是一种双底物特异性酶,能够双重水解cAMP和cGMP,作为中枢神经系统(CNS)疾病(包括阿尔茨海默病)的治疗靶点而备受关注。在这里,我们将展示如何利用普美瑞云平台上的虚拟筛选模块从庞大的化合物库中高效筛选出潜在的PDE2抑制剂,具体操作流程如下:
1.虚拟筛选任务提交:
①访问普美瑞生物官方网站(https://pumeirui.com/)选择虚拟筛选模块
②进入虚拟筛选界面,点击“立即运行”启动虚拟筛选模块
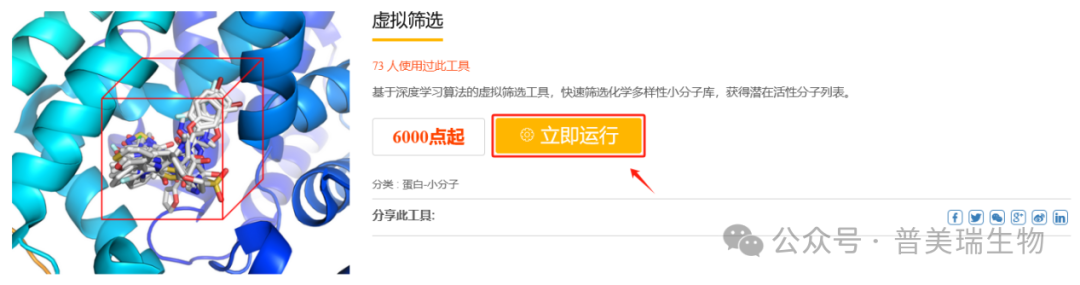
③在表单中填写任务名以及选择对接算法
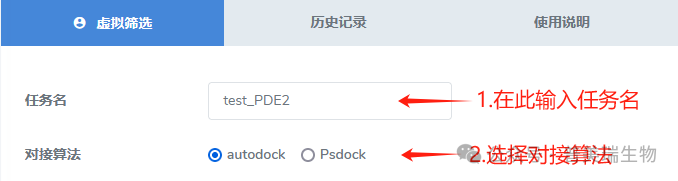
目前该平台提供了两种分子对接算法:Autodock Vina和Psdock,用户可以选择任何一种算法进行虚拟筛选。
④蛋白结构准备

蛋白结构可通过两种方式获得:方法1. 使用内置靶标库,用户可通过PDB_ID,TTD_ID,Protein_name,Gene_name,UniprotID,Accession number共计6种方式搜索蛋白结构,随后选定相应的PDB ID及其对接位点,示例如下:

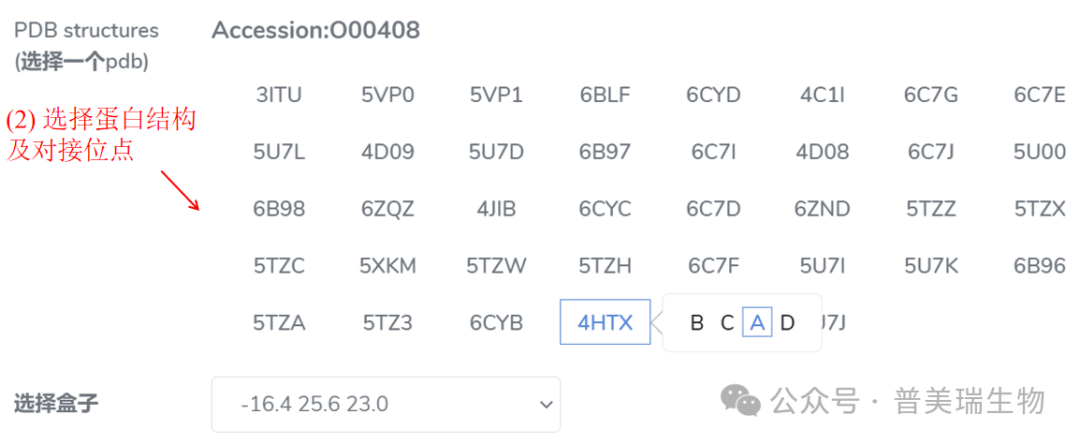
内置靶标库中的4HTX是一个四条链的多聚体结构,每条链上均有小分子结合位点,因此需用户选定某条链进行分子对接,如本例中我们选取A链进行对接。随后选择对接盒子,若该链上有多个小分子接位点,则需要从“选择盒子”下拉条进行选择,其中数字代表盒子的中心坐标。完成后点击“确定”即可返回参数设定主界面从分子视图中查看对接盒子包含的蛋白区域。
方法2. 用户自定义蛋白结构。如果在内置靶标库中找不到所要蛋白,用户可通过输入PDB ID选定一个pdb文件或从本地上传PDB文件两种方式来定义受体蛋白。

随后通过点击图像中蛋白上相应原子作为对接盒子中心并自动生成坐标,通常选择复合物结构中小分子配体上的原子为盒子中心;在“盒子大小”中输入x、y、z三个方向上的盒子长度,调节对接盒子大小,其默认参数为28Å×28Å×28Å。选定后点击“Apply”生成对接盒子,在分子视图中可查看盒子包含的区域。

⑤确认选择的结构文件和对接参数
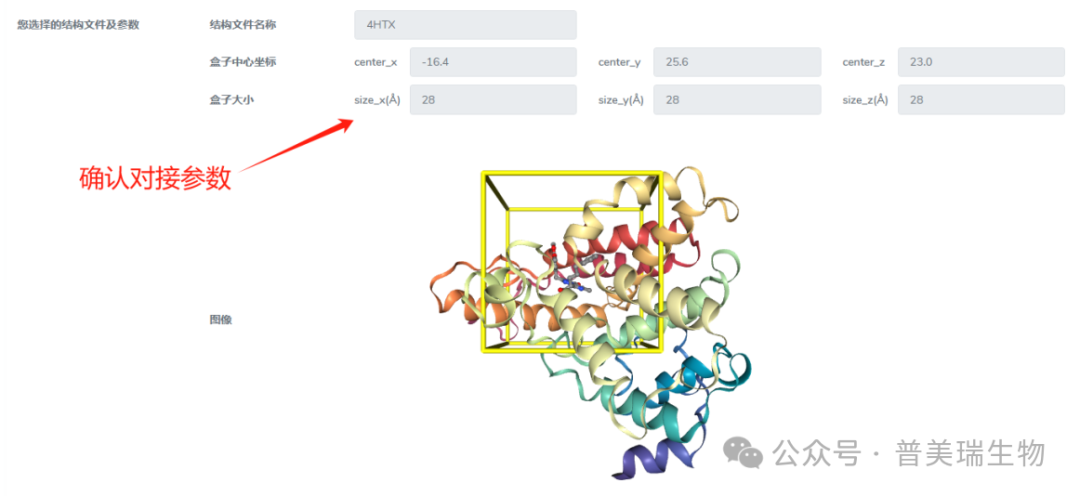
⑥选择小分子化合物库以及是否需要AI打分
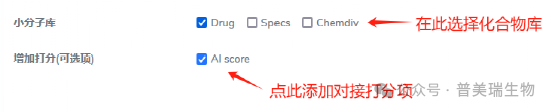
普美瑞云平台提供Drug,Specs和Chemdiv化合物库可选择:Drug库包含3110个已知药物分子,Specs库包括约20万个小分子,Chemdiv库包含160万个小分子。用户可根据自身需求选择对应化合物库。若用户有其他感兴趣化合物库可联系普美瑞云平台定制。虚拟筛选默认打分为vina对接打分(选择autodock vina为对接程序时),或基于统计势的打分函数(选择Psdock为对接程序时),以及机器学习方法修正的vina对接打分函数RF score作为参考。普美瑞云平台还特别提供了基于AI模型的打分函数,可通过勾选“AI score”进行额外的打分项计算。
⑦阅读免责声明,同意后提交
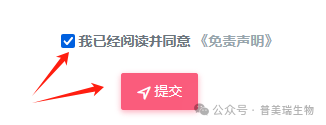
⑧核对要上传信息参数,确认无误后提交。任务一旦提交参数无法修改,请用户认真核对参数再提交
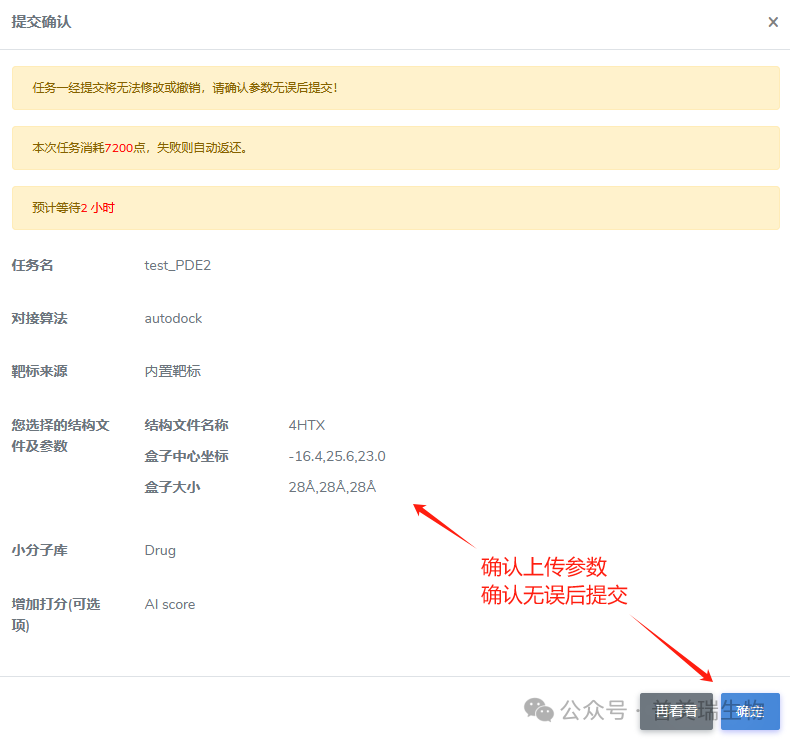
至此,任务提交完毕。
2.虚拟筛选结果查看
①用户登录后,进入控制台—>虚拟筛选页面,点击历史记录
②在历史任务列表中找到想要查看的任务,并点击查看
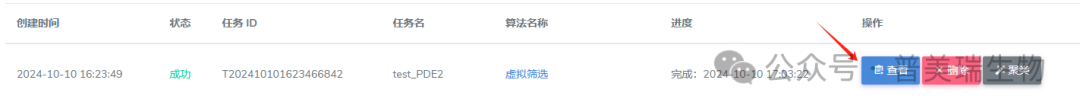
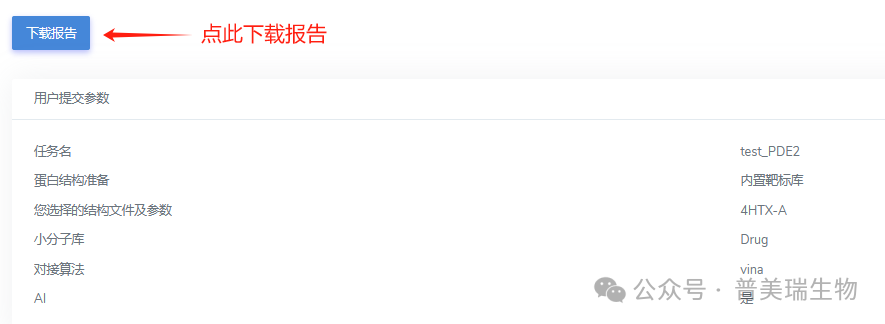
③查看、下载报告
总结
虚拟筛选作为一种高效的药物发现工具,凭借其强大的计算能力,能够精准地预测化合物的生物活性,从而显著地加速了新药从初步发现到深入开发的整个进程。这一技术的运用,不仅大大地缩减了药物研发所需的时间成本,还提升了研发过程的整体效率。随着近年来计算能力的持续飞跃、生物信息学领域的蓬勃发展以及实验技术的不断革新,虚拟筛选技术在药物设计这一关键领域内展现出了愈发重要的影响力,并持续推动着该领域的边界拓展。展望未来,相关领域的科学研究将致力于进一步优化虚拟筛选的方法论,力求在提升筛选准确性的同时,也大幅提高筛选过程的效率,以期能够为治疗包括罕见病、复杂慢性病在内的各类疾病,提供更加多样化、更具潜力的候选药物库,从而为全球医疗健康事业贡献更为深远的价值。
参考文献
[1]Fischer E. Einfluss der Configuration auf die Wirkung der Enzyme[J]. Berichte der deutschen chemischen Gesellschaft, 1894, 27(3): 2985-2993.
[2]Koshland Jr D. Application of a theory of enzyme specificity to protein synthesis[J]. Proc Natl Acad Sci, 1958, 44(2): 98-104.
[3]Yadav R, Imran M, Dhamija P, et al. Virtual screening, ADMET prediction and dynamics simulation of potential compounds targeting the main protease of SARS-CoV-2[J]. Journal of Biomolecular Structure and Dynamics, 2021, 39(17): 6617-6632.



























 433
433

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








