写在前面
ggClusterNet发展到现在已经被超过100篇文章引用,但是也不妨碍其中有许多反人类的操作。所以我都要进行更新,去年我更新了网络流程函数network.pip,可以封装多组网络数据,并且完成多网络统一标尺的展示,并且我还提到,可以无缝衔接后续分析。
但是如何衔接后续分析,我一直没有做一个详细的案例,这里我进行一个展示,大家有一个详细的认识,最后也欢迎大家应用ggClusterNet。
实战
网络流程分析-2023年末更新
在分析之前,如果你没使用过r语言,可以如此安装R包:
install.packages("BiocManager")
library(BiocManager)
install("remotes")
install("tidyverse")
install("tidyfst")
install("igraph")
install("sna")
install("phyloseq")
install("ggalluvial")
install("ggraph")
install("WGCNA")
install("ggnewscale")
install("pulsar")
install("patchwork")
remotes::install_github("taowenmicro/EasyStat")
remotes::install_github("taowenmicro/ggClusterNet")开始实战
主函数,用于网络计算和可视化
library(ggClusterNet)
library(phyloseq)
library(tidyverse)
data(ps)
otupath = "./"
# 新网络分析-2023年末更新#--------
netpath = paste(otupath,"/network.new/",sep = "")
dir.create(netpath)
rank.names(ps)
library(ggrepel)
library(igraph)
detach("package:MicrobiotaProcess")
# 8.1 网络分析主函数#--------
tab.r = network.pip(
ps = ps,
N = 400,
# ra = 0.05,
big = TRUE,
select_layout = FALSE,
layout_net = "model_maptree2",
r.threshold = 0.6,
p.threshold = 0.05,
maxnode = 2,
method = "spearman",
label = TRUE,
lab = "elements",
group = "Group",
fill = "Phylum",
size = "igraph.degree",
zipi = TRUE,
ram.net = TRUE,
clu_method = "cluster_fast_greedy",
step = 100,
R=10,
ncpus = 1
)
# 建议保存一下输出结果为R对象,方便之后不进行相关矩阵的运算,节约时间
saveRDS(tab.r,paste0(netpath,"network.pip.sparcc.rds"))
tab.r = readRDS(paste0(netpath,"network.pip.sparcc.rds"))
dat = tab.r[[2]]
cortab = dat$net.cor.matrix$cortab
# 大型相关矩阵跑出来不容易,建议保存,方便各种网络性质的计算
saveRDS(cortab,paste0(netpath,"cor.matrix.all.group.rds"))
cor = readRDS(paste0(netpath,"cor.matrix.all.group.rds"))
#-提取全部图片的存储对象
plot = tab.r[[1]]
# 提取网络图可视化结果
p0 = plot[[1]]
ggsave(paste0(netpath,"plot.network.pdf"),p0,width = 12,height = 5)
ggsave(paste0(netpath,"plot.network2.pdf"),p0,width = 16,height = 10)
zipi展示
plot[[2]]
与随机网络的比对
plot[[3]]
网络属性计算-丰富的网络属性,16个
i = 1
id = names(cor)
for (i in 1:length(id)) {
igraph= cor[[id[i]]] %>% make_igraph()
dat = net_properties.4(igraph,n.hub = F)
head(dat,n = 16)
colnames(dat) = id[i]
if (i == 1) {
dat2 = dat
} else{
dat2 = cbind(dat2,dat)
}
}
head(dat2)
FileName <- paste(netpath,"net.network.attribute.data.csv", sep = "")
write.csv(dat2,FileName,quote = F)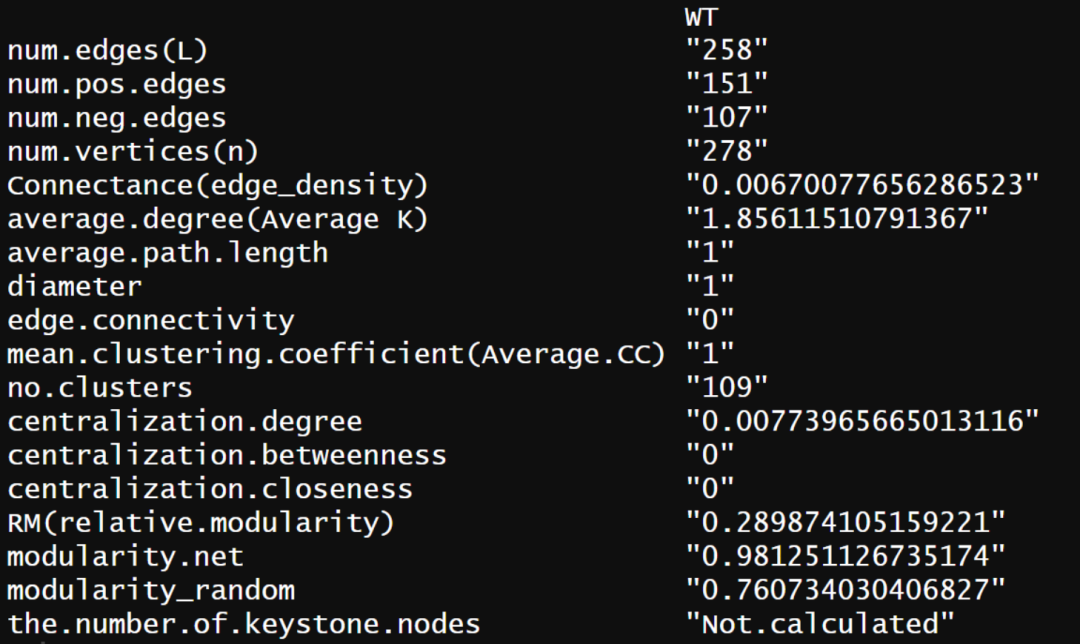
如何计算单个样本王璐属性用于和其他指标关联
for (i in 1:length(id)) {
pst = ps %>% subset_samples.wt("Group",id[i]) %>% remove.zero()
dat.f = netproperties.sample(pst = pst,cor = cor[[id[i]]])
# head(dat.f)
if (i == 1) {
dat.f2 = dat.f
} else{
dat.f2 = rbind(dat.f2,dat.f)
}
}
head(dat.f2)
FileName <- paste(netpath,"net.network.attribute.data.sample.csv", sep = "")
write.csv(dat.f2,FileName,quote = F)
map= sample_data(ps)
map$ID = row.names(map)
map = map %>% as.tibble()
dat3 = dat.f2 %>% rownames_to_column("ID") %>% inner_join(map,by = "ID")
FileName <- paste(netpath,"net.network.attribute.data.sample.add.group.info.csv", sep = "")
write.csv(dat3,FileName,quote = F)
节点属性计算
for (i in 1:length(id)) {
igraph= cor[[id[i]]] %>% make_igraph()
nodepro = node_properties(igraph) %>% as.data.frame()
nodepro$Group = id[i]
head(nodepro)
colnames(nodepro) = paste0(colnames(nodepro),".",id[i])
nodepro = nodepro %>%
as.data.frame() %>%
rownames_to_column("ASV.name")
# head(dat.f)
if (i == 1) {
nodepro2 = nodepro
} else{
nodepro2 = nodepro2 %>% full_join(nodepro,by = "ASV.name")
}
}
head(nodepro2)
FileName <- paste(netpath,"net.node.attribute.data.sample.csv", sep = "")
write_csv(nodepro2,FileName)可定制网络输出
dat = tab.r[[2]]
node = dat$net.cor.matrix$node
edge = dat$net.cor.matrix$edge
head(edge)
head(node)
node2 = add.id.facet(node,"Group")
head(node2)
p <- ggplot() + geom_segment(aes(x = X1, y = Y1, xend = X2, yend = Y2,color = cor
),
data = edge, size = 0.03,alpha = 0.1) +
geom_point(aes(X1, X2,
fill = Phylum,
size = igraph.degree),
pch = 21, data = node,color = "gray40") +
facet_wrap(.~ label,scales="free_y",nrow = 1) +
# geom_text_repel(aes(X1, X2,label = elements),pch = 21, data = nodeG) +
# geom_text(aes(X1, X2,label = elements),pch = 21, data = nodeG) +
scale_colour_manual(values = c("#6D98B5","#D48852")) +
# scale_fill_hue()+
scale_size(range = c(0.8, 5)) +
scale_x_continuous(breaks = NULL) +
scale_y_continuous(breaks = NULL) +
theme(panel.background = element_blank(),
plot.title = element_text(hjust = 0.5)
) +
theme(axis.title.x = element_blank(),
axis.title.y = element_blank()
) +
theme(legend.background = element_rect(colour = NA)) +
theme(panel.background = element_rect(fill = "white", colour = NA)) +
theme(panel.grid.minor = element_blank(), panel.grid.major = element_blank())
p
#-8.6 zipi可视化-定制#-----
dat.z = dat$zipi.data
head(dat.z)
x1<- c(0, 0.62,0,0.62)
x2<- c( 0.62,1,0.62,1)
y1<- c(-Inf,2.5,2.5,-Inf)
y2 <- c(2.5,Inf,Inf,2.5)
lab <- c("peripheral",'Network hubs','Module hubs','Connectors')
roles.colors <- c("#E6E6FA","#DCDCDC","#F5FFFA", "#FAEBD7")
tab = data.frame(x1 = x1,y1 = y1,x2 = x2,y2 = y2,lab = lab)
tem = dat.z$group %>% unique() %>% length()
for ( i in 1:tem) {
if (i == 1) {
tab2 = tab
} else{
tab2 = rbind(tab2,tab)
}
}
p <- ggplot() +
geom_rect(data=tab2,
mapping=aes(xmin=x1,
xmax=x2,
ymin=y1,
ymax=y2,
fill = lab))+
guides(fill=guide_legend(title="Topological roles")) +
scale_fill_manual(values = roles.colors)+
geom_point(data=dat.z,aes(x=p, y=z,color=module)) + theme_bw()+
guides(color= F) +
ggrepel::geom_text_repel(data = dat.z,
aes(x = p, y = z,
color = module,label=label),size=4)+
# facet_wrap(.~group) +
facet_grid(.~ group, scale='free') +
theme(strip.background = element_rect(fill = "white"))+
xlab("Participation Coefficient")+ylab(" Within-module connectivity z-score")
p
# 8.7 随机网络,幂率分布#-------
dat.r = dat$random.net.data
p3 <- ggplot(dat.r) +
geom_point(aes(x = ID,y = network,
group =group,fill = group),pch = 21,size = 2) +
geom_smooth(aes(x = ID,y = network,group =group,color = group))+
facet_grid(.~g,scales = "free") +
theme_bw() + theme(
plot.margin=unit(c(0,0,0,0), "cm")
)
p3
多网络比对-网络显著性
dat = module.compare.net.pip(
ps = NULL,
corg = cor,
degree = TRUE,
zipi = FALSE,
r.threshold= 0.8,
p.threshold=0.05,
method = "spearman",
padj = F,
n = 3)
res = dat[[1]]
head(res)
FileName <- paste(netpath,"net.compare.diff.sig.csv", sep = "")
write.csv(res,FileName,quote = F)
网络稳定性-模块比对
library(tidyfst)
res1 = module.compare.m(
ps = NULL,
corg = cor,
zipi = FALSE,
zoom = 0.2,
padj = F,
n = 3)
#不同分组使用一个圆圈展示,圆圈内一个点代表一个模块,相连接的模块代表了相似的模块。p1 = res1[[1]]
p1
#--提取模块的OTU,分组等的对应信息
dat1 = res1[[2]]
head(dat1)
#模块相似度结果表格
dat2 = res1[[3]]
head(dat2)
dat2$m1 = dat2$module1 %>% strsplit("model") %>%
sapply(`[`, 1)
dat2$m2 = dat2$module2 %>% strsplit("model") %>%
sapply(`[`, 1)
dat2$cross = paste(dat2$m1,dat2$m2,sep = "_Vs_")
# head(dat2)
dat2 = dat2 %>% filter(module1 != "none")
p2 = ggplot(dat2) + geom_bar(aes(x = cross,fill = cross)) +
labs(x = "",
y = "numbers.of.similar.modules"
)+ theme_classic()
p2
#--发现分组1和分组3网络更相似一些
FileName <- paste(netpath,"module.compare.groups.pdf", sep = "")
ggsave(FileName, p1, width = 10, height = 10)
FileName <- paste(netpath,"numbers.of.similar.modules.pdf", sep = "")
ggsave(FileName, p2, width = 8, height = 8)
FileName <- paste(netpath,"module.otu.csv", sep = "")
write.csv(dat1,FileName, quote = F)
FileName <- paste(netpath,"module.compare.groups.csv", sep = "")
write.csv(dat2,FileName, quote = F)

网络稳定性-鲁棒性
鲁棒性计算需要物种丰富,所以即使计算好了相关矩阵,也需要输入ps对象
-8.92 网络稳定性-去除关键节点-网络鲁棒性#-----
# 鲁棒性计算需要物种丰富,所以即使计算好了相关矩阵,也需要输入ps对象
library(patchwork)
res2= Robustness.Targeted.removal(ps = ps,
corg = cor,
degree = TRUE,
zipi = FALSE
)
p3 = res2[[1]]
p3
#提取数据
dat4 = res2[[2]]
# dir.create("./Robustness_Random_removal/")
path = paste(netpath,"/Robustness_Random_removal/",sep = "")
fs::dir_create(path)
write.csv(dat4,
paste(path,"random_removal_network.csv",sep = ""))
ggsave(paste(path,"random_removal_network.pdf",sep = ""), p3,width = 8,height = 4)
#-8.93 网络稳定性-随即取出任意比例节点-网络鲁棒性#---------
res3 = Robustness.Random.removal(ps = ps,
corg = cortab,
Top = 0
)
p4 = res3[[1]]
p4
#提取数据
dat5 = res3[[2]]
# head(dat5)
path = paste(netpath,"/Robustness_Targeted_removal/",sep = "")
fs::dir_create(path)
write.csv(dat5,
paste(path,"Robustness_Targeted_removal_network.csv",sep = ""))
ggsave(paste(path,"Robustness_Targeted_removal_network.pdf",sep = ""), p4,width = 8,height = 4)
网络稳定性-负相关比例
#-8.94 网络稳定性-计算负相关的比例#----
res4 = negative.correlation.ratio(ps = ps,
corg = cortab,
# Top = 500,
degree = TRUE,
zipi = FALSE)
p5 = res4[[1]]
p5
dat6 = res4[[2]]
#-负相关比例数据
# head(dat6)
path = paste(netpath,"/Vulnerability/",sep = "")
fs::dir_create(path)
write.csv(dat6,
paste(path,"Vnegative.correlation.ratio_network.csv",sep = ""))
ggsave(paste(path,"negative.correlation.ratio_network.pdf",sep = ""), p5,width = 4,height = 4)
网络稳定性-群落稳定性
#-8.95 网络稳定性-群落稳定性-只有pair样本使用#-----
treat = ps %>% sample_data()
treat$pair = paste( "A",c(rep(1:6,3)),sep = "")
# head(treat)
sample_data(ps) = treat
#一般性的处理,没有时间梯度的,这里设定time为F,意味着每两个群落的组合都进行比较
res5 = community.stability( ps = ps,
corg = cor,
time = FALSE)
p6 = res5[[1]]
p6
dat7 = res5[[2]]
path = paste(netpath,"/community.stability/",sep = "")
fs::dir_create(path)
write.csv(dat7,
paste(path,"community.stability.data.csv",sep = ""))
ggsave(paste(path,"community.stability..boxplot.pdf",sep = ""), p6,width = 4,height = 4)
#8.96 网络稳定性-网络抗毁性#------
library("pulsar")
res6 = natural.con.microp (
ps = ps,
corg = cor,
norm = TRUE,
end = 150,# 小于网络包含的节点数量
start = 0
)
p7 = res6[[1]]
p7
dat8 = res6[[2]]
path = paste(netpath,"/Natural_connectivity/",sep = "")
fs::dir_create(path)
write.csv(dat8,
paste(path,"/Natural_connectivity.csv",sep = ""))
ggsave(paste(path,"/Natural_connectivity.pdf",sep = ""), p7,width = 5,height = 4)
根际互作生物学研究室 简介根际互作生物学研究室是沈其荣院士土壤微生物与有机肥团队下的一个关注于根际互作的研究小组。本小组由袁军教授带领,主要关注:1.植物和微生物互作在抗病过程中的作用;2 环境微生物大数据整合研究;3 环境代谢组及其与微生物过程研究体系开发和应用。团队在过去三年中在 Nature Communications,ISME J,Microbiome,SCLS,New Phytologist,iMeta,Fundamental Research, PCE,SBB,JAFC(封面),Horticulture Research,SEL(封面),BMC plant biology等期刊上发表了多篇文章。欢迎关注 微生信生物 公众号对本研究小组进行了解。
翻译:牛国庆
修改:文涛
排版:杨雯儀
审核:袁军团队工作及其成果 (点击查看)了解 交流 合作-
小组负责人邮箱 袁军:junyuan@njau.edu.cn;
小组成员文涛:taowen@njau.edu.cn等
团队公众号:微生信生物 添加主编微信,或者后台留言。
加主编微信 加入群聊
目前营销人员过多,为了维护微生信生物3年来维护的超5500人群聊,目前更新进群要求:
1.仅限相关专业或研究方向人员添加,必须实名,不实名则默认忽略。
2.非相关专业的其他人员及推广宣传人员禁止添加。
3.添加主编微信需和简单聊一聊专业相关问题,等待主编判断后,可拉群。
4 微生信生物VIP微信群不受限制,给微生信生物供稿一次即可加入(群里发送推文代码+高效协助解决推文运行等问题+日常问题咨询回复)。























 2259
2259

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








