
研究论文
● 期刊:Cell Host & Microbe(IF:20.6)
● DOI:https://doi.org/10.1016/j.chom.2024.12.001
●原文链接: https://www.cell.com/cell-host-microbe/fulltext/S1931-3128(24)00446-3
● 第一作者:Jordy Evan Sulaiman
● 通讯作者:Ophelia S. Venturelli(ophelia.venturelli@duke.edu)
● 发表日期:2025-1-8
● 主要单位:
美国威斯康星大学麦迪逊分校、美国杜克大学
摘要Summary
艰难梭菌(Clostridioides difficile)能够短暂或长期定殖于人类肠道,并可能引发感染风险。这一过程受到人类肠道微生物群中复杂的分子与生态相互作用的深刻影响。通过研究艰难梭菌在人体肠道群落中经历数百代的动态变化,我们发现其定殖模式包括稳定共存、不稳定性或竞争性排除。降低碳水化合物浓度使包含艰难梭菌和常见肠道共生菌普通拟杆菌的群落从竞争性排除转变为共存,而这种转变得益于交互喂养的增强。在这种环境下,艰难梭菌发生了两处关键突变,使其代谢生态位从依赖脯氨酸转向依赖葡萄糖。这些代谢改变显著影响了艰难梭菌与肠道微生物群的种间相互作用,同时降低了小鼠的疾病的严重程度。综上所述,与普通拟杆菌的相互作用在塑造艰难梭菌的长期生长动态和进化适应中发挥了关键作用,为制定针对艰难梭菌的防治策略提供了重要启示。
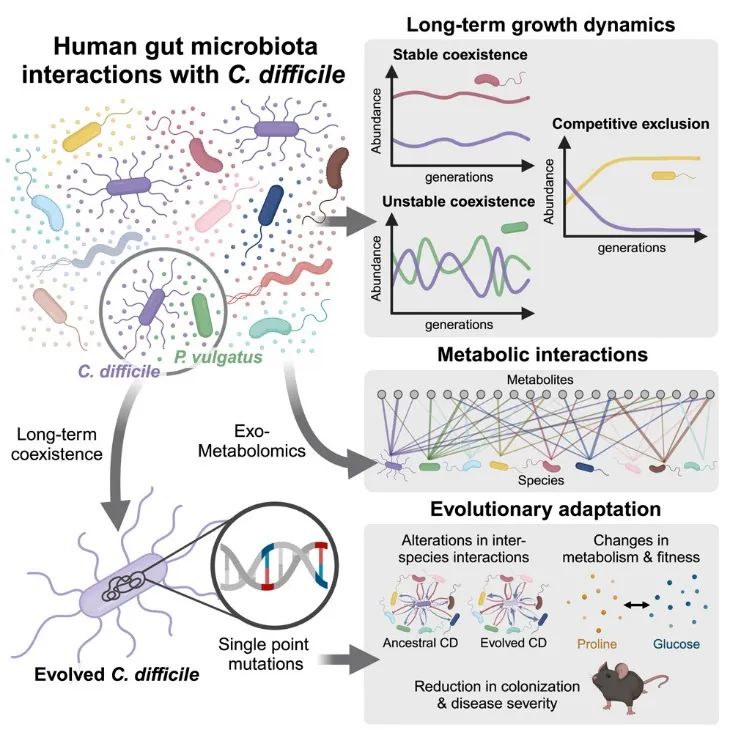
结果Results
人类肠道菌种在群落中对艰难梭菌的长期生长具有差异性影响
艰难梭菌(C. difficile)可在不同生命阶段的个体中定殖,持续时间各不相同,而人们对决定这些动态的因素仍然知之甚少。由于肠道微生物生态是艰难梭菌定殖的重要决定因素,我们采用了自下而上的微生物群落实验,研究种间相互作用如何塑造艰难梭菌的长期生长动态。为此,我们将艰难梭菌 DSM 27147(流行性核型 027 的参考菌株 R20291)与 9 种多样的人类肠道菌种共同培养在支持其生长的特定培养基中(图 1A 和 S1A;表 S1 和 S2)。这些人类肠道菌种在不同个体中高度普遍存在,并涵盖了人类肠道微生物群的系统发育多样性。该群落包括以下物种:闪烁梭菌(Clostridium scindens, CS):此前研究表明其在无菌小鼠中可抑制艰难梭菌的生长;梭菌(Clostridium hiranonis, CH):通过代谢生态位重叠抑制艰难梭菌,并导致其代谢发生显著变化;拟杆菌属物种(包括单形拟杆菌(Bacteroides uniformis, BU)、多形拟杆菌(Bacteroides thetaiotaomicron, BT)和普通拟杆菌(Phocaeicola vulgatus, PV):在不同环境下可抑制或促进艰难梭菌的生长。此外,脱硫弧菌(Desulfovibrio piger, DP)、迟缓埃格特菌(Eggerthella lenta, EL)、产气柯林斯菌(Collinsella aerofaciens, CA) 和 普雷沃菌(Prevotella copri, PC) 与艰难梭菌的相互作用也已在短时间尺度(约 5–10 代)内得到广泛研究。
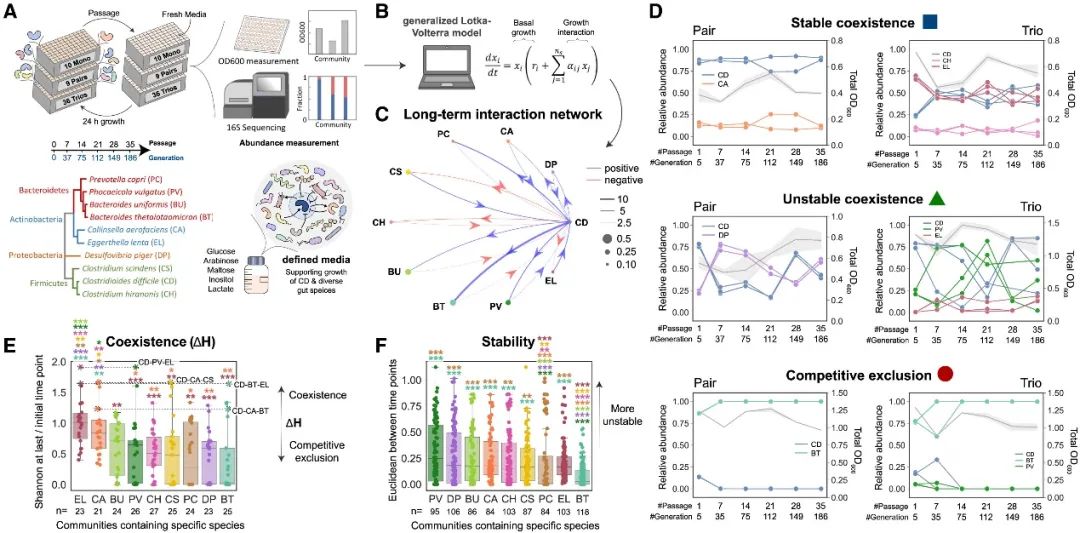
图1 | 艰难梭菌在人类肠道群落中的长期生长动态
(A)艰难梭菌在人类肠道群落中长期生长实验示意图。根据每个物种的 16S rRNA 序列生成了系统发育树。
(B)利用时间序列绝对丰度数据推断 gLV 模型参数。
(C)利用 35 次传代的丰度数据推断出艰难梭菌与 9 种肠道菌种之间的长期种间相互作用网络。节点大小代表物种的环境容纳量,边的宽度代表长期种间相互作用系数(aij)的大小。肠道菌种之间的完整 aij 见图 S3A。
(D)艰难梭菌与肠道细菌在成对(左)和三成员(右)群落中的代表性群落动态。由彩色线条连接的数据点表示每个物种的相对丰度(左 y 轴),而带有 95%置信区间阴影的灰色线条表示群落 OD600(右 y 轴)。完整的群落动态见图 S1C(成对)和图 S2A(三成员)。稳定共存、不稳定共存和竞争排斥的分类依据图 S2B。(E)使用最后一个时间点相对于初始时间点的香农多样性来测量所有包含 x 轴所示特定物种的成对和三成员群落中共存程度(ΔH)。
(F)在所有包含 x 轴所示特定物种的成对和三成员群落中,35 次传代过程中各时间点测量值之间欧几里得距离的箱线图。对于(E)和(F),由于组间重叠群落的存在,某些数据点跨越了较大的多样性空间,从而导致了变异性(示例数据点已突出显示)。图上方的星号表示该组与另一组之间双向曼 - 惠特尼 U 检验的统计学显著 p 值,星号颜色与另一组对应。*p < 0.05,**p < 0.01,***p < 0.001。确切的 p 值见表 S4。
我们组装了所有可能的双物种组合(共9种)和三物种组合(共36种),其中包含艰难梭菌(C. difficile)和肠道细菌。这些菌群培养了24小时后,取少量样品以40倍稀释接种至新鲜培养基,连续传代35次(约186代)(详见STAR方法)。通过16S rRNA测序确定每种物种的相对丰度,并乘以各时间点的总生物量,估算每种物种的绝对丰度。此外,我们单独培养了每种物种,采用相同的实验设计(图S1B)。结果显示,艰难梭菌在60%的群落中持续存在(相对丰度 >1%,直到最后一次传代),包括7/9的双物种群落和20/36的三物种群落(图S1C和S2A)。然而,在84%的群落中,艰难梭菌的绝对丰度从初代到最后一次传代显著下降(图S1D)。某些物种(如PC和BT)在明显抑制艰难梭菌的群落中被显著富集。所有拟杆菌属(Bacteroides)物种在单一培养或与艰难梭菌共培养时均显著降低了培养基的pH值(培养基pH从7.1降至约4.9–5.3;图S1E)。虽然其他肠道菌在单一培养或共培养中未显著降低pH值,但PC仅在与艰难梭菌共培养时显著酸化了培养基(pH从7.1降至5.1),这可能与PC对艰难梭菌的抑制效应有关(图S1D)。
为了量化人类肠道菌种对艰难梭菌长期生长的影响,我们采用广义Lotka-Volterra(gLV)模型,对单一培养、双物种和三物种群落在35次传代中的物种丰度时间序列数据进行了拟合(图1B;表S3 DATASET001;详见STAR方法)。gLV模型是一种动态生态模型,可根据各物种的生长和与群落成员的双物种相互作用预测群落动态。该模型广泛用于解析物种间相互作用并预测不同环境中的群落组装过程。推导出的gLV物种间相互作用系数量化了特定物种对另一物种长期生长的影响(图S3A)。模型与实验数据的拟合度较高(Pearson相关系数R = 0.89–0.91,p = 3.6E−42至2.3E−51;图S3B)。为了评估模型对验证数据的预测性能,我们进行了留一交叉验证(图S3C;详见STAR方法)。由于用于训练模型的数据有限,特别是肠道菌的相关数据,模型仅对艰难梭菌的丰度预测表现出中等一致性(艰难梭菌的Pearson相关系数R = 0.36–0.60,总体相关系数R = 0.63–0.70,p = 1.5E−13至1.9E−18)。此外,gLV模型未能捕捉物种间相互作用的进化变化,这可能是预测性能较低的原因之一。
与基于艰难梭菌(C. difficile)在人类肠道群落中短时间尺度测量推导出的gLV物种间相互作用网络(主要以负相互作用为主)相比,长期生态相互作用推导出的网络显示出较高频率的正相互作用(图1C)。在这一时间尺度上,PV和DP与艰难梭菌之间表现出双向正相互作用,而理论上这可能会导致群落动态的不稳定性(图1D,图S1C,图S2A和图S2B)。相反,EL与艰难梭菌之间表现出输出正相互作用和输入负相互作用。这种拓扑结构可能形成负反馈回路,从而稳定群落动态,并解释了观察到的EL与艰难梭菌之间的稳定共存现象。
然而,经常排斥艰难梭菌的物种(如PC和BT)也显示出与艰难梭菌之间的输出负相互作用和输入正相互作用。这与竞争性排除动态的稳定性相一致(图S2B)。CS和CH在双物种共培养中被艰难梭菌








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章

















 3528
3528

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








