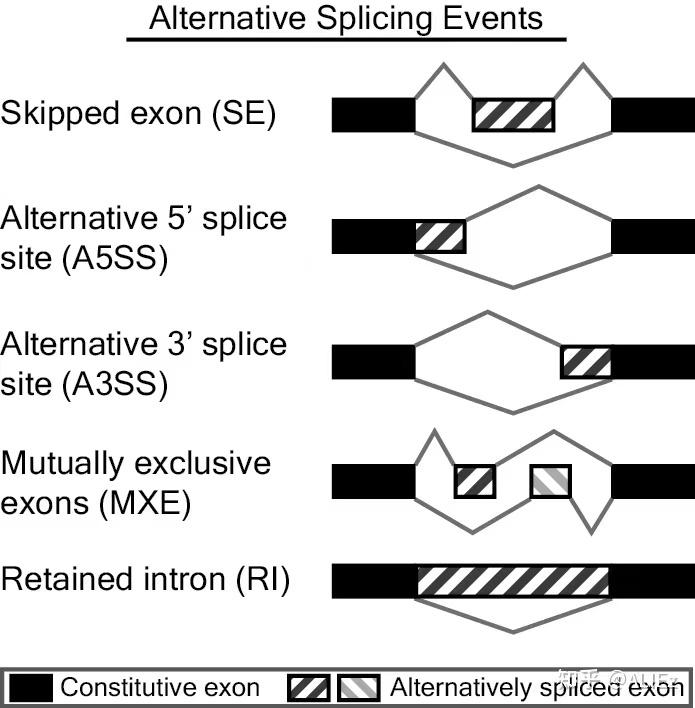
rmats4.1.2版本是2021年最新更新的rmats版本,该工具可以通过rna-seq数据分析mRNA可变剪切的情况,是各大可变剪切工具中引用最多的一种!可以分析以下几种可变剪切方式
下面是原作者介绍的所需安装依赖,但是并没有具体写出安装的代码,鸭鸭呕心沥血的整理了自己的安装代码,和大家分享。

鸭鸭是在win11系统上安装了wsl,装的是ubuntu20.04,丝滑的安装了最新版的anaconda,然后安装以下各种包,最后成功跑出数据结果,以下是鸭鸭的安装经验分享!
创建conda 环境
conda create -n rmats python=3.6.12下载rmats4.1.2并解压,下载地址是https://github.com/Xinglab/rmats-turbo
安装cython,鸭鸭装了这么多内容解决n个bug的,但也可能只装python版本的cython就可以了呢
pip install cython
sudo apt-get install cython
sudo apt-get install build-essential安装blas和lappack
sudo apt-get install libblas-dev
sudo apt-get install liblapack-dev安装gsl,注意如果没有update过一定要先update才能安装成功喔
sudo apt-get update
sudo apt-get install libgsl0-dev安装gfortran,fort77也一定要装,不然会gg的
sudo apt-get install gfortran
sudo apt-get install fort77安装cmake
sudo apt-get install cmake好啦,装了这么多,基本必须要装的包就都装好啦,鸭鸭没有单独装gcc,在装以上包的时候就自动装好了呢
虽然optional的内容没有装,但是只要不是pairstats的数据,到这一步只要build一下就可以得到完整的rmats结果了!虽然有报错,但是不用在意。进入解压好的rmats turbo文件夹,运行build就可以配置好环境啦
./build_rmats如果是完美主义者会有使用pair stats model的需要,那optional的包也需要解决一下
安装PAIRADISE,这个是github上的r包,所以没有安装github的话要安装github,没有安装r的话要安装r,还需要安装需要用到的三个R library。相信大家安装r包熟能生巧,就不多做介绍啦。
sudo apt-get install git
sudo apt-get install r-base-core安装samtools,这个是从fastq到bam比对时需要用到的包,如果是从bam文件开始运行rmats的话,是不需要安装的。如果需要安装,可以新建一个环境,然后使用conda就可以轻易安装好了呢。如果不换一个环境,可能会有conflicts产生。
conda install -c samtools openssl=1.0安装star,这个也是从fastq到bam比对时需要用到的包,如果是从bam文件开始运行rmats的话,也是不需要安装的。如果需要安装,可以安装在samtools时新建的环境,然后使用conda就可以轻易安装好了呢
conda install -c bioconda star希望以上的安装攻略对正在看博客的你有所帮助~如果还是安装失败,可以回帖提问或者私聊鸭鸭,鸭鸭每天都会查看帖子,是鸭鸭踩过的坑的话,鸭鸭一定会热心解答的~
可以关注鸭鸭或收藏文章,会持续发布关于可变剪切分析的内容的!喜欢并有余力的话,可以打赏点奶茶钱~
























 1914
1914

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








