Linux环境:可变剪切分析软件rMATS安装、使用与解读
安装前准备
一、安装背景
- 操作系统:Linux(Linux version 5.13.0-52-generic (buildd@lcy02-amd64-046) (gcc (Ubuntu 11.2.0-7ubuntu2) 11.2.0, GNU ld (GNU Binutils for Ubuntu) 2.37))
- 已安装依赖项:
GCC、gfortran(gcc version 11.2.0 (Ubuntu 11.2.0-7ubuntu2))
CMake(cmake version 3.18.4)
二、其他依赖项安装
-
BLAS,LAP ACK 安装
参考:linux关于blas、lapack的安装和使用 -
GNU Scientific Library (GSL 2.5) 安装
参考: Linux 安装GSL库
gsl2.5下载指路:Gsl-2.5 -
python环境配置
强烈建议下载Anaconda安装包管理虚拟环境,后续也能直接使用–conda命令完成软件的安装。
参考:Linux环境下的Anaconda安装及使用安装完成后添加下载源镜像:
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/main
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/free
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/r
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/pro
conda config --add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/msys2
conda config --set show_channel_urls yes
rMATS安装
一、安装
根据Documentation中的指示,直接下载rMATS安装包,解压之后进入安装包当前路径,在base环境中运行即可自动安装:
./build_rmats --conda
可能遇到的报错:
(1)conda环境设置出错,具体表现为:
CommandNotFoundError: Your shell has not been properly configured to use 'conda activate'.
To initialize your shell, run
$ conda init <SHELL_NAME>
Currently supported shells are:
- bash
- fish
- tcsh
- xonsh
- zsh
- powershell
See 'conda init --help' for more information and options.
IMPORTANT: You may need to close and restart your shell after running 'conda init'.
为解决这个问题,需要打开build_rmats文件,在function create_and_activate_conda_env()中conda执行之前,加入以下内容:
function create_and_activate_conda_env() {
### 添加的脚本
if [ -f "[Anaconda路径]/Anaconda3/etc/profile.d/conda.sh" ]; then
. "[Anaconda路径]/Anaconda3/etc/profile.d/conda.sh"
else
export PATH="[Anaconda路径]/Anaconda3/bin:$PATH"
fi
### 添加的脚本
local CONDA_ENV_PATH="${SCRIPT_DIR}/conda_envs/rmats"
conda create --prefix "${CONDA_ENV_PATH}" || return 1
conda activate "${CONDA_ENV_PATH}" || return 1
}
保存更新之后,接着运行
./build_rmats --conda
即可成功安装。
(2)PAIRADISE下载失败
服务器联网原因,可能无法直接从github上下载PAIRADISE包:
###build_rmats 文件中:
function install_pairadise() {
if [[ ! -d PAIRADISE ]]; then
git clone https://github.com/Xinglab/PAIRADISE.git || return 1
fi
Rscript install_r_deps.R || return 1
}
可以直接本地进行下载:
PAIRADISE
将zip上传到服务器中rMATS所在目录下,解压后将文件夹更名为PAIRADISE,重新运行
./build_rmats --conda
若暂时不使用配对模型(paired model),则在安装的时候可以直接运行:
./build_rmats --conda --no-paired-model
二、运行测试
在rMATS解压路径下,直接运行
./test_rmats
检验是否安装成功。
可能接着出现conda设置的问题:

同样地,打开test_rmats文件,在如下位置加上脚本:
function create_and_activate_conda_env() {
### 添加的脚本
if [ -f "[Anaconda路径]/Anaconda3/etc/profile.d/conda.sh" ]; then
. "[Anaconda路径]/Anaconda3/etc/profile.d/conda.sh"
else
export PATH="[Anaconda路径]/Anaconda3/bin:$PATH"
fi
### 添加的脚本
local CONDA_ENV_PATH="${SCRIPT_DIR}/conda_envs/test_rmats"
conda create --prefix "${CONDA_ENV_PATH}" || return 1
conda activate "${CONDA_ENV_PATH}" || return 1
}
保存更新之后再次运行
./test_rmats
成功。
rMATS使用
一、基本操作
在rMATS解压路径,运行
./run_rmats {arguments}
All arguments注释:
-h,--help: 输出帮助信息
--version: 输出版本信息
--gtf GTF: 基因和转录本注释gtf文件
--b1 B1: 文本文件,包含sample_1的bam文件的逗号分隔列表(只有使用bam时)
--b2 B2: 文本文件,包含sample_2的bam文件的逗号分隔列表(只有使用bam时)
--s1 S1: 文本文件,其中包含sample_1的FASTQ文件的逗号分隔列表。如果使用配对read(paired reads),使用":"来分隔对,","来分隔重复(replicates)。(仅当使用fastq时)
--s2 S2: 文本文件,其中包含sample_2的FASTQ文件的逗号分隔列表。如果使用配对read(paired reads),使用":"来分隔对,","来分隔重复。(仅当使用fastq时)
--od OD: post步骤的最终输出目录
--tmp TMP: 中间输出的目录,例如准备步骤(prep step)中的".rmats"文件
-t {paired,single}: read类型,双端测序则为paired,单端测序则为single;默认为paired
--libType {fr-unstranded,fr-firststrand,fr-secondstrand}: 库类型,对特定于链的数据使用fr-firststrand或fr-secondstrand;默认fr-unstranded
--readLength READLENGTH: 每一条read的长度,根据转录结果指定
--anchorLength ANCHORLENGTH: 锚(anchor)的长度;默认1
--tophatAnchor TOPHATANCHOR: aligner中使用的"anchor length"或"overhang length",至少"anchor length"NT必须映射到给定连接的每一端。默认值为6。(仅当使用fastq时)
--bi BINDEX: STAR二进制索引的目录名称(包含SA文件的目录名称)(仅当使用fastq时)
--nthread NTHREAD: 线程数,最佳线程数应等于CPU内核数。(默认1)
CPU内核数查看:cat /proc/cpuinfo | grep "processor" |wc -l
--tstat TSTAT: 统计模型的线程数,如果未设置,则使用--nthread的值
--cstat CSTAT: 剪切差异cutoff,用于差异剪接的零假设检验的截止值。默认值0.0001,表示0.01%的差异。valid范围: 0 <= cutoff < 1。不适用于配对统计模型
--task {prep,post,both,inte,stat}: 指定要运行的rMATS步骤。默认值both(prep+post)
prep: 预处理BAM并生成.rmats文件
post: 将.rmats文件加载到内存中,检测和统计可变剪接事件并计算P值(如果不是--statoff)
inte(完整性): 检查准备任务记录的BAM文件名是否与当前命令行的BAM文件名匹配
stat:对现有输出文件运行统计测试
--statoff: 跳过统计分析
--paired-stats: 使用配对统计模型
--novelSS: 启用新剪接位点(未注释的剪接位点)的检测,默认是不检测新的剪接位点
--mil MIL: 最小内含子长度,仅影响--novelSS行为。默认值:50
--mel MEL: 最大外显子长度,仅影响--novelSS行为。默认值:500
--allow-clipping: 允许使用软(soft)或硬(hard)剪切的比对方式
--fixed-event-set FIXED_EVENT_SET: 包含fromGTF.[AS].txt文件的目录,用于代替检测一组新事件
二、 从fastq文件开始分析
1、基因组注释文件
测序得到的是几百bp的短read, 相当于是打散的拼图,因此需要参考基因组将测得的序列mapping回去,可以去UCSC下载hg19参考基因组(Downloads-Genome Data-Human-Dec. 2013 (GRCh38/hg38)-Genome sequence files and select annotations (2bit, GTF, GC-content, etc)-Standard genome sequence files and select annotations (2bit, GTF, GC-content, etc)-hg38.Fa.gz)。
注,hg19、GRCH37、 ensembl 75这3种基因组版本应该是见得比较多的国际通用的人类参考基因组,其实他们储存的是同样的fasta序列,只是分别对应着三种国际生物信息学数据库资源收集存储单位,即NCBI,UCSC及ENSEMBL各自发布的基因组信息而已。
然而参考基因组是一部无字天书,要想解读书中的内容,需要额外的注释信息协助。因此还需要去gencode数据库下载基因组注释文件。
附ENSEMBL基因序列文件及数据文件下载:转录组分析数据准备
2、安装STAR并比对参考基因组
一是为了生成基因组索引文件;二是为了比对从fastq文件生成bam文件。
如果直接使用rMATS从fastq输入文件到结果输出文件,中间的bam文件会保留在tmpout文件夹中。同时由于rmats2sashimiplot可视化则需要bam文件作为输入,所以可以尝试先用STAR比对得到bam文件,再用rMATS做差异可变剪切分析。
使用conda命令安装
conda install -c bioconda star
构建基因组索引
STAR --runThreadN 50 --runMode genomeGenerate --genomeDir ./testData/STAR_hg38_index --genomeFastaFiles hg38.fa --sjdbGTFfile gencode.v42.annotation.gtf
#############################
runThreadN ##构建时使用的线程数
runMode genomeGenerate ##让STAR执行基因组索引的生成工作
genomeDir ##构建好的参考基因组存放的位置,最好是单独建立的一个文件夹
genomeFastaFiles ##参考基因组序列文件
sjdbGTFfile ##基因注释文件
此时得到的./testData/STAR_hg38_index文件夹中即包含着STAR binary indices。
接着进行比对,生成bam文件
STAR
--runThreadN 50 --genomeDir ./testData/STAR_hg38_bam --readFilesIn ./testData/fastq/231ESRP.25K.rep-1.R1.fastq ./testData/fastq/231ESRP.25K.rep-1.R2.fastq --outSAMtype BAM SortedByCoordinate --outFileNamePrefix ./testData/STAR_hg38_bam/231ESRP.25K.rep-1 --chimSegmentMin 2 --outFilterMismatchNmax 3 --outSAMstrandField intronMotif --alignEndsType EndToEnd --alignSJDBoverhangMin 6 --alignIntronMax 299999
################################
readFilesIn ##输入文件的位置,对于双末端测序文件,用空格分隔开
outSAMtype 默认输出的是sam文件,我们这里的BAM SortedByCoordinate是让他输出为bam文件,并排序
outFileNamePrefix 表示的是输出文件的位置和前缀(需要将文件位置也写上)
--chimSegmentMin 2 --outFilterMismatchNmax: 比对时允许的最大错配数 --outSAMstrandField intronMotif --alignEndsType EndToEnd --alignSJDBoverhangMin 6 --alignIntronMax: 最长的内含子长度(根据GTF文件计算) ## rMATS软件调用STAR时所用参数多添加的默认参数
若是gz压缩的fastq文件,加上
--readFilesCommand gunzip -c
输出文件位置中,.bam文件即为我们需要的。
3、 认识fastq文件
在illumina的测序文件中,采用双端测序(paired-end),一个样本得到的是seq_1.fastq.gz和seq_2.fastq.gz两个文件,每个文件存放一段测序文件。
在illumina公司测得的序列文件经过处理以fastq文件协议存储为*.fastq格式文件。在fastq文件中每4行存储一个read:
第一行:以@开头,接ReadID和其他信息; 第二行:read测序信息; 第三行:规定必须以“+”开头,后面跟着可选的ID标识符和可选的描述内容,如果“+”后面有内容,该内容必须与第一行“@”后的内容相同; 第四行:每个碱基的质量得分。记分方法是利用ERROR P经过对数和运算分为40个级别分别,与ASCII码的第33号 ! 和第73号 I 对应。用ASCII码表示碱基质量是为了减少文件空间占据和防止移码导致的数据损失。fastq文件预览如下:

4、rMATS运行
(1)从fastq文件开始进行rMATS分析
s1.txt / s2.txt文件整理:
# s1.txt为例
/path/to/1_1.R1.fastq:/path/to/1_1.R2.fastq,/path/to/1_2.R1.fastq:/path/to/1_2.R2.fastq
表明SAMPLE 1组中,有2组配对read(R1,R2)的fastq文件;“:”用于分离配对read,“,”分离replicates。
若数据为fast.gz文件,用法与fastq类似,只需把 .fastq 改为 .fastq.gz 即可。
示例命令行运行
./run_rmats --s1 /path/to/s1.txt --s2 /path/to/s2.txt --gtf /path/to/the.gtf --bi /path/to/STAR_binary_index -t paired --readLength 50 --nthread 4 --od /path/to/output --tmp /path/to/tmp_output
其中–bi 索引文件夹位置即为上述得到的基因组索引文件位置。
若出现报错:
error in mapping sample_0, 231ESRP.25K.rep-1.R1.fastq 231ESRP.25K.rep-1.R2.fastq: 127
error detail: /bin/sh: 1: STAR: not found
首先找到STAR安装位置:
whereis STAR
接着在.bashrc中加上环境变量:
export PATH=$PATH:[STAR安装路径(到STAR所在文件夹即可)]
即可解决。
(2)从bam文件开始进行rMATS分析
较为简单,参考Documentation即可。
rMATS结果解读
一、结果文件解读
rMATS的结果文件是以各个可变剪切事件的分布的,主要有
[AS_Event].MATS.JC.txt(Final output),
[AS_Event].MATS.JCEC.txt(Final output),
fromGTF.[AS_Event].txt(所有已识别的源自GTF和RNA的可变剪接 (AS) 事件),
fromGTF.novelJunction.[AS_Event].txt(选择性剪接 (AS) 事件,仅在考虑RNA后才被识别(而不是单独分析GTF),其不包括具有未注释剪接位点的事件),
fromGTF.novelSpliceSite.[AS_Event].txt(仅包含那些包含未注释拼接位点的事件。仅在使用–novelSS 时相关),
JC.raw.input.[AS_Event].txt(Event counts),
JCEC.raw.input.[AS_Event].txt(Event counts) 。
JC:Junction Counts,把那些会全部比对到 alternatively spliced exon(ASE) 的 reads 进行剔除;
ICEC:Junction Counts+Exon Counts,不仅考虑前者的reads还考虑到全部mapping到剪切exon的reads,Junction counts 是指有mapping到ASE以外部分的 reads count,也就是这些 reads 整体只比对到 ASE 区域的某个部分,而exon counts是指全部mapping到ASE区域的reads数;
为了理解junction count与exon count的具体含义,需要从测序原理来看。测序时选择的readlength是固定的,那么如何从reads结果得到可变剪切事件呢?

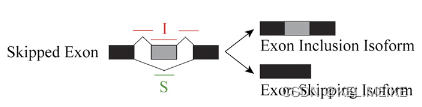
以skipping exon类型为例,上图左侧表示exon inclusion isoform,右侧表示exon skipping isoform,短线表示不同位置的reads,对于黑色的reads来说,是无法区分来自左侧还是右侧的转录本,而绿色和红色的reads则部分或者全部比对到可能skipping的exon上,因此是可以确定来自左侧事件;同样地,紫色reads同时包含来自可能skipping exon的左右连接的片段,因此可以确定是来自右侧事件。
对于exon inclusion isoform来说,绿色的reads数量对应的即是junction counts,而红色的reads数量即为exon counts。
[AS_Event]:可以替换为[SE(skipped exon)、MXE(mutually exclusive exons)、A3SS(alternative 3’ splice site)、A5SS(alternative 5’ splice site)、RI(retained intron)]

如果只是单纯的比较两组样品间可变剪切的差异的话,一般使用 JC.raw.input.[AS_Event].txt的结果就够了。
这几类文件中比较重要的要数[AS_Event].MATS.JC.txt,因为其他文件的信息差不多最终都整合在这个文件里面。
以SE.MATS.JC.txt为例,不同的列包含的信息为:
ID(rMATS事件ID),
GeneID(可变剪切事件所在基因编号),
geneSymbol(可变剪切事件所在基因名称),
chr(可变剪切事件所在染色体),
strand(可变剪切事件所在染色体链);
exonStart_0base,exonEnd,upstreamES,upstreamEE,downstreamES,downstreamEE,6列分别为外显子的位置信息,下图可以很好地解释:

IJC_SAMPLE_1,SJC_SAMPLE_1,IJC_SAMPLE_2,SJC_SAMPLE_2,展示两组样品在inclusion junction(IJC)和skipping junction(SJC)下的count数,重复样本的结果以逗号分隔,可以从下图来理解:
IncFormLen: 可变剪切事件Exon Inclusion Isoform的有效长度,用于归一化,计算:IncFormLen = 2 * (Junction_length - read_length + 1);
SkipFormLen:可变剪切事件Exon Skipping Isoform的有效长度,用于归一化,计算:SkipFormLen = Junction_length - read_length + 1;
其中 Junction_length = 2 * (read_length - anchor), anchor = 8 bp(default)
PValue: 可变剪切事件差异表达显著性p值,仅在使用统计模型时可用;
FDR: 可变剪切事件差异表达显著性FDR值,仅在使用统计模型时可用;
IncLevel1: sample 1中的exon inclusion level(ψ),是Exon Inclusion Isoform在总(Exon Inclusion Isoform + Exon Skipping Isoform)所占比例,计算公式如下:

I 是指mapping到exon inclusion isoform上的reads数,对应结果表格中的IJC_SAMPLE_1
S 是指mapping到exon skipping isoform上的reads数,对应结果表格中的SJC_SAMPLE_1
lI 是指effective length of the exon inclusion isoform,对应结果表格中的IncFormLen,
lS 是指effective length of the exon skipping isoform,对应结果表格中的SkipFormLen。
组中重复样本的结果以逗号分隔。
IncLevel2: sample 2中的exon inclusion level(ψ)。组中重复样本的结果以逗号分隔。
IncLevelDifference: IncLevel1与IncLevel2的差值(average(IncLevel1) - average(IncLevel2)),rMATS采用的是Likelihood-ratio test来比较两组样本可变剪切是否有差异。跟参数**–cstat**设置有关(默认是0.0001),基于marginal distribution(在零假设前提下,近似χ2 distribution),备择假设是|ψi1 −ψi2|> c,因此粗略的理解就是,ψi1和ψi2差异比c越大,P值就越小。
二、结果可视化
rmats2sashimiplot软件专门用来对rMATS分析结果做可视化。
1、软件下载与安装
尝试了github上的软件下载与setup.py安装,没有成功。于是直接在base环境下conda安装:
conda install -c bioconda rmats2sashimiplot
成功。
2、可视化
(1)所有样本可视化
数据来源可以是fastq文件做rmats分析之后得到的中间文件.bam,也可以是STAR比对生成的.bam文件,这里尝试利用fastq文件做rmats分析得到的.bam中间文件:
rmats2sashimiplot --b1 ./fastq_tmpout/2022-11-12-01_05_44_876200_bam1_1/Aligned.sortedByCoord.out.bam,./fastq_tmpout/2022-11-12-01_05_44_876200_bam1_2/Aligned.sortedByCoord.out.bam --b2 ./fastq_tmpout/2022-11-12-01_05_44_876200_bam2_1/Aligned.sortedByCoord.out.bam,./fastq_tmpout/2022-11-12-01_05_44_876200_bam2_2/Aligned.sortedByCoord.out.bam -t SE -e ./fastq_output/SE.MATS.JC.txt --l1 SAMPLE_1 --l2 SAMPLE_2 -o sashimiplot_output
##############################
--b1 ##第一组bam,replicates逗号分隔
--b2 ##第二组bam
-t SE -e ./output/SE.MATS.JC.txt #这里选择SE event来可视化
--l1 --l2 #标记可视化结果中每个组样本名字前缀
-o #输出目录
##other arguments
-c COORDINATE 格式:-c {chromosome}:{strand}:{start}:{end}:{/path/to/gff3}
##在输入文件为.bam时使用,注意annotation文件为.gff3。(可以采用以下命令将gtf转换为gff3: gffread --keep-genes ./some_file.gtf -o ./some_file.gff3)
--exon_s EXON_S ## 外显子缩放比例,Default: 1
--intron_s INTRON_S ## 内含子缩放比例。 Default: 1(如实际长度为100,--introns 5表示显示为100/5=20长度)
查看得到的结果
cd ./sashimiplot_output/Sashimi_plot ##在该文件夹中
(2)分组可视化
首先创建一个grouping.gf文件,包含分组信息:
group1name: 1-3
group2name: 4-6
接着
rmats2sashimiplot --b1 ./fastq_tmpout/2022-11-12-01_05_44_876200_bam1_1/Aligned.sortedByCoord.out.bam,./fastq_tmpout/2022-11-12-01_05_44_876200_bam1_2/Aligned.sortedByCoord.out.bam --b2 ./fastq_tmpout/2022-11-12-01_05_44_876200_bam2_1/Aligned.sortedByCoord.out.bam,./fastq_tmpout/2022-11-12-01_05_44_876200_bam2_2/Aligned.sortedByCoord.out.bam -t SE -e ./fastq_output/SE.MATS.JC.txt --l1 SAMPLE_1 --l2 SAMPLE_2 -o sashimiplot_output_group --group-info grouping.gf
同样地,进入
cd ./sashimiplot_output_group/Sashimi_plot ##在该文件夹中
查看结果。

3、可视化结果解读

RPKM的值表示,将特定gene的计数均匀地分布在坐标上。例如,如果读取长度为50,则在这50长度中,每个单独的坐标将显示1/50的计数值。同时还将该值进行归一化。
注意,sashimiplot中得到的junction count与rMATS中得到的不太一致。
此外,sashimiplot运行时间过长,为了减少计算时间,可以将
-e ./output/SE.MATS.JC.txt
中txt文件先过滤一遍,筛出感兴趣的event进行plot。
参考:
rMATS这款差异可变剪切分析软件的使用体验
可变剪切 rMATS和rmats2sashimiplot
差异剪接分析软件rMATS结果文件解读(v4.1.2)
笔记:rmats绘图结果怎么看
























 3907
3907

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








