一个简单的方法,从 NCBI 批量下载一个物种的所有 RefSeq 基因组序列文件
1. 先说方法
#!/bin/bash
# 下载 Ecoli 基因组索引表格
wget https://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/Escherichia_coli/assembly_summary.txt -O Ecoli_assembly_summary.txt
# 从表格中获取基因组的 FTP 地址
grep -v "^#" Ecoli_assembly_summary.txt | cut -f 20 > Ecoli_assembly_ftp.txt
# 下载
for url in $(cat Ecoli_assembly_ftp.txt); do
cd ~/Download/Ecoli_Genomes
file_prefix=$(basename $url)
accession_name=$(echo "$url" | grep -oE "GCF_[0-9]+\.[0-9]+")
mkdir ${accession_name}
cd ${accession_name}
wget ${url}/${file_prefix}_genomic.fna.gz -O ./${accession_name}_genomic.fna.gz
wget ${url}/${file_prefix}_genomic.gff.gz -O ./${accession_name}_genomic.gff.gz
wget ${url}/${file_prefix}_protein.faa.gz -O ./${accession_name}_protein.faa.gz
for zip_file in $( ls ); do
gunzip ${zip_file}
done
done- 将以上文件保存为
download.sh,随后运行
nohup bash download.sh 2> download.err &- 实现效果

2. 实现逻辑
- 通常从 NCBI 上下载基因组,是在 Genome 中搜索目标物种,然后选择需要下载的项目,点 Download 下载
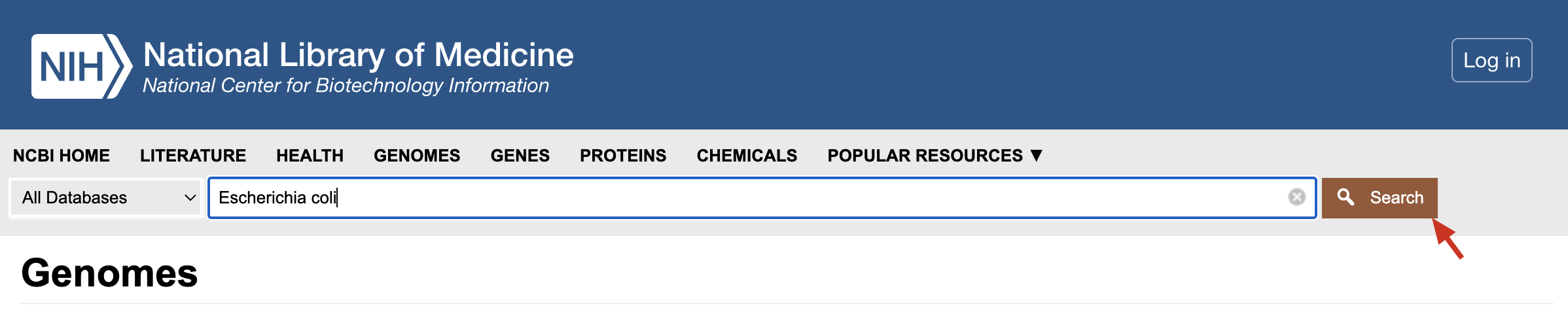
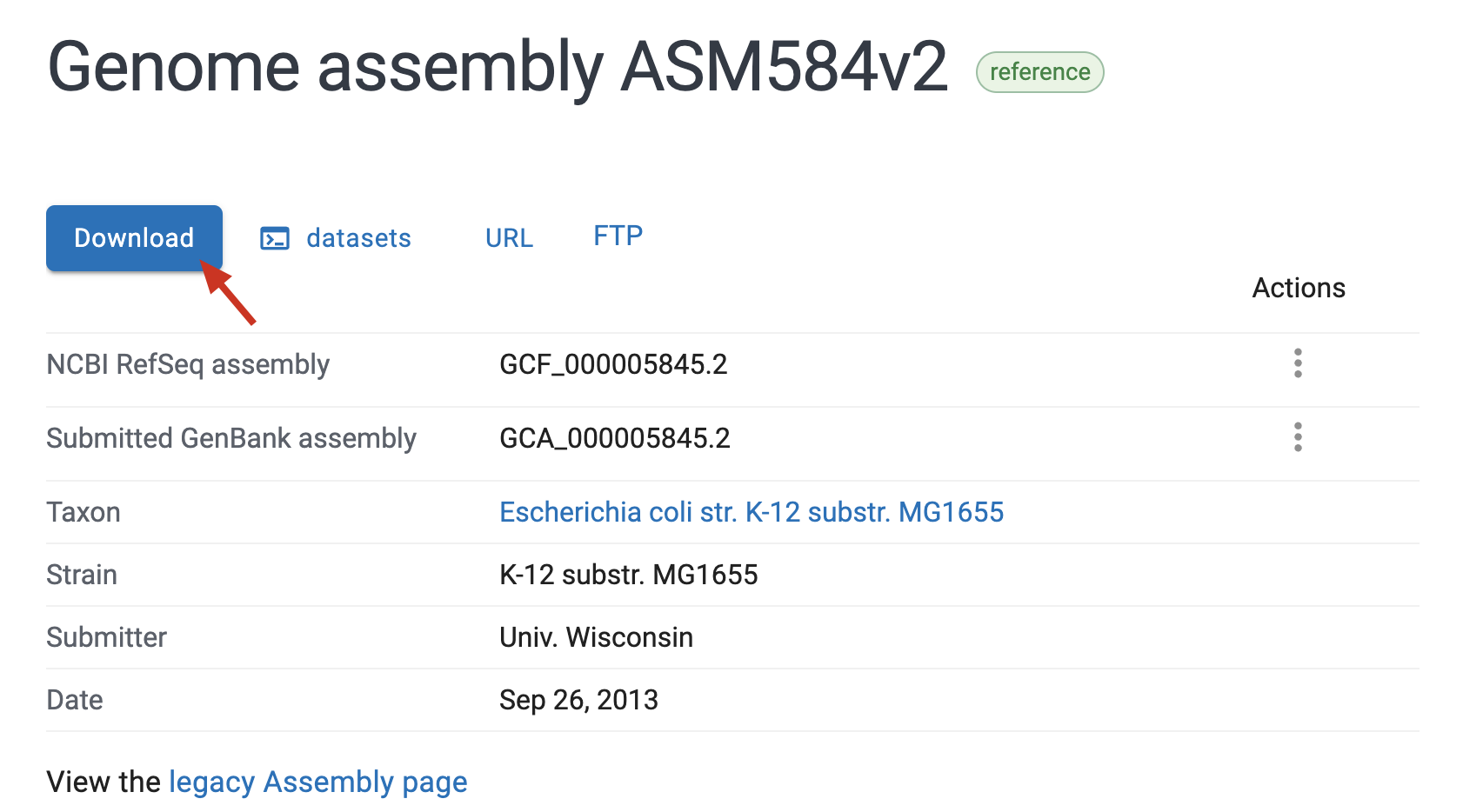
- 但是当我们需要下载大量基因组做群体分析的时候,成千上万的基因组一个一个点击下载显然是不科学的
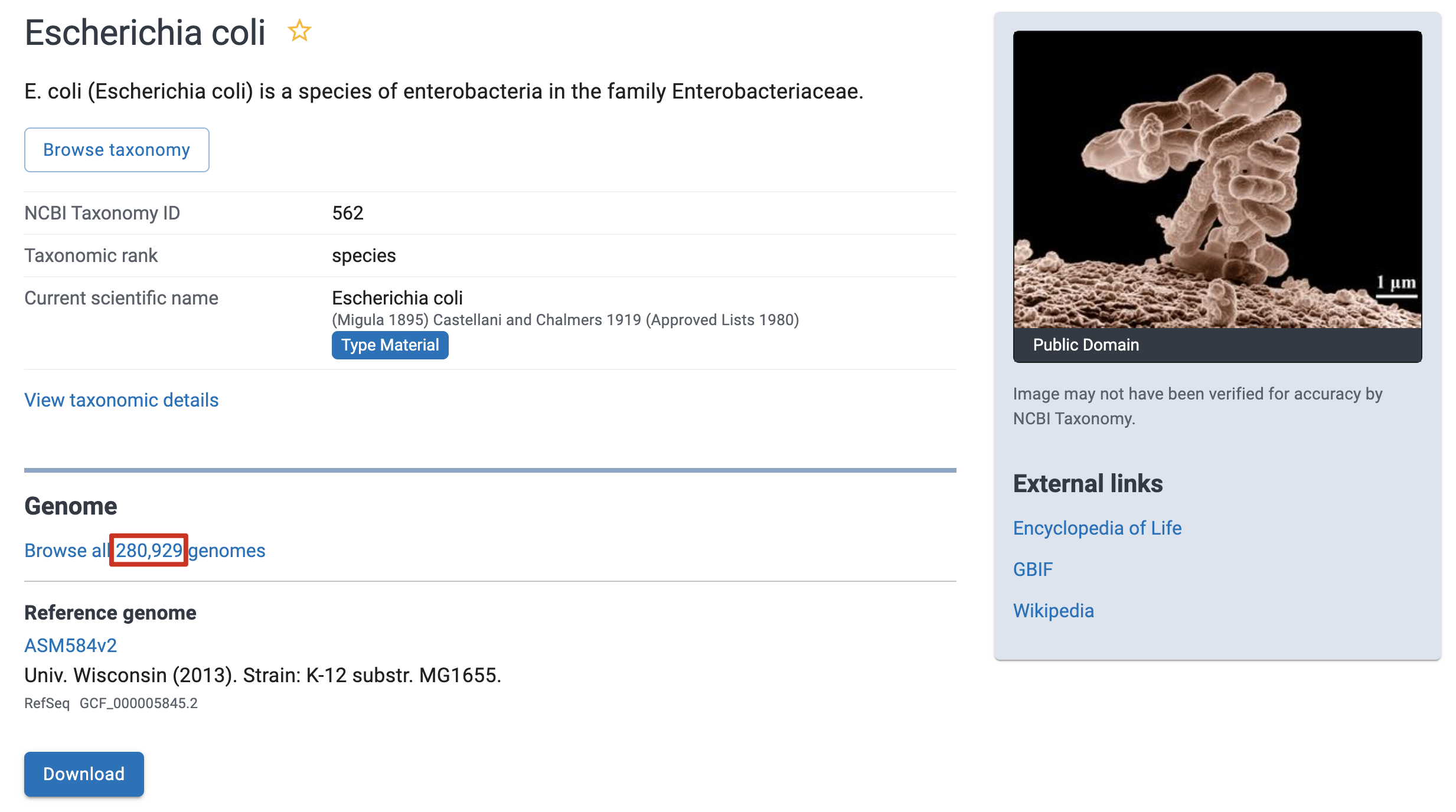
- 搜索到网上有一些现成的工具,如一些 Python 写的脚本工具 ncbi-genome_download,但是不知道为什么我下载不了

- 后来想想,估计工具都是从 NCBI 的 FTP 网站下载的,不如简单一点自己从 NCBI 的 FTP 站点去下载
2.1. 从 FTP 网站下载索引文件
- NCBI FTP 基因组存储站点:Index of /genomes/refseq
 https://ftp.ncbi.nlm.nih.gov/genomes/refseq/
https://ftp.ncbi.nlm.nih.gov/genomes/refseq/ - 在 FTP 站点中会存在一个汇总文件,这个文件中记载了所有基因组的 FTP 下载地址
# 所有物种的索引
https://ftp.ncbi.nlm.nih.gov/genomes/refseq/assembly_summary_refseq.txt
# 细菌届所有物种的索引
https://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/assembly_summary.txt
# 大肠杆菌所有菌种的索引
https://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/Escherichia_coli/assembly_summary.txt- 以大肠杆菌为例,下载大肠杆菌所有 RefSeq 基因组的索引文件
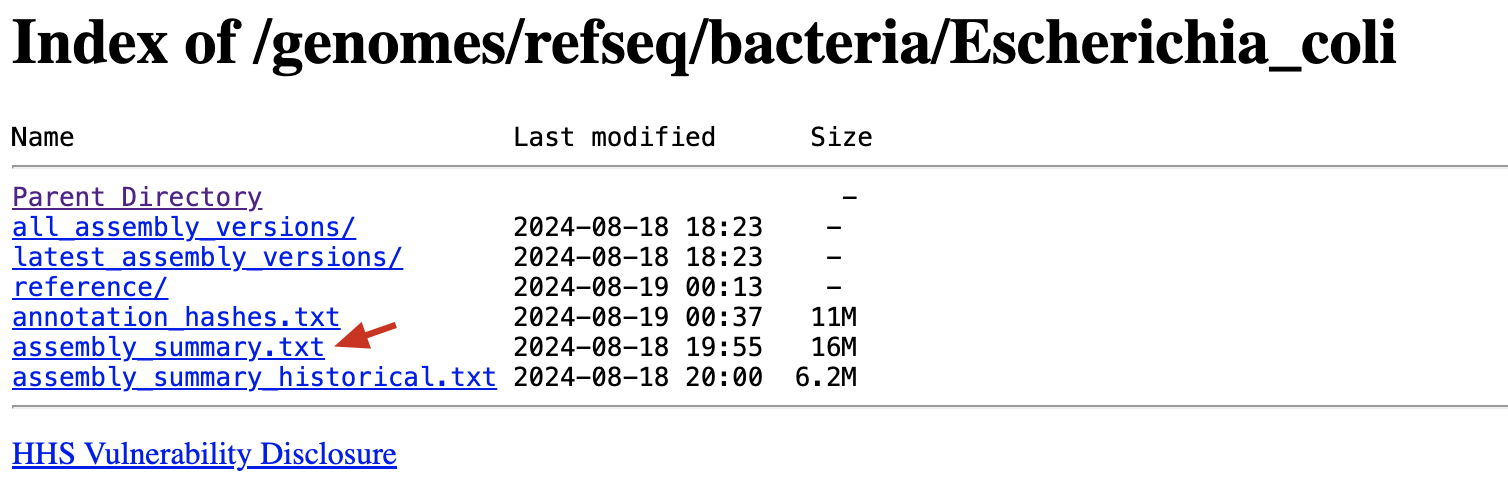
wget https://ftp.ncbi.nlm.nih.gov/genomes/refseq/bacteria/Escherichia_coli/assembly_summary.txt -O Ecoli_assembly_summary.txt2.2. 获取索引文件中的 FTP 地址
- 这个 txt 文件是以制表符分隔的文本文件,其中第 20 列是所有基因组的 FTP 地址
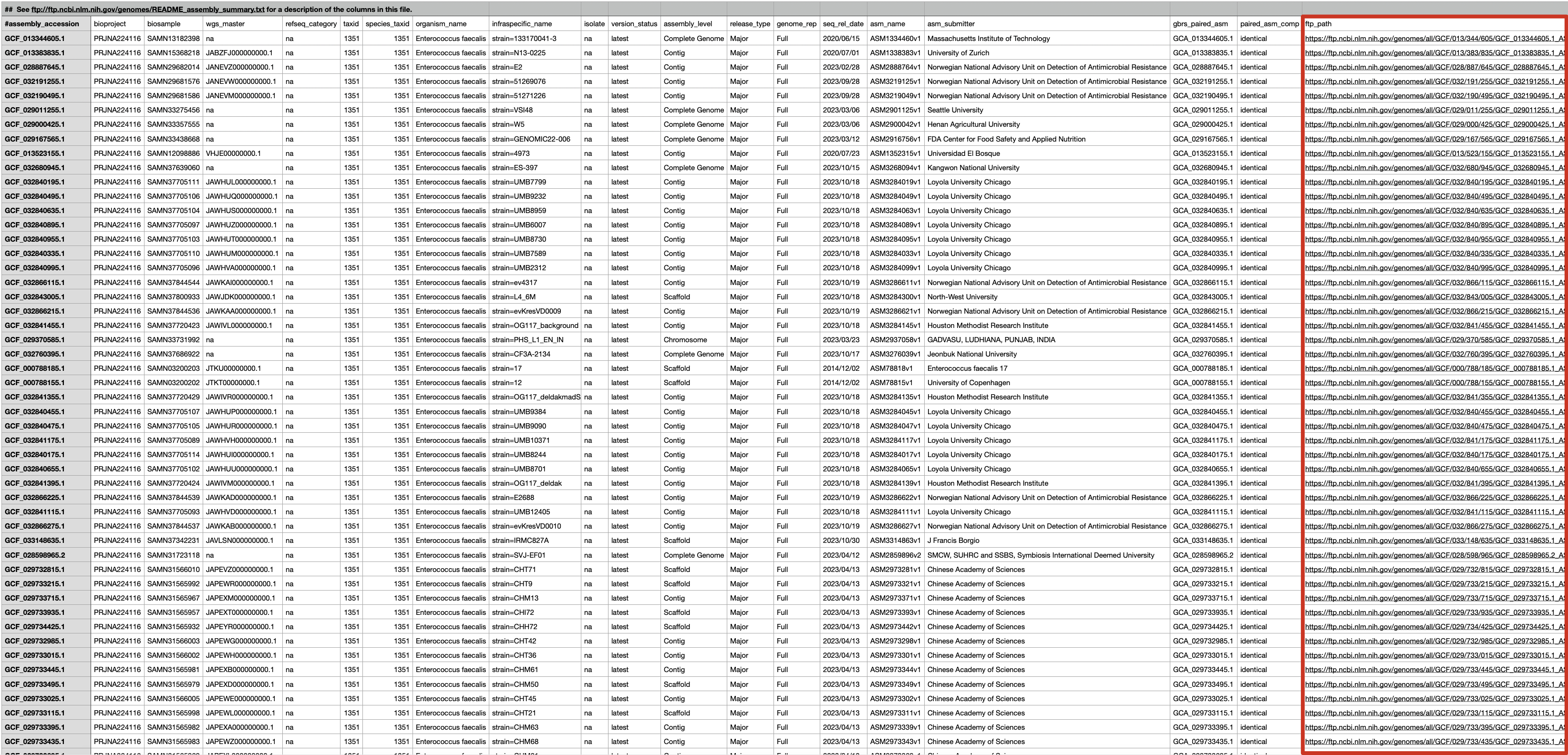
- 将这些 FTP 地址提取到一个新的文本文件
grep -v "^#" Ecoli_assembly_summary.txt | cut -f 20 > Ecoli_assembly_ftp.txt2.3. 从 FTP 地址中获取完整文件的下载地址
- 表格中的 FTP 地址打开后可以发现是一个存储了基因组组装序列等文件的目录
- 里面的文件是以地址的基名为前缀的
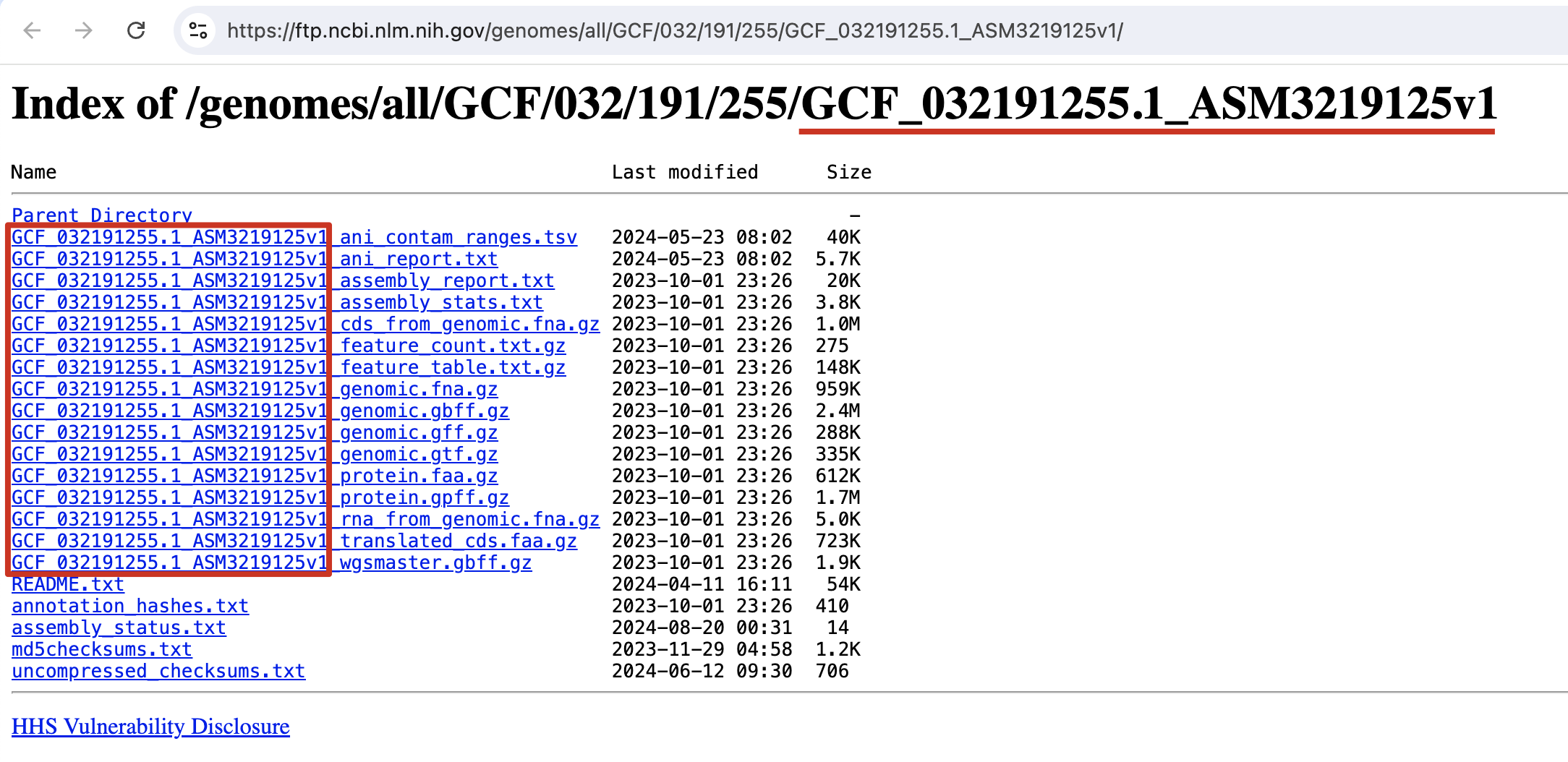
- 只需要在这个基名后面加上所需要数据的后缀即可获得对应文件的下载地址
# 基因组序列
**_genomic.fna.gz
# 基因组蛋白序列
**_protein.faa.gz
# 基因结构注释
**_genomic.gff.gz - 补全文件下载链接
for url in $(cat Ecoli_assembly_ftp.txt); do
file_prefix=$(basename $url)
echo "${url}/${file_prefix}_genomic.fna.gz" >> Ecoli_genome_ftp.txt
echo "${url}/${file_prefix}_protein.faa.gz" >> Ecoli_protein_ftp.txt
echo "${url}/${file_prefix}_genomic.gff.gz" >> Ecoli_annotation_ftp.txt
done2.4. 下载对应文件
- 以下载蛋白序列为例,下载大肠杆菌所有 RefSeq 基因组的蛋白序列,共计 3572 个
for link in $(cat Ecoli_protein_ftp.txt); do
wget ${link}
done






















 8623
8623

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








