10x Genomics Visium 空间转录组是基于完整的组织切片而进行的基因表达无偏检测。该技术通过对置于载玻片上特定捕获区域内的组织切片进行H&E染色和拍照以保留样本的组织形态学信息,然后对样本进行透化处理,将RNA释 放并与捕获区域内的寡核苷酸探针进行杂交,使得不同位点的样本基因信息被SpatialBarcode标记。该技术将基因表达活跃度信息与组织形态学信息结合起来,呈现了组织和基因表达复杂性的新视图。2024年6月,10X公司第一篇 Visium HD的文章在bioRxiv预印,介绍了分辨率可达2 μm, 能实现单细胞分辨率的全转录组空间分析技术Visium HD,以其单细胞分辨率、连续的组织覆盖、高质量的空间数据特性引起了大家的注意。
为了学习Visium HD数据分析流程,我们准备使用10x官方公布的人结直肠癌样本数据,从原始数据开始,一步步实现文章中的分析结果。我们下载了几个CRC样本测序原始数据、CytAssist导出的组织图像、组织切片的H&E高清图像,公开数据下载地址:https://www.10xgenomics.com/products/visium-hd-spatial-gene-expression/dataset-human-crc

Visium HD技术原理
Visium HD工作流程类似于v2版本CytAssist支持的Visium空间基因表达工作流程。这个流程将标准的组织学过程与简单的分子生物学方案相结合,在获取组织切片的H&E或IF图像的同时,获取单细胞尺度的全转录组空间基因表达读数。Visium HD已经支持人和小鼠的FFPE、FF样本,样本准备要求与Visium CytAssist(V2)相同。为获得理想的测序数据结果,应确保组织样本的细胞核形态正常、有高质量的RNA以及在实验过程中不会出现脱片等异常情况。Visium HD的实验流程与Visium CytAssist(V2)相同,包括以下主要实验步骤:
(1)样本准备与成像:将FFPE样本切片贴于载玻片有效转片区域内,进行脱蜡、HE染色与图像扫描、脱色和解交联,然后立即进行探针杂交;
(2)探针过夜杂交并清洗后,进行探针连接;
(3)在CytAssist转片仪上完成转片;
(4)将Visium HD玻片置于PCR仪上进行探针延伸;
(5)洗脱并收回探针进行预扩增与文库构建;
(6)高通量测序与数据分析。

Visium HD空间基因表达载玻片包含两个6.5 x 6.5 mm 捕获区域,其中寡核苷酸连续排列在数百万个2 x 2μm正方形中,且芯片无组织间隙,实现单细胞分辨率空间转录组测序的同时达到了全组织覆盖度。数据分析可以以2 μm为最小分辨率,也可以展示多个Bin的分辨率,我们以8x8μm Bin为最小分辨率进行后续分析。

Space Ranger软件安装
软件及参考基因组下载地址:https://www.10xgenomics.com/support/software/space-ranger/downloads

Visium HD图像手动对齐导出对齐参数
Space Ranger虽然有自动图像检测算法来确定基准标记的位置并识别组织边界,但是经常结果不是很理想,所以通常需要Loupe Browser以交互方式将图像与载玻片的基准标记位置对齐,并导出手动对齐参数以在Space Ranger运行中使用。

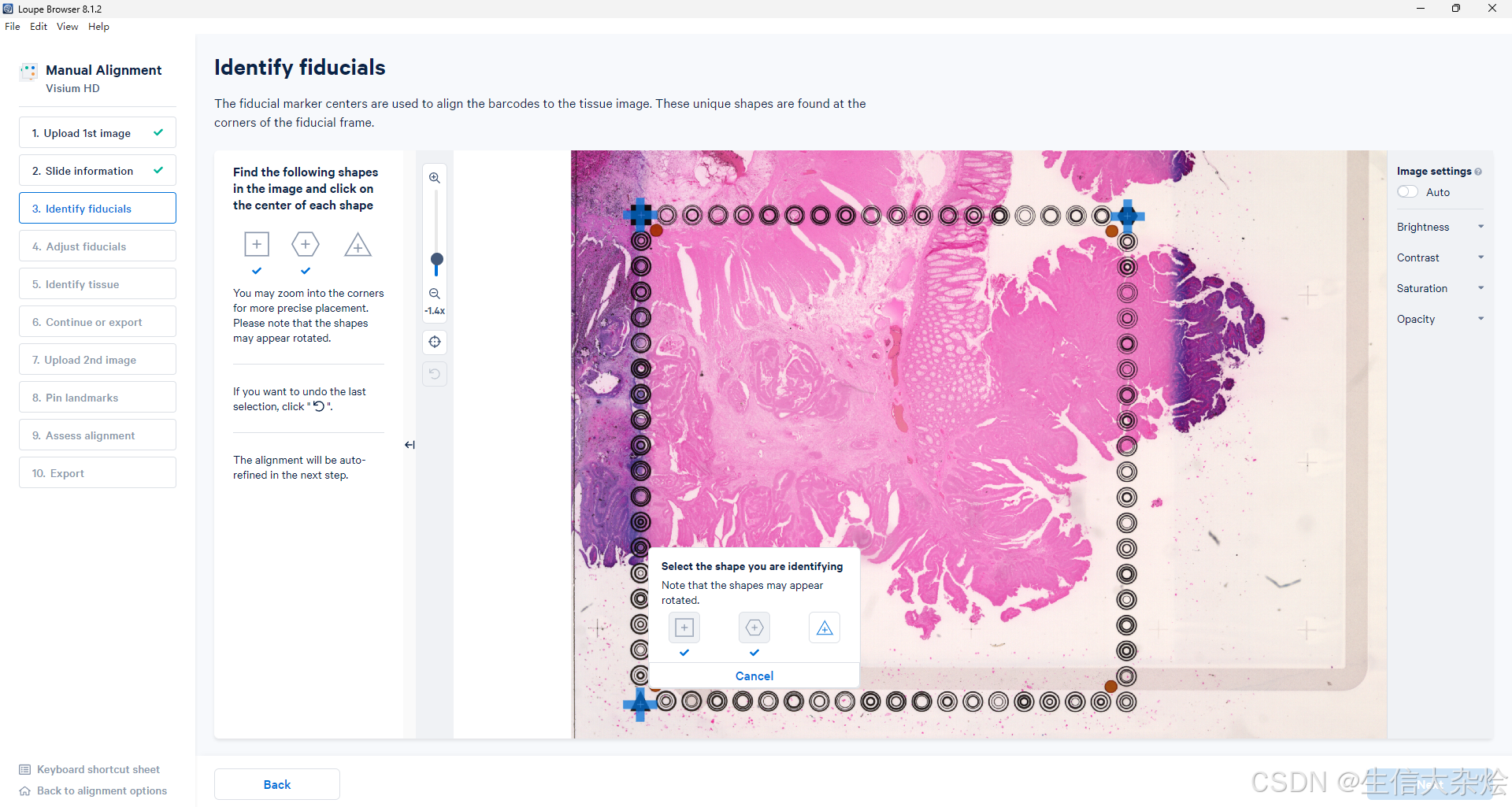
上传Visium HD CytAssist 图像

填入HD芯片信息,直接导入CytAssist的图像后会自动识别芯片编号和捕获区域

按照提示,校准捕获区域
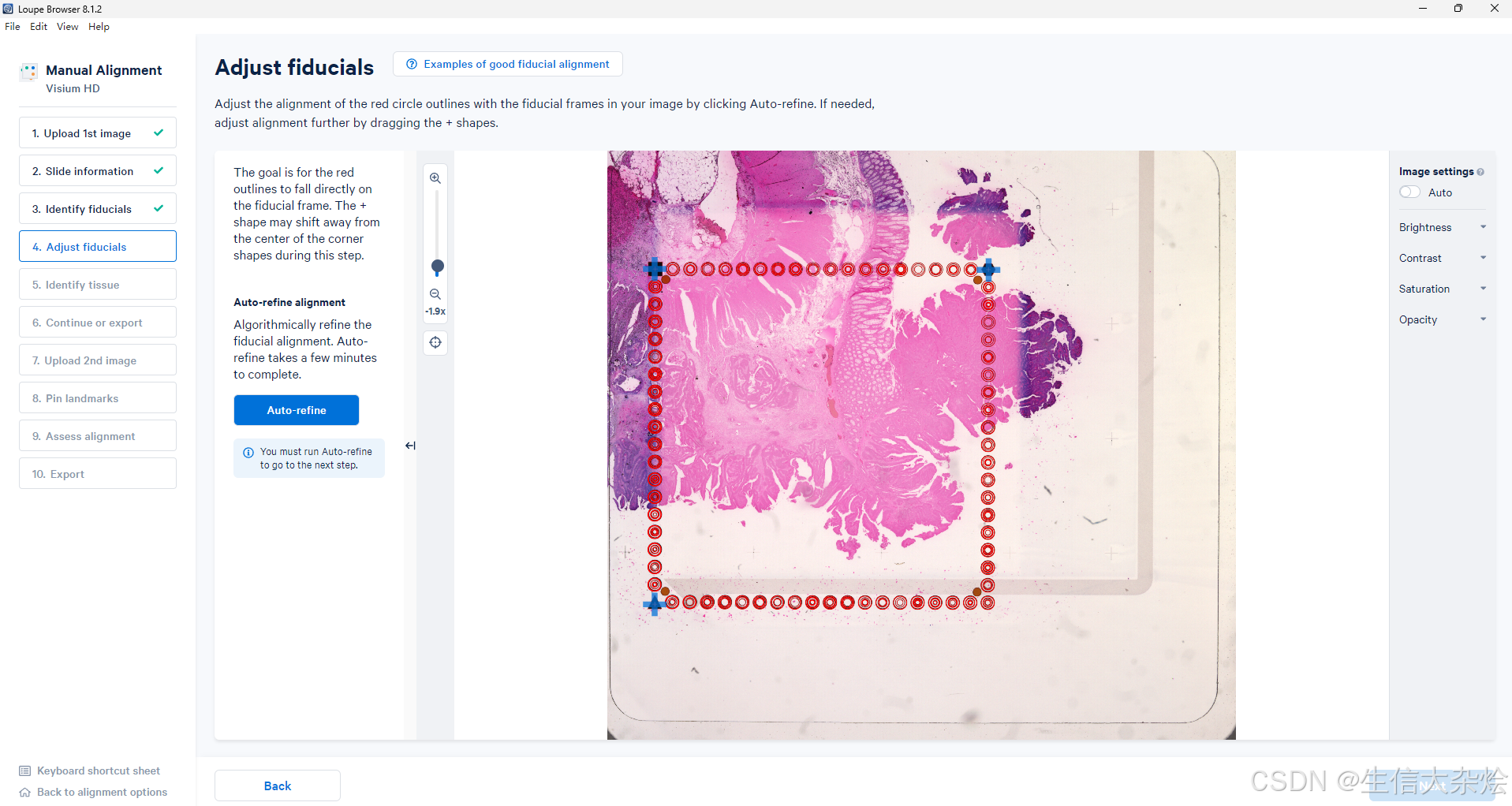

上传组织H&E高清图像

左右两侧选定对应锚点,一般3~5个左右即可

导出对齐参数json文件,该文件后续Space Ranger分析时需要传入


Space Ranger分析
spaceranger count \
--id=P1_CRC \
--transcriptome=/database/refdata-gex-mm10-2020-A \
--fastqs=/home/data/Visium_HD_Human_Colon_Cancer/fastq \
--sample=P1_CRC \
--image=/home/data/Visium_HD_Human_Colon_Cancer/images/Visium_HD_Human_Colon_Cancer_image.tif \
--slide=H1-VM2JXXK \
--area=A1 \
--localcores=32 \
--localmem=64 \
--create-bam false \
--probe-set=/home/software/spaceranger-3.0.0/probe_sets/Visium_Mouse_Transcriptome_Probe_Set_v2.0_mm10-2020-A.csv \
--cytaimage=/home/data/Visium_HD_Human_Colon_Cancer/images/Visium_HD_Human_Colon_Cancer_tissue_image.btf \
--loupe-alignment=/home/data/H1-VM2JXXK-A1-fiducials-image-registration.jsonSpace Ranger完成后生成结果文件夹

Space Ranger输出文件结构
binned_outputs文件夹内默认生成了2um,8um,16um bin的结果,Spaceranger v3.1版本后可以自定义生成2-100um的偶数bin,同时也会生成相应的cloupe文件,之前版本只会生成8um bin的cloupe文件。我们使用10x文章中推荐的8x8um bin进行后续分析。

web_summary结果


微信公众号:生信大杂烩
























 633
633

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








