#map/ped文件#
-
map文件
记录变异位置信息
第一列 染色体编号
第二列 SNP名称
第三列 遗传距离
第四列 snp物理位置
-
ped文件

第一列 家系编号 第二列 个体编号
第三列 父本 第四列 母本
第五列 性别 第六列 表现型
第七列往后 每个SNP位点基因型
 第七列第八列是第一个基因型
第七列第八列是第一个基因型
#二进制格式 bed / bim / fam #
-
bed文件
SNP数据,是二进制格式,不能由Notepad++等文本编辑器打开
-
bim文件
SNP位置信息

第一列 染色体编号 第二列 SNP名字
第三列 遗传距离 第四列 物理距离
第五列 次要等位基因(minor allel) 第六列 主要等位基因(major allel)
-
fam文件
家系表型信息

第一列 FID 第二列 IID 第三列 父本
第四列 母本 第五列 性别 第六列 表型
# 转置格式tped / tfam 文件#
-
tped文件
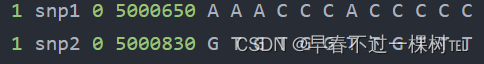
前四行 = map文件
后面代表所有样本在该snp位点基因型信息
-
tfam文件

= ped文件1~6列
#plink文件格式相互转换#
-
将map/ped文件转化为二进制文件
plink --file GENO --make-bed --out geno-
将tped/tfam文件转化为二进制文件
file变为tfile
结果:
![]()
-
将ped/map文件互相转化tped/tfam
# map/ped转tped/tfam #
plink --file GENO --recode --out GENO
# tped/tfam转map/ped #
plink --tfile GENO --recode --transpose --out GENO
-
将二进制文件转化为map/ped
plink --bfile geno --recode --out GENO-
将二进制文件转化为tped/tfam
recode后加transpose
-
二进制文件转化为vcf
plink --bfile geno --recode vcf --out geno --chr-set 27-
二进制文件转化为bgen
plink --bfile geno --recode bgen --out geno-
vcf文件转化成map/ped
plink --vcf geno.vcf --recode --out geno























 1143
1143











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








