文件准备
需要一个数据框
第一列为snp名称
第二列为snp所在的染色体
第三列为snp位点
install.packages("CMplot")
library(CMplot)
df <- read.csv("out_filter.delta.snps_cmplot",header = F,sep = "\t")
head(df)
CMplot(df, plot.type="d", bin.size=1e6, col=c("darkgreen", "yellow", "red"),
file="pdf", dpi=400, file.output=TRUE, verbose=TRUE)
# plot.type: 绘图类型,此处为 "d",表示绘制距离矩阵图
# bin.size: 绘图时基因组距离矩阵的间隔大小,此处为 1e6,表示每隔 1e6 个碱基绘制一个格子
# col: 绘图时不同距离区间的颜色,此处为一个颜色向量,分别表示低、中、高距离区间的颜色
# file: 输出文件的格式,此处为 "pdf",表示输出为 PDF 格式
# dpi: 输出文件的 DPI(每英寸点数),此处为 400
# file.output: 是否输出文件,此处为 TRUE,表示输出文件
# verbose: 是否显示运行信息,此处为 TRUE,表示显示详细信息
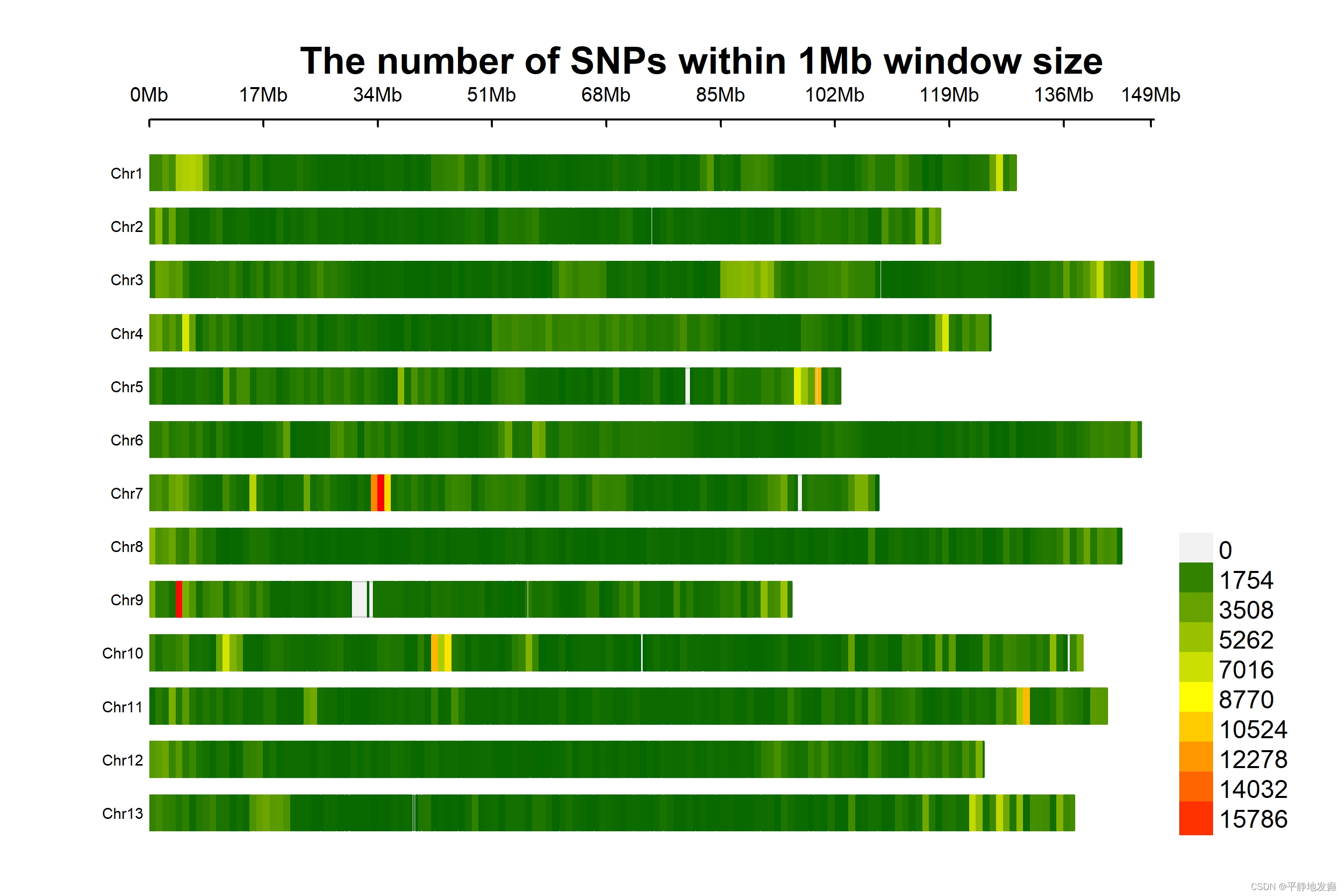








 该篇文章介绍了如何使用R语言中的CMplot包对SNP数据进行处理,通过指定参数如plot.type=d、bin.size=1e6和col设置颜色代码,生成SNP位点在不同染色体上的距离矩阵图,并将结果保存为PDF格式,输出文件选项被设为True。
该篇文章介绍了如何使用R语言中的CMplot包对SNP数据进行处理,通过指定参数如plot.type=d、bin.size=1e6和col设置颜色代码,生成SNP位点在不同染色体上的距离矩阵图,并将结果保存为PDF格式,输出文件选项被设为True。
















 9520
9520

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








