1 基本信息
博士退学前,做完斑马鱼的Phylogenomics分析,系统进化树冲突、基因流、ILS和种群历史动态等分析了之后,需要看一下Danio属物种的地理分布,希望能跟Phylogenomics分析结果有所印证。所以,自己在网上搜索生物分布数据库,找到了很多有物种分布数据的数据库(可惜当时没有好好整理成文档),要不就是不全,要不就是不容易整理(还是编程能力不强)。最后发现了GBIF(Global Biodiversity Information Facility,https://www.gbif.org/)数据库,一个开源的生物多样性数据库,在R中有API接口rgbif包,可以物种拉丁名等形式搜索和下载物种分布数据和绘制分布地图,果断就用起来了。
新版rgbif进行了重大更改,2020年记录的代码都不能使用了。绘制地图功能也放在了gistr包。因此,重新学习了rgbif包,在这里做一下记录。但是用R绘制的地图太简单,最后还是找物海的同学用ArcGIS画的,在国际粮农组织下载的水文数据。本文简单的记录rgbif包的功能以及绘制简单物种分布地图。
2 分析流程
2.1 设置工作路径并调用R包
# 设置工作路径
#knitr::opts_knit$set(root.dir="D:\\EnvStat")# 使用Rmarkdown进行程序运行
setwd("D:\\EnvStat") # 设置工作目录
Sys.setlocale('LC_ALL','C') # Rmarkdown全局设置
# 调用R包,不是每个包都用到,但是需要了解的R包
install.packages("ggmap")
library(ggmap) # 可从谷歌下载地图,但是现在每天只能使用一次
library(maps)
help(package="maps") # 查看包信息
BiocManager::install("rgbif")
library(rgbif)
help(package="rgbif")
install.packages("gistr") # rgbif旧版中的gbifmap()迁移到了此包
library(gistr)
#install.packages("ropensci")
#library(plyr)
#library(XML)
#library(httr)
library(ggplot2)
library(rgdal) #提供readOGR()函数,可以读取shp,JSON等地图文件
library(dplyr)
library(RColorBrewer)
# library(ggpubr) # ggarrange(),可以合并图
install.packages("raster")
library(raster)2.2 rgbif下载GBIF数据库数据
需要注意的物种名会有更改,鉴种结果可能存在争议,采样点物种可能存在多次鉴种结果不同的情况,一般以最后一次鉴种结果为准,且GBIF数据库中数据也不一定完全正确,还是要多查询几个数据库,还要人工对数据进行校正。
# 2.2.1 使用物种拉丁名下载数据,使用属名下载属中物种分布数据
?rgbif # 有很多功能,如根据国家、地区和经纬度范围下载物种数据等等这里就不展开了,可以查看帮助文件
## 下载Danio物种,rgbif使用的是3.6.0版本,经历了大的改版,很多之前的函数都不能用了。可以??旧版函数,帮助文章会显示对应的新版函数
## keys <- taxonsearch(scientificname = "Danio") # 这是旧版的函数,3.60版的rgbif中已经没有了,改成了occ_search()
keys <- occ_search(scientificName = "Danio")
keys$data[1,]# 显示第一行数据,包括key,分类单元数据、十进制经纬度和参考链接等信息
dim(keys$data)
## 获取Danio物种分布数据以及分类单元数据后,可以根据经纬度绘制点描法地图
Danio=keys$data[c(1:4,9,18,28:33,47)] # 提取需要的信息
Danio
##
## 保存结果到本地,数据中包含逗号,不要保存为csv格式。
write.table(Danio, file = "Danio_distribution.xlsx",quote = F, sep = "\t",row.names = F)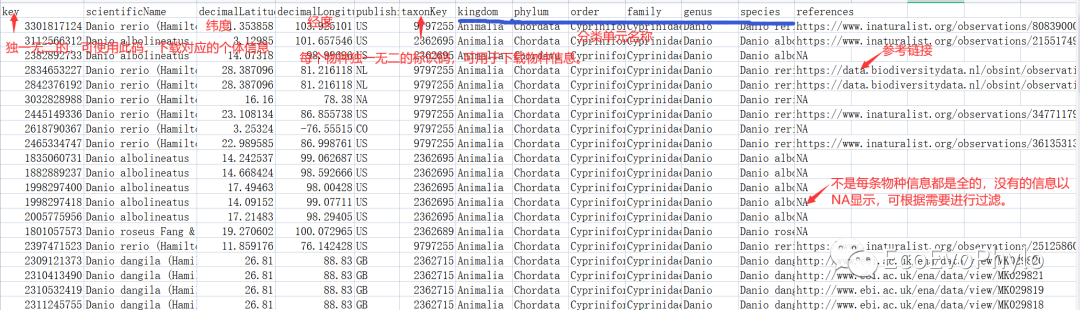
图1|根据拉丁名下载的物种分布数据,Danio_distribution.xlsx。
2.3 绘制物种分布地图
从GBIF获取物种分布的经纬度数据之后,可以使用ggplot2将物种点投影到地图上。maps包和ggmap包可用于在线获取地图数据。rgdal包的readOGR函数可将shp(国家地理信息统计局和世界地理信息统计单位可以下载)和Json格式地图信息,读入R中,在逐步转换为ggplot()可以使用的数据框格式。
# 2.3.1 去除无经纬度信息的物种,|不能用于字符型变量,或者会报错。
Danio <- Danio[-c(which(is.na(Danio$decimalLatitude | Danio$decimalLongitude)),is.na(Danio$species)),]
Danio
dim(Danio)
# 2.3.2 获取地图信息
## 可以使用maps包和ggmap在线获取地图信息
print((data(package='maps'))) # 查看R包中的数据集
#data(world.cities)
#head(world.cities)
world_map <- map_data("world") # maps包map_data()中获取世界地图数据
world_map
## 读入shp地图文件,推荐看《R数据可视化之美:专业图表绘制指南(增强版)》第11章
#dataProjected <- readOGR("Virtual_Map1.shp")
#dataProjected # 列表形式数据
#dataProjected@data$id <- rownames(dataProjected@data)
#watershedPoints <- fortify(dataProjected) # fortify()之后可能会被放弃,推荐使用broom包
#watershedPoints # 形成数据框形式
#df_Map <- full_join(watershedPoints, dataProjected@data, by = "id")
#df_Map
# 2.3.3 绘制点描法地图
## (a):地图 + 物种分布散点,还可以添加文本(物种和地域标签)
p1 <- ggplot()+
geom_polygon(data = world_map, aes(x = long, y = lat,group = group),
fill='white',colour="black",size=0.25)+
geom_point(data = Danio,aes(x = decimalLongitude, y = decimalLatitude,fill = species),
alpha = 0.5,shape = 21,size = 3)
#+geom_text(data=Danio,aes(x=decimalLongitude, y=decimalLatitude, label=species),vjust=1.5,colour="black",size=3) # 添加物种标签
#+geom_label(data = world_map,aes(x = long, y = lat, label = region),fill='orange',colour="black",size=3) # 给地图安区域添加地域标签
p1
## (b):修改主题细节
world_map$
p2 <- ggplot()+
geom_polygon(data = world_map, aes(x = long, y = lat, group = group),fill='white',colour="grey",size=0.25)+
geom_point(data = Danio,aes(x = decimalLongitude, y = decimalLatitude,fill = species),
alpha = 0.5,shape = 21,size = 3)+theme_bw()
p2注:当需要使用带有国界线地图数据的时候,使用的地图数据(特别是国外的)一定要小心。利用下载的地理信息数据编制地图的,应当严格执行《地图管理条例》有关规定;编制的地图如需向社会公开的,还应当依法履行地图审核程序。
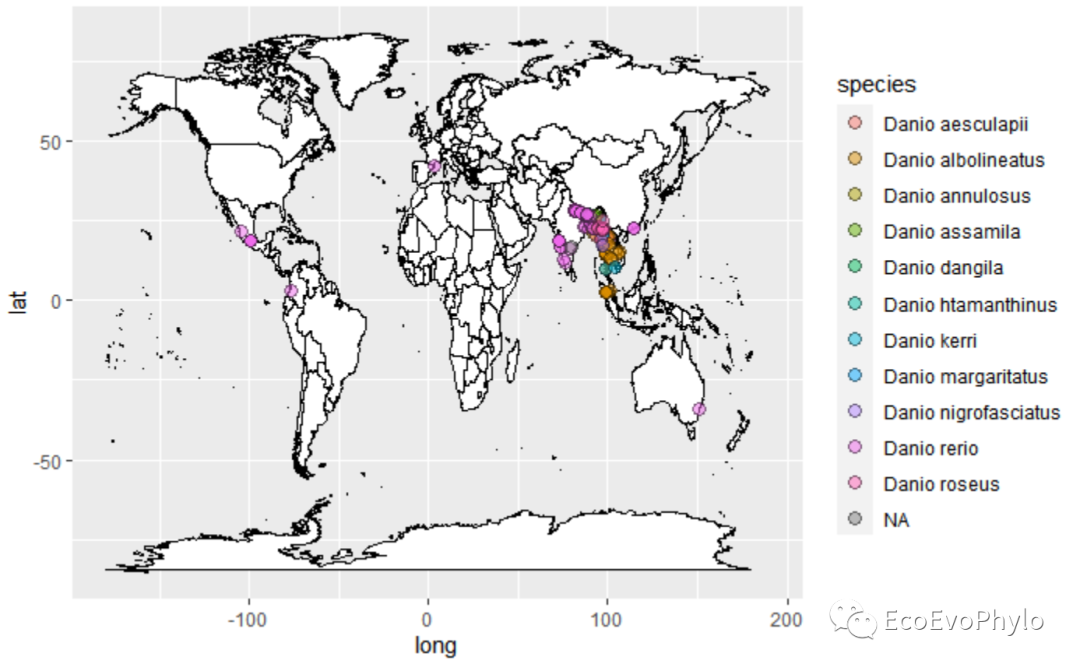
图2|Danio属物种分布图,p1。这是在世界地图上添加的物种数据,也可以直接根据物种的经纬度确定限时地图的显示范围。
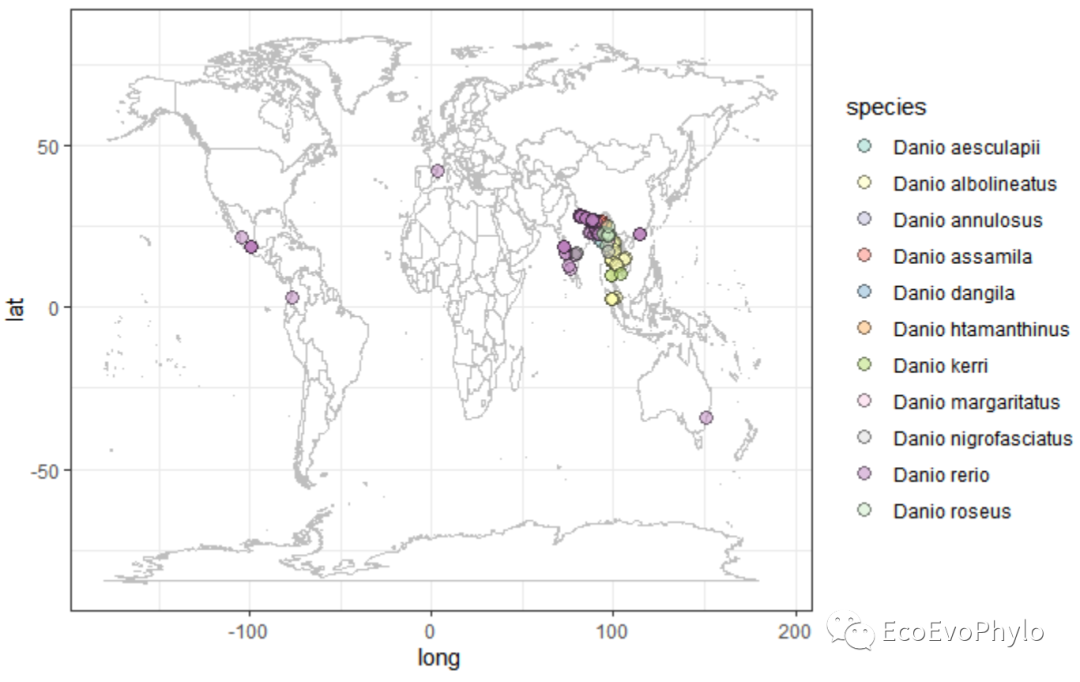
图3|Danio属物种分布图,p2。更改物种点颜色和主题细节。还有很多可以修改,但是其实还是推荐用ArcGIS画,要求高的话,最好找专业的人画。
2.4 rgbif其他功能
rgbif作为GBIF数据库的API接口,其实功能很强大,还有很多函数。推荐阅读参考资料。下面做一些简单功能介绍。
# 使taxonKey下载数据,以Danio htamanthinus为例,它的taxonKey=9103430
Dh <- occ_data(taxonKey = 9103430) # 最高只能下载 100,000条数据
Dh
# 对下载的数据根据datasetKey进行汇总统计
Dh_Counts <- Dh$data %>% count(datasetKey, sort=TRUE)
Dh_Counts
occ_get(key=c(855998194, 240713150)) # 下载指定条目数据,返回结果是一个列表
# 获取个体或数据集信息的参考信息
## 使用key或UID号获取单条数据的参考信息
gbif_citation(x=1265576727)
gbif_citation(x='d52d01f6-7472-4dbf-9ea8-a33c343255ac')
# occ_count()计数GBIF中某个类型数据包含的数据个数
occ_count(georeferenced=TRUE) # GBIF中所有具有经纬度数据的个体数量
occ_count(taxonKey=9103430, georeferenced=TRUE) # GBIF中具有经纬数据Danio htamanthinus的数据条数
occ_count(datasetKey='e707e6da-e143-445d-b41d-529c4a777e8b', basisOfRecord='OBSERVATION') # 特定数据集中的数据量
occ_count(datasetKey='e707e6da-e143-445d-b41d-529c4a777e8b', year=2012) # 特定数据集中2012年的数据量
# 获取分类单元值
taxrank()
# name_lookup()根据 scientific和vernacular名、物种描述、分布以及分类单元等信息搜索数据库,返回结果按相关性排序
?name_lookup()
z <- name_lookup(query='Danio', rank="genus") # 搜索Danio属数据
z
# name_usage()可使用不同的名称搜索数据
?name_usage
name_usage()
name_usage(key=3119195, language="ENGLISH",rank = "GENUS")
# raster包的map_fetch()可视化数据
?map_fetch
x <- map_fetch(taxonKey = 2480498, year = 2000:2017)
plot(x)
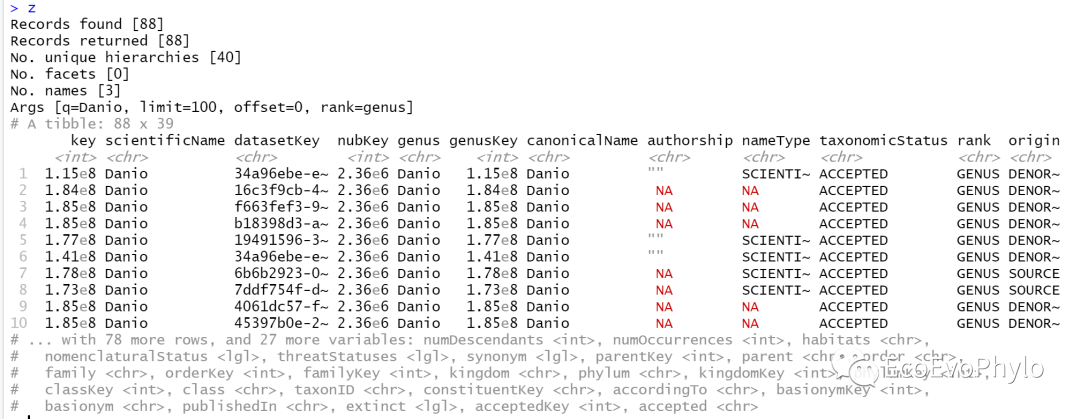
图4|name_lookup搜索Danio属物种数据结果,z。

图5|name_usage搜索结果。
rgbif还有很多函数没有介绍,有时间再写一篇推文进行介绍。公众号后台回复"R数据可视化之美"获取书籍中的全部R代码和数据。

扫描上方二维码,关注EcoEvoPhylo数据分析小店,购买数据分析书籍和服务。
参考文献
《R数据可视化之美:专业图表绘制指南(增强版)》
https://www.sogou.com/link?url=hedJjaC291NPJpyiesH6HTlDOV9YK_p3sGYM_rVBLGM.
https://github.com/ropensci/rgbif
http://tools.gbif.org/
https://docs.ropensci.org/rgbif/articles/rgbif.html
https://docs.ropensci.org/rgbif/reference/index.html
https://docs.ropensci.org/rgbif/articles/index.html
https://cran.r-project.org/web/packages/rgbif/rgbif.pdf
推荐阅读
R统计-PCA/PCoA/db-RDA/NMDS/CA/CCA/DCA等排序分析教程
R统计绘图-单、双、三因素重复测量方差分析[Translation]
开放转载
公众号原创文章开放转载,在文末留言告知即可

EcoEvoPhylo :主要分享微生物生态和phylogenomics的数据分析教程。扫描上方二维码,即可关注EcoEvoPhylo。让我们大家一起学习,互相交流,共同进步。


























 3076
3076











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










