用R语言绘制Cell文章中的散点图(火山图)。

读入差异基因分析的文件,并加载ggplot2。
setwd("E:/生物信息学")library(ggplot2)A <- read.csv("A.csv",header = T)
文件中包含log2FC,-log10(Pval),还有对基因的注释:
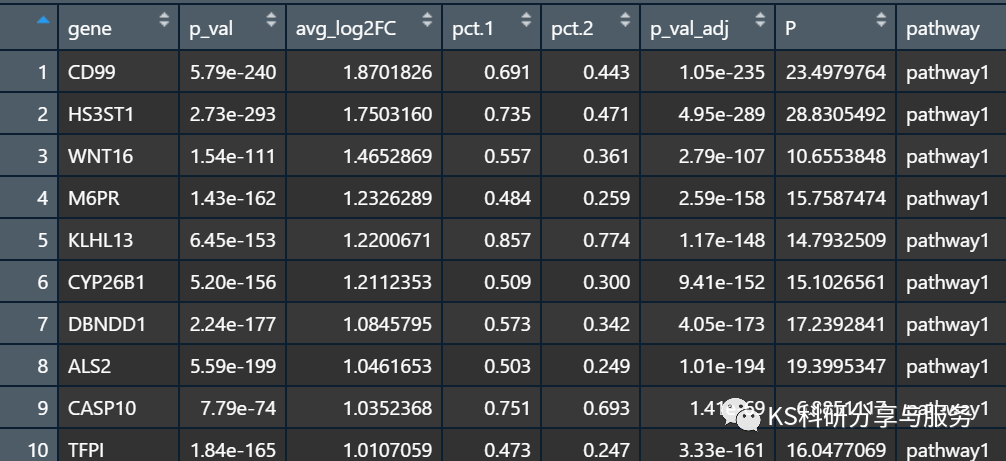
以log2FC为横轴,-log10(Pval)为纵轴画散点图:
#ggplot2画散点图ggplot(data=A, aes(x=avg_log2FC, y=P)) + geom_point()
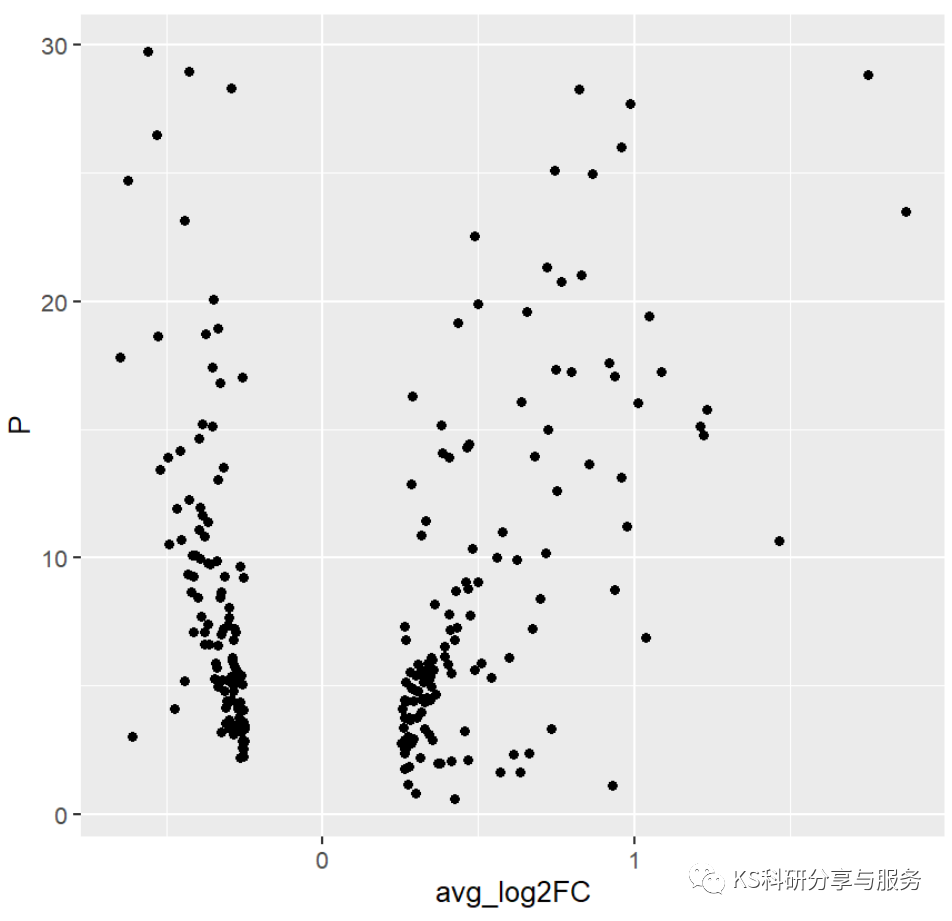
这个图是远远不够的,要对其坐标轴进行修饰。
ggplot(data=A, aes(x=avg_log2FC, y=P)) + geom_point()+theme(panel.background =element_blank())+theme(axis.line=element_line(colour="black", size = 0.5))+labs(y="-log10(Padj)", x = "Log2FC")

这样图的基本雏形就出来了,接着需要做的就是将不同功能的基因用不同的颜色的点标记出来。
A$pathway <- factor(A$pathway)ggplot(data=A, aes(x=avg_log2FC, y=P,color=pathway)) + geom_point()+theme(panel.background =element_blank())+theme(axis.line=element_line(colour="black", size = 0.5))+labs(y="-log10(Padj)", x = "Log2FC")

颜色是标记不同了,但是比较乱,所以自定义一下:
ggplot(data=A, aes(x=avg_log2FC, y=P,color=pathway)) + geom_point()+theme(panel.background =element_blank())+theme(axis.line=element_line(colour="black", size = 0.5))+labs(y="-log10(Padj)", x = "Log2FC")+scale_colour_manual(name ="other", values =c("grey","orange","red","purple","green"))

这样就完成了,与Cell文章的图可以说是一模一样。但是还缺乏点东西,我需要在图上添加文字,表明左侧是对照组,右侧是实验组(知识点:ggplot2画图在任意位置添加自定义文本)。这字可不是P上去的!

ggplot(data=A, aes(x=avg_log2FC, y=P,color=pathway)) + geom_point()+theme(panel.background =element_blank())+theme(axis.line=element_line(colour="black", size = 0.5))+labs(y="-log10(Padj)", x = "Log2FC")+scale_colour_manual(name ="other", values =c("grey","orange","red","purple","green"))+annotate("text", label = "Control", x = -0.5, y = 32)+annotate("text", label = "Tset", x = 1.5, y = 30)
至此,完美的复刻了Cell文章的图。回到本质,这个图还是很有用的,即表现出了差异基因,也表现出了不同差异基因的类型或者功能!




















 945
945











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








