%load_ext rpy2.ipython
%%R -o counts -o meta
suppressMessages(library(splatter))
params <- newSplatParams()
params <- setParam(params, "nGenes", 5000)
params <- setParam(params, "batchCells", c(500,500,500))
params <- setParam(params, "batch.facLoc", 0.5)
params <- setParam(params, "batch.facScale", 0.5)
params <- setParam(params, "group.prob", c(1/3,1/3,1/3))
sim <- splatSimulate(params, method="groups", verbose=FALSE)
counts = data.frame(counts(sim))
meta = data.frame(colData(sim))
import anndata
import scanpy as sc
adata = anndata.AnnData(X=counts.values.T, obs=meta)
sc.pp.normalize_per_cell(adata,counts_per_cell_after=10000)
sc.pp.log1p(adata)
sc.tl.pca(adata,svd_solver='arpack')
sc.pp.neighbors(adata, n_neighbors=9)
sc.tl.umap(adata)
sc.pl.umap(adata,color=['Batch','Group'])
可以看到数据是不用加入“./”, 否则就会报错




今天又发现一个值得记录的地方,就是如果想保存scanpy的umap图像到一个指定位置,之前我以为只能设置sc.settings.figdir,这个其实看起来很不美观
我今天发现一个很简单的方式
import matplotlib.pyplot as plt
sc.pl.umap(adata,color=['Batch','Group'],show=False)
plt.savefig("./aabbcc.png")
我最近又发现一个问题就是说,有时标签太多,会影响到可视化,例如
import scanpy as sc
adata=sc.read("./Immune_ALL_hum_mou.h5ad")
sc.pp.normalize_total(adata,target_sum=1e4)
sc.pp.log1p(adata)
sc.pp.highly_variable_genes(adata,n_top_genes=1000,subset=True)
sc.pp.scale(adata)
sc.tl.pca(adata)
sc.pp.neighbors(adata)
sc.tl.umap(adata)
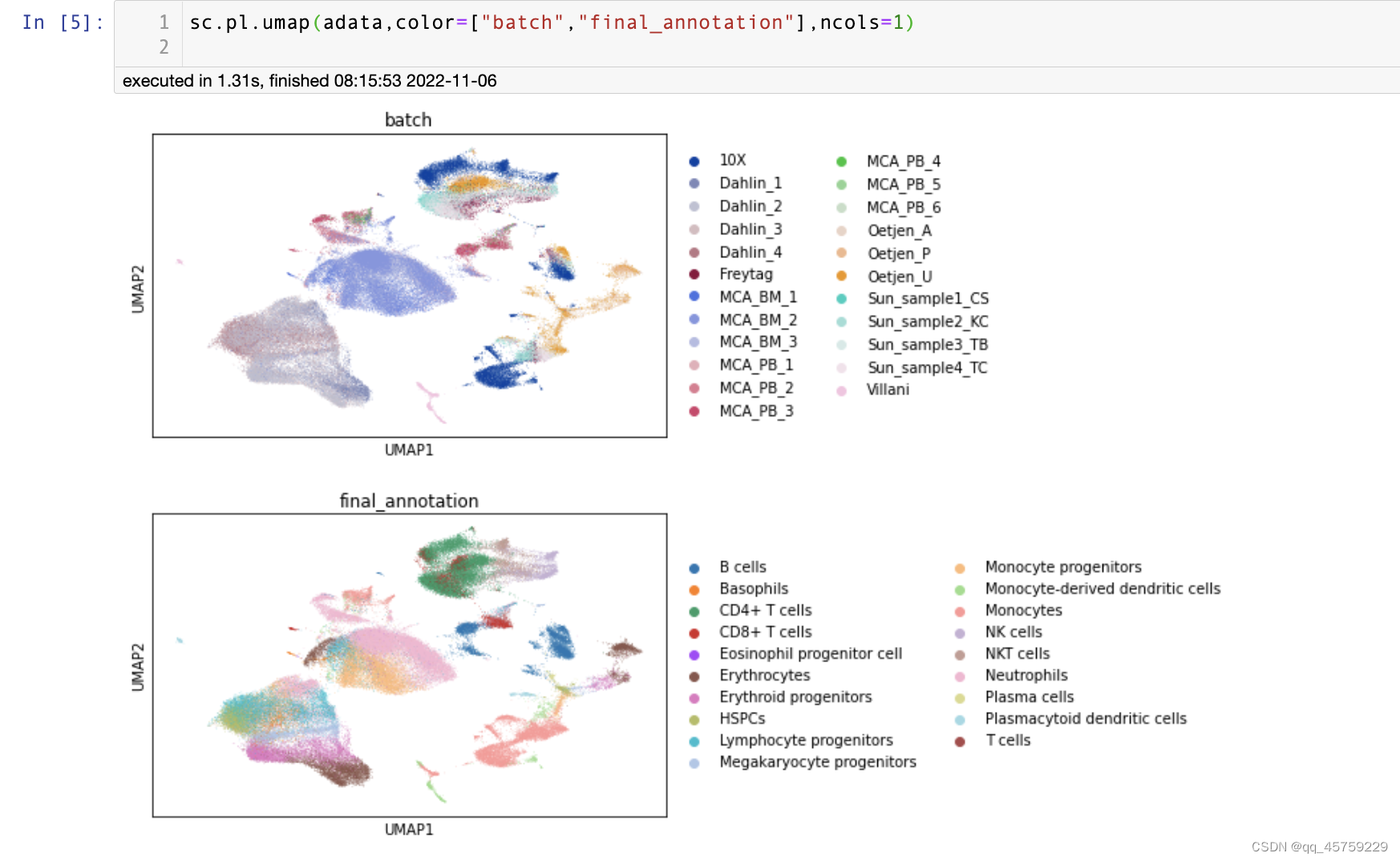
sc.pl.umap(adata,color=["batch","final_annotation"],ncols=1)
结果如下
 但是要想看的更清楚点,使用ncols参数即可
但是要想看的更清楚点,使用ncols参数即可
今天我发现这个保存图片的时候发现另一个问题
 在jupyter中显示的标签么有什么问题,但是保存的图片文件是这样的
在jupyter中显示的标签么有什么问题,但是保存的图片文件是这样的
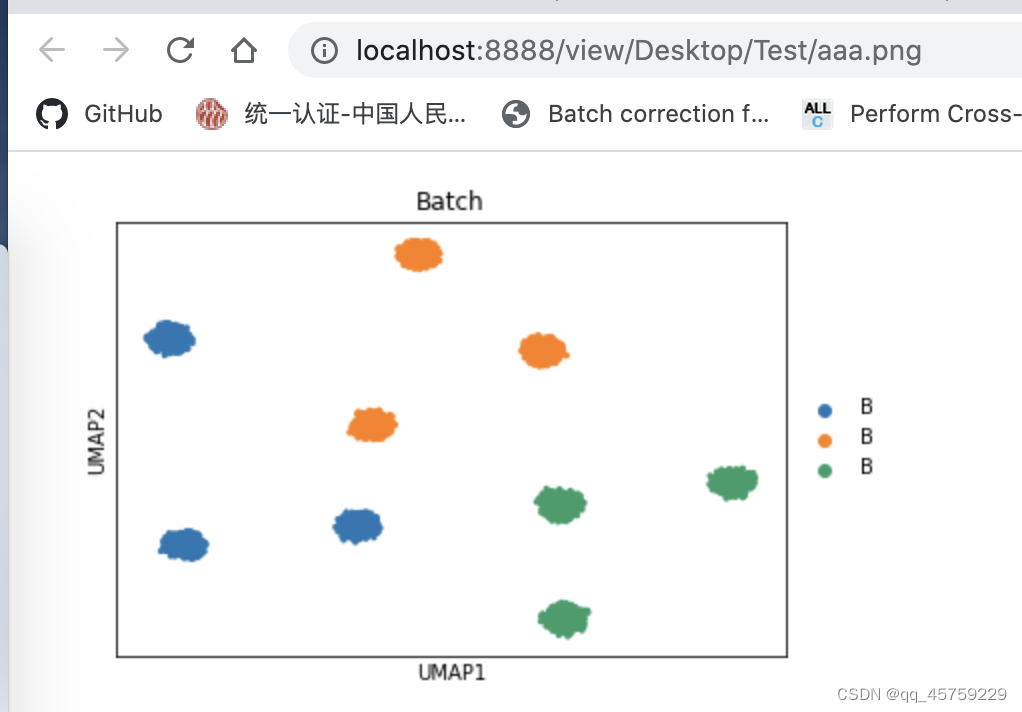 可以看到这个标签值显示一个字母
可以看到这个标签值显示一个字母
解决办法如下
import matplotlib.pyplot as plt
fig,ax=plt.subplots(figsize=(10,6))
sc.pl.umap(adata,color="Batch",show=False,ax=ax)
plt.savefig("./abb.png")

可以看到这个结果就正常了
import anndata
import matplotlib.pyplot as plt
import scanpy as sc
adata=sc.read("/Users/xiaokangyu/Desktop/dataset/single_cell/splatter_sim/4batch_4celltype.h5ad")
sc.pp.normalize_per_cell(adata,counts_per_cell_after=10000)
sc.pp.log1p(adata)
sc.tl.pca(adata,svd_solver='arpack')
sc.pp.neighbors(adata, n_neighbors=9)
sc.tl.umap(adata)
sc.tl.tsne(adata)
sc.tl.louvain(adata)
fig, axs = plt.subplots(4, 2, figsize=(20,16),constrained_layout=True)
sc.pl.tsne(adata, color="Group", title="Group tsne", ax=axs[0,0], show=False)
sc.pl.tsne(adata, color="Batch", title="Batch tsne", ax=axs[0,1], show=False)
sc.pl.umap(adata, color="Group", title="Group umap", ax=axs[1,0], show=False)
sc.pl.umap(adata, color="Batch", title="Batch umap", ax=axs[1,1], show=False)
sc.pl.umap(adata, color="louvain", title="Group umap", ax=axs[2,0], show=False,legend_loc="on data")
sc.pl.umap(adata, color="louvain", title="Batch umap", ax=axs[2,1], show=False,legend_loc="on data")
sc.pl.umap(adata, color="louvain", title="Group umap", ax=axs[3,0], show=False,legend_loc="on data")
sc.pl.tsne(adata, color="Group", title="Group tsne", ax=axs[3,1], show=False)
结果如下

关于legend 只显示部分,还有一种解决办法
sc.pl.tsne(adata, color='Group', title='Denoised counts',show=False)
plt.savefig("./aaa.png")
sc.pl.umap(adata, color="Group", title="Denoised counts",show=False)
plt.savefig("./bbb.png")
sc.pl.tsne(adata, color='celltype', title='Denoised counts',show=False)
plt.savefig("./ccc.png",bbox_inches='tight')
sc.pl.umap(adata, color="celltype", title="Denoised counts",show=False)
plt.savefig("./ddd.png")
这个可视化结果在jupyter中是可以正常显示的,但是保存图片中就会出现问题
 而保存图片时结果如下
而保存图片时结果如下


反正最重要的是要注意两点
sc.pl.umap(adata,color=["label"],title ="label",show=False) ## show=False,一定要加
plt.savefig(save_dir + "{}_full_label.png".format(sample),bbox_inches='tight') # bbox_inches一定要加
adata_sub = adata[adata.obs["label"]!="undetermined"].copy()
sc.pl.umap(adata_sub,color=["label"],title="without undetermined",show=False) # show=False一定要加
plt.savefig(save_dir + "{}_delete_label.png".format(sample),bbox_inches='tight') # bbox_inches一定要加
show=False是一定要加的,否则保存图片是空白的,bbox_inches="tight这个也是一定要加的,否则保存的图片的legend出现问题
























 395
395

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








