叶绿体基因组 四分体结构鉴定
B站视频
序列获取
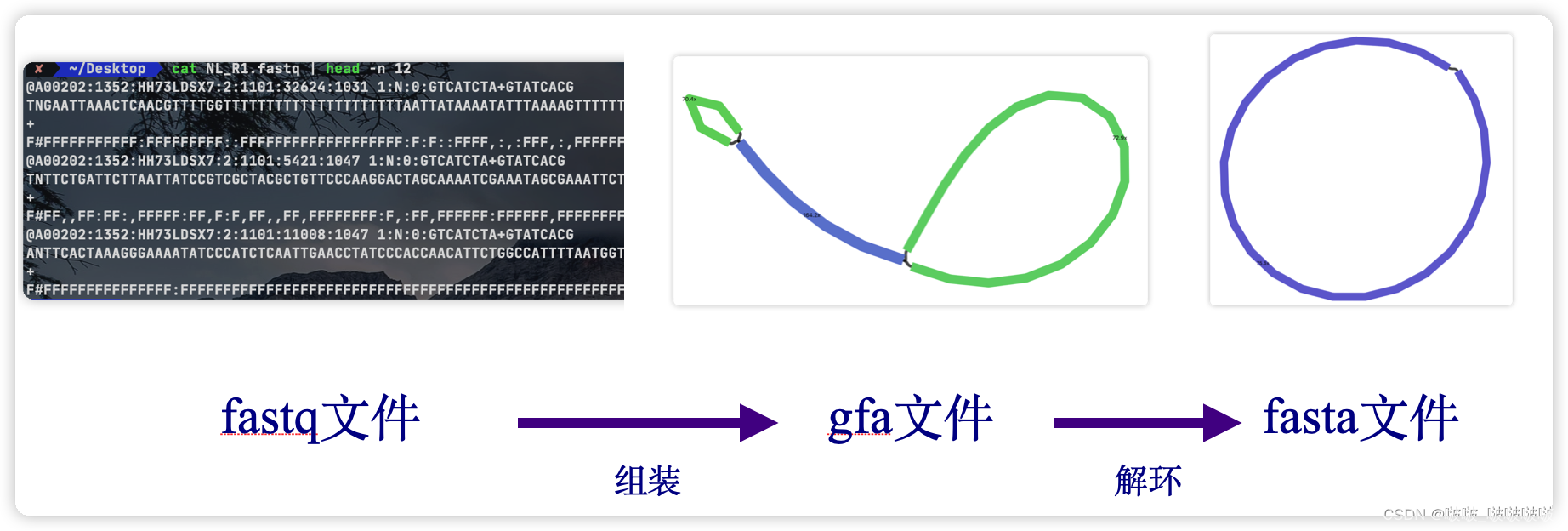
getorganelle和Spades等组装软件,得到的fastg或者gfa文件解环得到fasta文件
序列调整

由于IR区域的存在,因此叶绿体基因组具有四分体结构,分别包括了一个LSC区域,一个LSC区域,两个IR区域,分别命名为IRa和IRb
而在叶绿体基因组分析过程中,首先要做的就是将叶绿体基因组序列调整至以LSC第一个碱基作为序列的起始,但叶绿体基因组为环状结构,可以在任何位置切开,且当跨区域序列较短时,使用Geseq等软件一般很难得到准确的结果,因此自己开发了鉴定四分体结构的脚本,脚本的获取请看上面的B站视频,里面讲解了具体的用法
SSC调整
SSC的调整请使用nucmer实现
conda install nucmer #或者使用源码安装
nucmer --mum -p chloroplast ref.fasta test.fasta
delta-filter -m chloroplast.delta > chloroplast.filter
show-coords -T -r -l chloroplast.filter > chloroplast.1coords
mummerplot --postscript -p chloroplast chloroplast.delta
ps2pdf chloroplast.ps chloroplast.pdf
组装序列SSC方向与参考序列相同时,绘图如下

组装序列SSC方向与参考序列相反时,绘图如下

写在最后

我们首先应该确定参考序列的SSC方向是我们想要的方向,一般是ycf1长基因位于SSC末端的是NCBI中常见的序列。
然后进行共线性分析,
如果结果如图3,表现出良好的共线性时(即一条直线时,则表示SSC方向相同),
如果如图4时,则表示SSC方向相反,因此需要进行SSC方向的调整。具体的脚本和使用方法,请看B站视频。























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








