最近回头重新看了illlumina paired end sequence的测序原理视频,发现了以前没有注意的一些问题,而这些问题也是大家平时容易搞错的,因此花了几天时间将illumina 的paired end sequence 从构建文库到上机测序的整个过程以及原理较为详细的写了出来。
基础知识:illumina测序的核心在于利用可逆终止的、荧光标记的dNTP进行边合成边测序
Flowcell(流动池)是有着2个或8个lane(泳道)的玻璃板,。每个lane可以测一个样本或者多样本的混合物,且随机布满了能够与文库两端接头分别互补配对或一 致的寡核苷酸(oligos,P7和P5接头)。一个lane包含两列,每一列有60个tile,每个tile会种下不同的cluster,每个tile在一次循环中会拍照4次(每个碱基一次)。
paried-end sequencing
一、Library Preparation文库的构建
1. 利用转座子(transposome)对双链DNA进行剪切以及接头(adapter)的连接

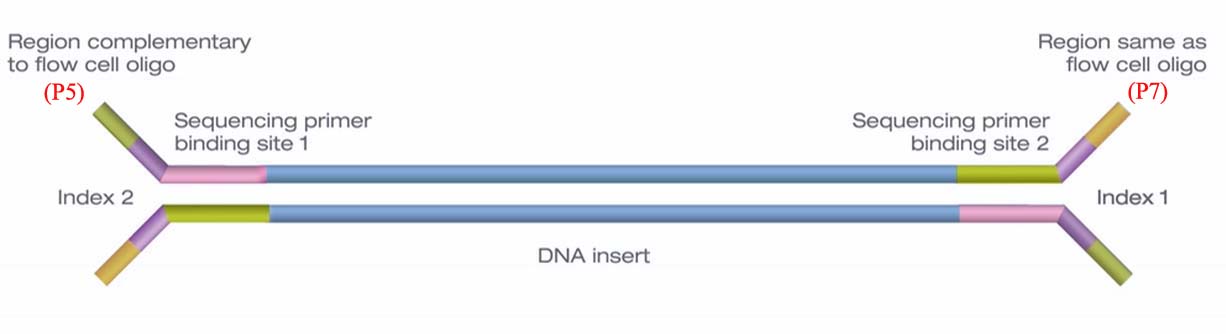
2. 接头连接成功后,利用低循环扩增技术在接头处进行修饰,分别在两端添加sequencing primer binding site1/sequencing primer binding site2(即测序引物结合位点)、index1/index2以及我们称之P5和P7的寡核苷酸序列
上图并没有将之前的adapter标志出来,下图是维基百科的示意图,详细一些。
这里要注意两点(1)P5和P7是不同的,它们分别和flowcell上的接头互补和相同。为了方便阐述,将与P5互补的接头称为P5’,与P7互补的接头称为P7’。(2)index1和index2也是不同的,与P5相连的是index2,与P7相连的是index1。
关于index,也叫barcodes,因为一个lane可以同时测多个样品,为了避免混淆样品的read products,每种样品的DNA由一种index修饰,这样测序得到的reads都是具有index标记的,在测序结果中,依据之前标签与样品的对应关系,就可以获得对应样品的数据。而这里的 index1和index2是为了区分paired-end测序得到的双端reads。二、Cluster generation 簇生成
1. Flowcell上随机分布了两种不同的寡核苷酸序列&#










 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 5178
5178

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








