Illumina测序原理
illumina二代测序平台特点:基于可逆终止的、荧光标记dNTP,实现边合成、边测序的工作
文字描述着实有很多局限性,文章起草基于这份视频(双端index),想要更轻松的阅读体验建议配合视频一起看:http://www.bilibili.com/video/av13107081/?share_source=copy_link&p=1&ts=1610448077&share_medium=iphone&bbid=a68e072c29d899785ac6735b234b9654](https://links.jianshu.com/go?to=http%3A%2F%2Fwww.bilibili.com%2Fvideo%2Fav13107081%2F%3Fshare_source%3Dcopy_link%26p%3D1%26ts%3D1610448077%26share_medium%3Diphone%26bbid%3Da68e072c29d899785ac6735b234b9654%5D(http%3A%2F%2Fwww.bilibili.com%2Fvideo%2Fav13107081%2F%3Fshare_source%3Dcopy_link%26p%3D1%26ts%3D1610448077%26share_medium%3Diphone%26bbid%3Da68e072c29d899785ac6735b234b9654))
注意⚠️:文章内容纯属个人见解,如有错误欢迎批评指正~
1. 文库制备
DNA文库的定义:所谓的DNA文库,实际上是许多个DNA片段,在两头接上了特定的DNA接头。
DNA文库的特点:
- 中间插入的DNA片段:是未知的各式各样的DNA片段,也正是测序仪要检测的序列片段
- 接头序列:是人工特地加上去的,其序列是已知的
如何制作DNA文库:
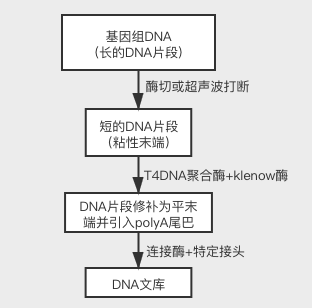
文库制备过程
基因组DNA用超声波打断(也可以用通过酶切的方法)获得短的DNA片段,其粘性末端使用T4-DNA聚合酶补成平末端,然后用klenow酶在3’端加上polyA碱基(方便接头嫁接到DNA片段),最后,连接酶将特定的接头连接上去。连接好接头的DNA片段混合物,我们称为文库。
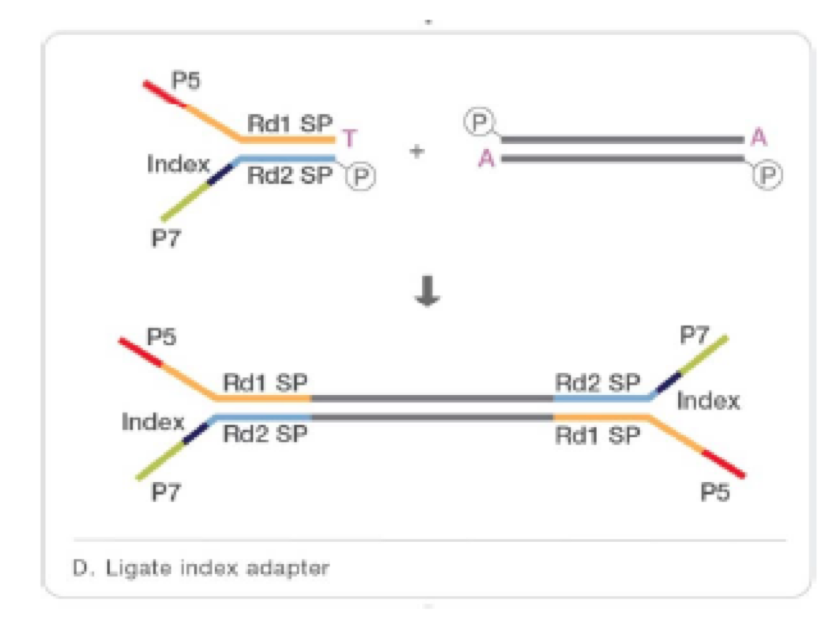
加入接头
2. 成簇反应
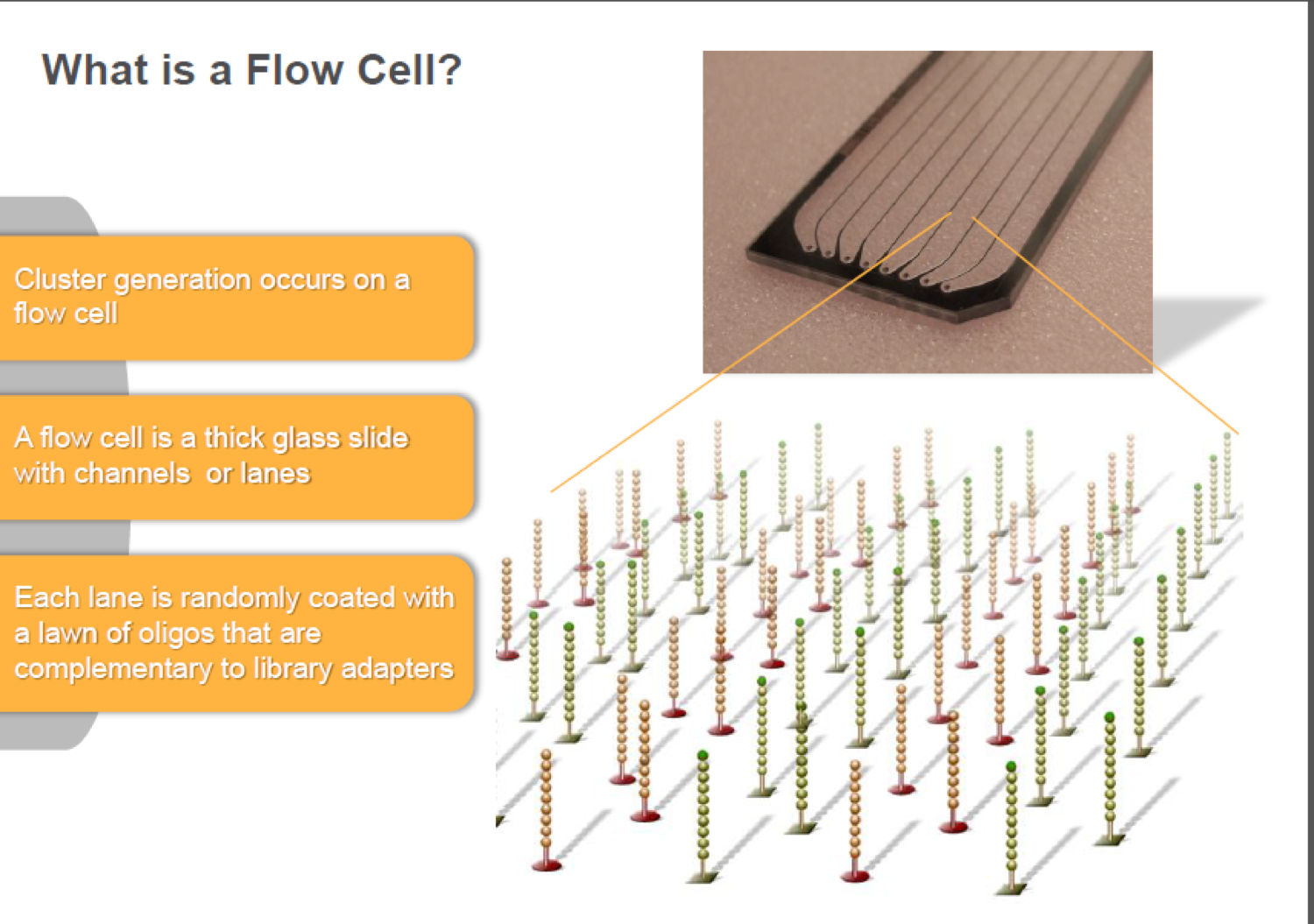
了解flowcell
flowcell(流动池)为一个载玻片大小的芯片,里面做了8条通道,通道的内表面做了专门的化学修饰:主要是用2种寡核苷酸(oligo)通过“共价键”种在玻璃表面-在flowcell通道有液体流动时不会轻易冲掉,这2种寡核苷酸(oligo)后面会和测序DNA文库的接头序列相互互补。
flowcell
1)文库片段附着到“flowcell上的oligo”
DNA文库两头的序列和芯片上的引物碱基互补,因此可以通过氢键力互补杂交。待测序的DNA片段通过氢键力与第一种oligo配对从而固定在flowcell上。
2)被文库片段附着的oligo延伸出互补的DNA链
加入DNA聚合酶和dNTP,flowcell上的oligo做引物,以文库片段为模版,合成出一条全新的DNA链(和原来的文库片段序列完全互补)
3)冲走文库片段
加入NaOH碱溶液,破坏氢键力,模版链(文库片段)被冲走,只剩下与芯片通过共价键连接的DNA链
4)通过“桥式PCR”将序列信息复制到第2种oligo
加入中性液体,中和碱液,创造可以产生氢键力的环境。
此时,第一种oligo所在DNA链上的自由端,会通过成桥的方式和flowcell上的第二种oligo互补配对(形成“单链桥”)。我们加入聚合酶和dNTP,聚合酶就沿着第二个oligo合成出一条新的链来(形成“双链桥”)。
5)DNA链线性化
加入NaOH碱溶液,破坏氢键力,“双链桥”解开(两个单链被固定在flowcell)
6)重复“桥式PCR”
再加入中性液体,中和碱液。DNA链的游离末端又和flowcel上新的oligo成桥,再加酶和dNTP,在尚未被占用的oligo上合成新的DNA链。
连续重复以上过程,DNA链的数量以指数方式增长
7)形成测序链
多次桥式PCR完成之后,实现了DNA文库序列在flowcell上的成倍扩增(实则是为了放大测序阶段的荧光信号)
后面要把合成的双链,变成可以测序的单链
**方法:**把反义链上的特定基团切断,即断开了反义链与oligo的连接,然后用碱溶液冲洗,被切断了的DNA链被冲掉,只留下通过共价键连接的正义链。
同时,DNA链的3’端被封锁,以防止与oligo的非特异结合。
3. 测序阶段
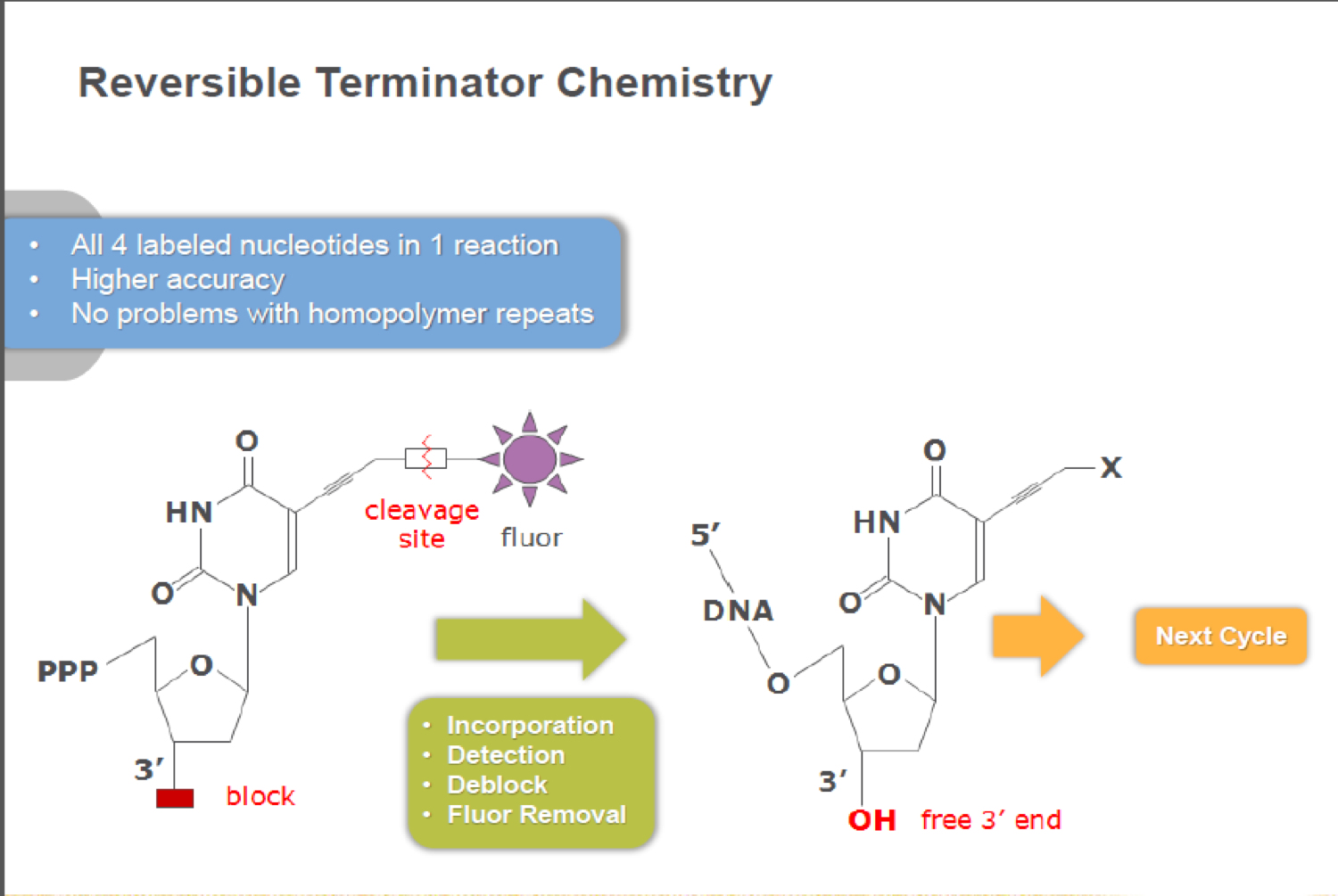
了解 可逆终止、荧光标记的dNTP:
荧光标记的dNTP:不同碱基的识别信号(4种dNTP,每一种上面标的荧光素都不一样)
阻断基团:通过控制 3’羟基,控制碱基的合成节奏
特殊的dNTP
1)read1读取
溶液状态:加入中性溶液,使其适应测序阶段。
所需原料:加入read1测序引物,荧光标记的dNTP,DNA聚合酶
测序过程:
- 合成一个碱基:根据碱基互补的原理,DNA聚合酶将正确的dNTP合成到新的链上,合成一个碱基后即停止
- 激光扫描:用溶液把多余的dNTP和酶冲掉,然后激光扫描,根据发出来的荧光判断它是那个碱基,根据碱基互补的原则,反推模版链上的碱基
- 切掉荧光基团和阻断基团:加入化学试剂,把叠氮碱基和旁边标记的荧光基团切掉(暴露出3’羟基)
- 加入新的dNTP和酶:又延长一个碱基,把多余的酶和dNTP冲掉,激光扫描判断碱基类型
- 重复以上过程,把上百个碱基读出来(循环的次数取决于读长)
- read1读完之后,读段产物会被碱液冲洗掉
2)读 index1 序列
因为测序仪的测序通量很大,一个样本用不到几百万条DNA,因此常常多个样本的DNA文库混在一起在同一条lane测序。而每一个样本,都有一个特定的索引序列相互区分(也就是我们常说的index序列)。
- 加入index1引物,与模版碱基互补杂交
- 与reads1的判读过程类似,加入荧光标记的dNTP和DNA聚合酶判读碱基,读完后用碱液冲洗掉
3)读 index2 序列
- 3’端解除封锁
- 正义链折叠结合到flowcell上的第2个oligo(形成“单链桥”)
- 加入荧光标记的dNTP和DNA聚合酶,读取index2
4)读read2序列
形成反义链:
index2读完之后,聚合酶继续延伸,形成双链桥
然后加入碱液将DNA双链线性化
正义链上的特定基团被切除并洗掉,只留下反义链
加入read2的测序引物,与reads1的读取一样,重复测序步骤
4. 数据分析
测序获得的数百万个读段序列都通过 index序列 分离归类到对应的样本。
具有相似延伸碱基的reads被聚类在一起,正向和反向reads配对形成连续序列,它们与参考基因组比对,用于突变识别(variant identification)

类在一起,正向和反向reads配对形成连续序列,它们与参考基因组比对,用于突变识别(variant identification)
[外链图片转存中…(img-0irlMJH7-1649923447618)]
突变类型的判读






















 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








