
点击关注,桓峰基因
桓峰基因
生物信息分析,SCI文章撰写及生物信息基础知识学习:R语言学习,perl基础编程,linux系统命令,Python遇见更好的你
97篇原创内容
公众号
桓峰基因的教程不但教您怎么使用,还会定期分析一些相关的文章,学会教程只是基础,但是如果把分析结果整合到文章里面才是目的,觉得我们这些教程还不错,并且您按照我们的教程分析出来不错的结果发了文章记得告知我们,并在文章中感谢一下我们哦!
公司英文名称:Kyoho Gene Technology (Beijing) Co.,Ltd.
如果您觉得这些确实没基础,需要专业的生信人员帮助分析,直接扫码加微信nihaoooo123,我们24小时在线!!

前 言
梯度提升机是一个强大的机器学习技术家族,在广泛的实际应用中显示了相当大的成功。它们可以根据应用程序的特定需求进行高度定制,就像学习不同的损失函数一样。这篇文章提供了一个教程,介绍梯度提升方法的方法论,重点关注建模的机器学习方面。理论信息是补充描述性的例子和插图,涵盖梯度推进模型设计的所有阶段。讨论了处理模型复杂性的注意事项。给出了三个梯度助推应用实例,并进行了综合分析。

基本原理
梯度提升法的主要思想是,先建立一个某种形式的初始模型(线性、样条、树或其他),称为基学习器;然后检查残差,在残差的基础上围绕损失函数拟合模型。损失函数测量模型和现实之间的差别,例如,在回归问题中可以用误差的平方。一直继续这个过程,直到满足某个特定的结束条件。这与下面的情形有点相似:一个学生进行模拟考试,100道题中错了30道,然后只研究那30道错题;在下次模考中,30道题中又错了10道,然后只研究那10道题,以此类推。

实例解析
1. 软件安装
if (!require(xgboost)) install.packages("xgboost")
if (!require(caret)) install.packages("caret")
library(xgboost)
library(caret)
2. 数据读取
数据来源《机器学习与R语言》书中,具体来自UCI机器学习仓库。地址:http://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/ 下载wbdc.data和wbdc.names这两个数据集,数据经过整理,成为面板数据。查看数据结构,其中第一列为id列,无特征意义,需要删除。第二列diagnosis为响应变量(B,M),字符型,一般在R语言中分类任务都要求响应变量为因子类型,因此需要做数据类型转换。剩余的为预测变量,数值类型。查看数据维度,568个样本,32个特征(包括响应特征)。
BreastCancer <- read.csv("wisc_bc_data.csv", stringsAsFactors = FALSE)
dim(BreastCancer)
## [1] 568 32
table(BreastCancer$diagnosis)
##
## B M
## 357 211
3. 数据分布
比较恶性和良性之间的差距
library(reshape2)
bc <- BreastCancer[, -1]
bc.melt <- melt(bc, id.var = "diagnosis")
head(bc.melt)
## diagnosis variable value
## 1 M radius_mean 20.57
## 2 M radius_mean 19.69
## 3 M radius_mean 11.42
## 4 M radius_mean 20.29
## 5 M radius_mean 12.45
## 6 M radius_mean 18.25
ggplot(data = bc.melt, aes(x = diagnosis, y = log(value + 1), fill = diagnosis)) +
geom_boxplot() + theme_bw() + facet_wrap(~variable, ncol = 8)
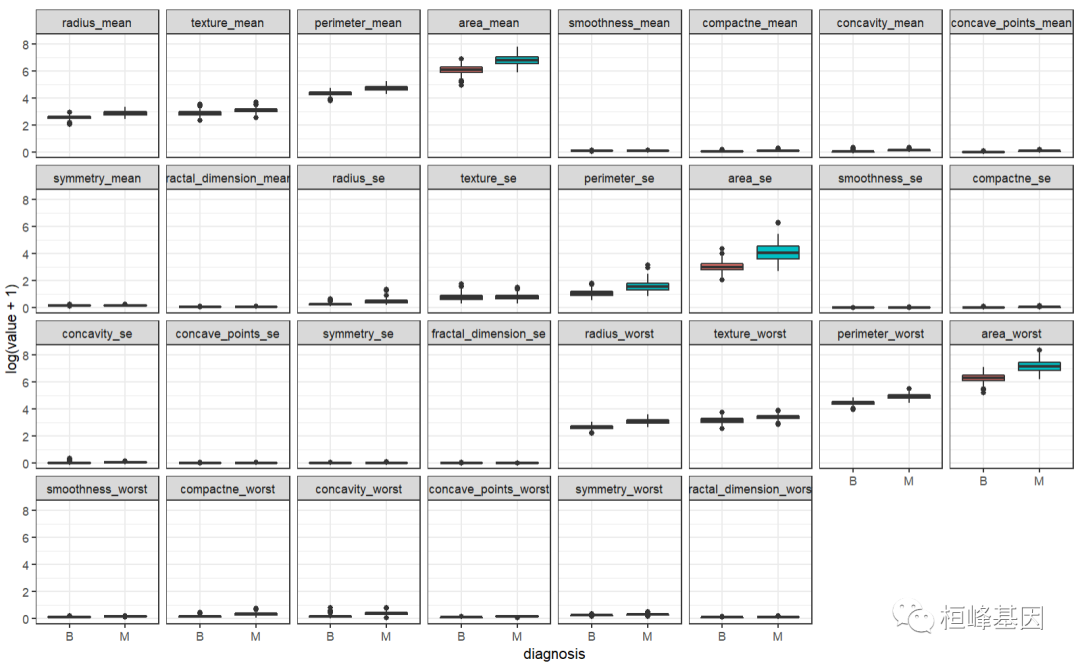
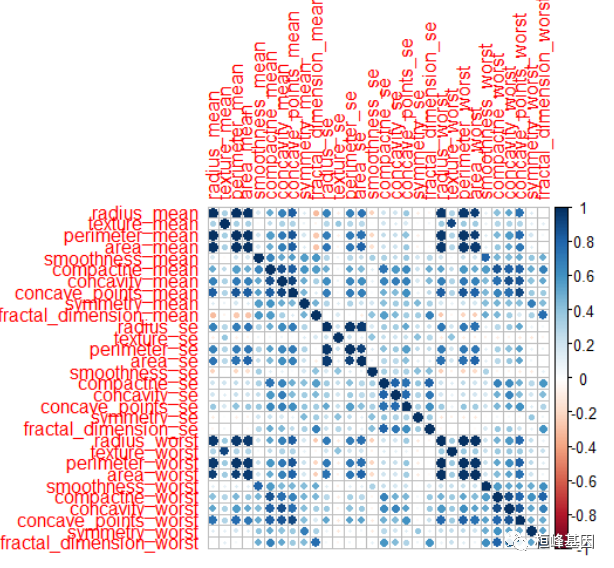
比较变量之间的相关性,如下:
library(tidyverse)
data <- select(BreastCancer, -1) %>%
mutate_at("diagnosis", as.factor)
data$diagnosis = ifelse(data$diagnosis == "B", "B", "M")
sum(is.na(data))
## [1] 0
corrplot::corrplot(cor(data[, -1]))
4. 数据分割
当我们只有一套数据的时候,可以将数据分为训练集和测试集,具体怎么分割可以看公众号的专题:Topic 5. 样本量确定及分割我们将整个数据进行分割,分为训练集和测试集,并保证其正负样本的比例,如下:
# 数据分割 install.packages('sampling')
library(sampling)
set.seed(123)
# 每层抽取70%的数据
train_id <- strata(data, "diagnosis", size = rev(round(table(data$diagnosis) * 0.7)))$ID_unit
# 训练数据
train_data <- data[train_id, ]
# 测试数据
test_data <- data[-train_id, ]
# 查看训练、测试数据中正负样本比例
prop.table(table(train_data$diagnosis))
##
## B M
## 0.6281407 0.3718593
prop.table(table(test_data$diagnosis))
##
## B M
## 0.6294118 0.3705882
5. 模型构建
参数配置
在这一节要使用已经加载的xgboost包。因为这种方法的效果如雷贯耳,所以我们直接用它解决最有挑战性的乳腺癌诊断性问题。正如在提升算法简介中所说,我们要调整一些参数。(1) nrounds:最大迭代次数(最终模型中树的数量)。(2) colsample_bytree:建立树时随机抽取的特征数量,用一个比率表示,默认值为1(使用100%的特征)。(3) min_child_weight:对树进行提升时使用的最小权重,默认为1o(4) eta:学习率,每棵树在最终解中的贡献,默认为0.3。(5) gamma:在树中新增一个叶子分区时所需的最小减损。(6)subsample:子样本数据占整个观测的比例,默认值为1(100%)。(7) max_depth:单个树的最大深度。
使用expand.grid()函数可以建立实验网格,以运行caret包的训练过程。对于前面列出的参数,如果没有设定具体值,那么即使有默认值,运行函数时也会收到出错信息。下面的参数取值是基于我以前的一些训练迭代而设定的。建议各位亲自实验参数调整过程。使用以下命令建立网格:
###参数选择
grid = expand.grid(
nrounds = c(75, 100),
colsample_bytree = 1,
min_child_weight = 1,
eta = c(0.01, 0.1, 0.3), #0.3 is default,
gamma = c(0.5, 0.25),
subsample = 0.5,
max_depth = c(2, 3)
)
grid
## nrounds colsample_bytree min_child_weight eta gamma subsample max_depth
## 1 75 1 1 0.01 0.50 0.5 2
## 2 100 1 1 0.01 0.50 0.5 2
## 3 75 1 1 0.10 0.50 0.5 2
## 4 100 1 1 0.10 0.50 0.5 2
## 5 75 1 1 0.30 0.50 0.5 2
## 6 100 1 1 0.30 0.50 0.5 2
## 7 75 1 1 0.01 0.25 0.5 2
## 8 100 1 1 0.01 0.25 0.5 2
## 9 75 1 1 0.10 0.25 0.5 2
## 10 100 1 1 0.10 0.25 0.5 2
## 11 75 1 1 0.30 0.25 0.5 2
## 12 100 1 1 0.30 0.25 0.5 2
## 13 75 1 1 0.01 0.50 0.5 3
## 14 100 1 1 0.01 0.50 0.5 3
## 15 75 1 1 0.10 0.50 0.5 3
## 16 100 1 1 0.10 0.50 0.5 3
## 17 75 1 1 0.30 0.50 0.5 3
## 18 100 1 1 0.30 0.50 0.5 3
## 19 75 1 1 0.01 0.25 0.5 3
## 20 100 1 1 0.01 0.25 0.5 3
## 21 75 1 1 0.10 0.25 0.5 3
## 22 100 1 1 0.10 0.25 0.5 3
## 23 75 1 1 0.30 0.25 0.5 3
## 24 100 1 1 0.30 0.25 0.5 3
交叉验证
使用car包的train()函数之前,我要创建一个名为cntrl的对象,来设定trainControl的参数。这个对象会保存我们要使用的方法,以训练调优参数。我们使用10折交叉验证,代码如下所示:
cntrl = trainControl(method = "cv", number = 10, verboseIter = FALSE, returnData = FALSE,
returnResamp = "final")
根据配置好的参数,进行建模
set.seed(123)train.xgb = train(x = train_data[, -1], y = train_data[, 1], trControl = cntrl, tuneGrid = grid, method = "xgbTree")
train.xgb
## eXtreme Gradient Boosting ## ## No pre-processing## Resampling: Cross-Validated (10 fold) ## Summary of sample sizes: 359, 358, 358, 358, 358, 359, ... ## Resampling results across tuning parameters:## ## eta max_depth gamma nrounds Accuracy Kappa ## 0.01 2 0.25 75 0.9448077 0.8815793## 0.01 2 0.25 100 0.9448718 0.8825558## 0.01 2 0.50 75 0.9448077 0.8811347## 0.01 2 0.50 100 0.9448077 0.8814371## 0.01 3 0.25 75 0.9498718 0.8926807## 0.01 3 0.25 100 0.9473077 0.8868425## 0.01 3 0.50 75 0.9448077 0.8815793## 0.01 3 0.50 100 0.9473077 0.8868425## 0.10 2 0.25 75 0.9623718 0.9187054## 0.10 2 0.25 100 0.9598718 0.9133019## 0.10 2 0.50 75 0.9548077 0.9022024## 0.10 2 0.50 100 0.9623718 0.9192980## 0.10 3 0.25 75 0.9648718 0.9247034## 0.10 3 0.25 100 0.9673718 0.9298262## 0.10 3 0.50 75 0.9548077 0.9022182## 0.10 3 0.50 100 0.9598718 0.9128120## 0.30 2 0.25 75 0.9623718 0.9190002## 0.30 2 0.25 100 0.9648718 0.9241266## 0.30 2 0.50 75 0.9623077 0.9184364## 0.30 2 0.50 100 0.9598077 0.9133136## 0.30 3 0.25 75 0.9649359 0.9247785## 0.30 3 0.25 100 0.9699359 0.9357355## 0.30 3 0.50 75 0.9748718 0.9457522## 0.30 3 0.50 100 0.9748718 0.9457522## ## Tuning parameter 'colsample_bytree' was held constant at a value of 1## ## Tuning parameter 'min_child_weight' was held constant at a value of 1## ## Tuning parameter 'subsample' was held constant at a value of 0.5## Accuracy was used to select the optimal model using the largest value.## The final values used for the model were nrounds = 75, max_depth = 3, eta## = 0.3, gamma = 0.5, colsample_bytree = 1, min_child_weight = 1 and subsample## = 0.5.
最优参数设置
由此可以得到最优的参数组合来建立模型。模型在训练数据上的正确率是0.975, Kappa值是0.946。下面要做的事情有点复杂,但我认为这才是最佳实践。首先创建一个参数列表,供xgboost包的训练函数xgb.train()使用。然后将数据框转换为一个输入特征矩阵,以及一个带标号的数值型结果列表(其中的值是0和1)。接着,将特征矩阵和标号列表组合成符合要求的输入,即一个xgb.Dmatrix对象。代码如下:
param <- list(objective = "binary:logistic", booster = "gbtree", eval_metric = "error",
eta = 0.1, max_depth = 2, subsample = 0.5, colsample_bytree = 1, gamma = 0.5)
x <- as.matrix(train_data[, -1])
y <- ifelse(train_data$diagnosis == "B", 0, 1)
train.mat <- xgb.DMatrix(data = x, label = y)
优化模型
set.seed(123)
xgb.fit <- xgb.train(params = param, data = train.mat, nrounds = 100)
xgb.fit
## ##### xgb.Booster
## raw: 87 Kb
## call:
## xgb.train(params = param, data = train.mat, nrounds = 100)
## params (as set within xgb.train):
## objective = "binary:logistic", booster = "gbtree", eval_metric = "error", eta = "0.1", max_depth = "2", subsample = "0.5", colsample_bytree = "1", gamma = "0.5", validate_parameters = "TRUE"
## xgb.attributes:
## niter
## callbacks:
## cb.print.evaluation(period = print_every_n)
## # of features: 30
## niter: 100
## nfeatures : 30
可视化变量
从图上我们可以出前几个重要的变量占比几乎达到0.3,这个阈值是在之前grid网格中设置:eta = c(0.01, 0.1, 0.3), #0.3 is default。
impMatrix <- xgb.importance(feature_names = dimnames(x)[[2]], model = xgb.fit)
impMatrix
## Feature Gain Cover Frequency
## 1: concave_points_worst 0.2967527144 0.147574780 0.074766355
## 2: concave_points_mean 0.2341084307 0.144084708 0.079439252
## 3: radius_worst 0.0776151887 0.075881804 0.056074766
## 4: perimeter_worst 0.0742989918 0.080783308 0.065420561
## 5: area_se 0.0707442192 0.075966262 0.065420561
## 6: texture_worst 0.0431688078 0.084473059 0.107476636
## 7: area_worst 0.0349753102 0.054806620 0.056074766
## 8: concavity_worst 0.0274044659 0.045700578 0.060747664
## 9: texture_mean 0.0242682068 0.042618830 0.056074766
## 10: smoothness_worst 0.0180421347 0.030139745 0.046728972
## 11: area_mean 0.0137108659 0.022756702 0.018691589
## 12: compactne_mean 0.0105402518 0.012610045 0.028037383
## 13: symmetry_se 0.0104209161 0.020799297 0.042056075
## 14: smoothness_mean 0.0090815731 0.020561037 0.032710280
## 15: radius_se 0.0084606195 0.029712377 0.018691589
## 16: compactne_se 0.0080685708 0.014326525 0.032710280
## 17: symmetry_worst 0.0076659203 0.027927952 0.037383178
## 18: fractal_dimension_mean 0.0057942369 0.008967406 0.014018692
## 19: smoothness_se 0.0050382309 0.018076694 0.023364486
## 20: fractal_dimension_se 0.0043507637 0.007250846 0.018691589
## 21: fractal_dimension_worst 0.0029493268 0.006075975 0.009345794
## 22: symmetry_mean 0.0026459958 0.006109480 0.014018692
## 23: concavity_mean 0.0023260374 0.003932079 0.009345794
## 24: perimeter_mean 0.0022726213 0.003359381 0.004672897
## 25: compactne_worst 0.0019021532 0.003878213 0.009345794
## 26: concave_points_se 0.0011308883 0.002550426 0.004672897
## 27: radius_mean 0.0010039937 0.002045465 0.004672897
## 28: perimeter_se 0.0008485220 0.002095077 0.004672897
## 29: texture_se 0.0004100425 0.004935332 0.004672897
## Feature Gain Cover Frequency
xgb.plot.importance(impMatrix, main = "Gain by Feature")

模型性能评估
library(InformationValue)
pred <- predict(xgb.fit, x)
optimalCutoff(y, pred)
## [1] 0.2692855
测试集验证
testMat <- as.matrix(test_data[, -1])
xgb.test <- predict(xgb.fit, testMat)
y.test <- ifelse(test_data$diagnosis == "B", 0, 1)
optimalCutoff(y.test, xgb.test)
## [1] 0.1793153
混淆矩阵计算假阳和假阴个数,如下:
confusionMatrix(y.test, xgb.test, threshold = 0.29)
## 0 1
## 0 103 0
## 1 4 63
1 - misClassError(y.test, xgb.test, threshold = 0.29)
## [1] 0.9765
绘制ROC曲线
最后就是模型的准确性评估,这里我们使用的是InformationValue软件包中的plotROC,绘制ROC曲线,如下:
###
library(InformationValue)
plotROC(y.test, xgb.test)

结果解读
我们再回忆一下对乳腺癌数据我们都用过哪些机器学习方法。
基于乳腺癌的数据我们已经做过四种类型的机器学习算法,如下:
这期使用梯度提升准确率为0.997,准确率嗖的一下就上去了,这模型应该是让老板十万分满意。
其实这说明做模型时,不应单一只是用一种算法,需要多种算法比较,找到最优的选择!
还有就是注意绘制ROC的方法,在做分类随机森林时我们使用的是ROSE软件包,而在做回归随机森林时我们使用的是ROCR,需要注意使用时方法的选择。在这里我们使用InformationValue软件包里面的plotROC
References:
- Frontiers in Neurorobotics, Gradient boosting machines,a tutorial,Natekin A., Knoll A.(2013)






















 522
522











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








