
有了R语言的基础,以及ggplot2绘图基础,我们的生信常用分析图形的绘制就可以提上日程了!本系列,师兄就开始带着大家一起学习如何用R语言绘制我们自己的各种分析图吧!
由于本系列的所有分析代码均为师兄细心整理和详细注释而成的!欢迎点赞、收藏、转发!
您的支持是我持续更新的最大动力!
系列内容包括:
- 各种类型的热图你学会了吗?
- 普通热图
- 环形热图
- 解锁火山图真谛!
- plot函数就能画火山图?
- 高级函数绘制火山图–ggplot2、ggpurb
- 经典富集分析及气泡图、柱状图绘制
- 气泡图绘制
- 柱状图绘制
- 富集分析圈图
- 富集分析弦图
- 绘制一张可以打动审稿人的桑基图
- 生存分析 – KM曲线图
- 基础PCA图
- 云雨图
- 韦恩图
- 环形互作网络图
- 相互作用网络图
- 聚类树美化
- 富集分析气泡图进阶版
- mantel test相关性图
- 词云图
- 瀑布图
- 森林图
- 曼哈顿图
- 哑铃图
- 三线表
- 嵌套圈图
- 列线图
- 蜂群图
- 箱线图+贝塞尔曲线
- 矩阵散点图
- 等等,想到再继续补充!!!
本期富集分析圈图效果展示
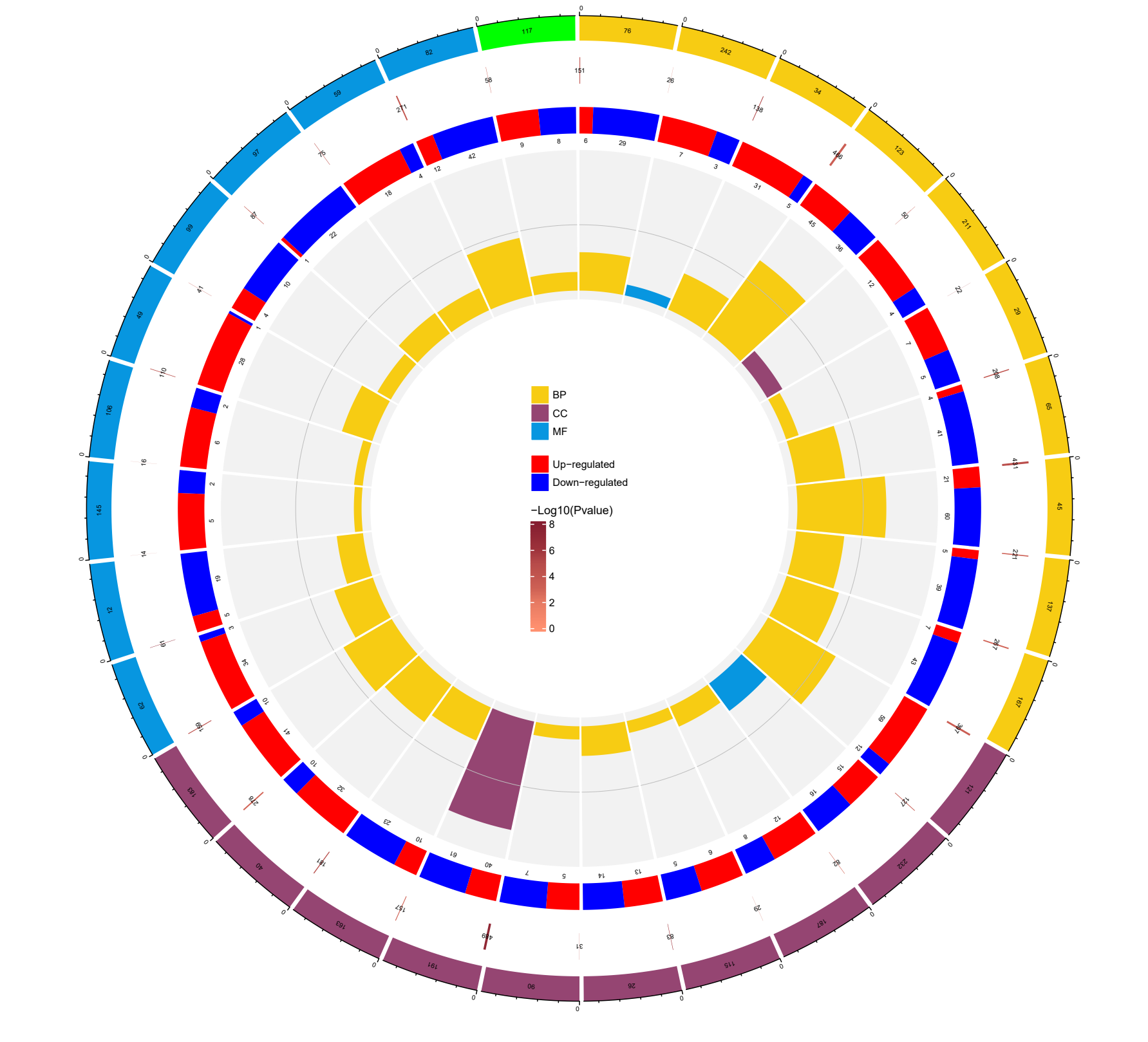
示例数据构建和展示:
# 绘制富集分析圈图
library(circlize)
library(grid)
library(graphics)
library(ComplexHeatmap)
data <- read.table("GO_enrichment_stat.txt",header = T)
data_new <- data[,c(2,1)]
# 通路总基因数:min -- 0、 max -- 生物的总基因数、rich表示该通路中的基因数;
data_new$gene_num.min <- 0
data_new$gene_num.max <- as.numeric(strsplit(data$BgRatio[1], "/")[[1]][2])
data_new$gene_num.rich <- as.numeric(unlist(lapply(data$BgRatio,
function(x) strsplit(x,"/")[[1]][1])))
# -log10 p值
data_new$"-log10Pvalue" <- -log10(data$pvalue)
# 随机创建一个上下调的比例:
ratio <- runif(nrow(data),0,1)
# 根据这个比例计算上调和下调各自占多少:
rich_gene_num <- as.numeric(unlist(lapply(data$GeneRatio,
function(x) strsplit(x,"/")[[1]][1])))
data_new$up.regulated <- round(rich_gene_num*ratio)
data_new$down.regulated <- rich_gene_num - data_new$up.regulated
# 富集分数:差异基因中有多少比例在这个通路中:
# 我这里为了让柱状图更清楚,虚构了一个ratio,不具有实际意义!
data_new$rich.factor <- rich_gene_num/120
colnames(data_new)[c(1,2)] <- c("id", "category")
# 随机取30个GO作图,这里大家可以根据具体情况选择合适的通路:
dat <- data_new[sample(247,30),]
dat$id <- factor(rownames(dat), levels = rownames(dat))
# 查看改造后的示例数据:
> head(dat)
id category gene_num.min gene_num.max gene_num.rich -log10Pvalue up.regulated down.regulated
76 76 BP 0 18866 151 4.038230 6 29
242 242 MF 0 18866 26 3.090989 7 3
34 34 BP 0 18866 138 5.335862 31 5
123 123 BP 0 18866 466 3.397800 45 36
211 211 CC 0 18866 50 3.718427 12 4
29 29 BP 0 18866 22 5.741276 7 5
rich.factor all.regulated up.proportion down.proportion up down
76 0.29166667 35 0.1714286 0.8285714 3234.171 18866
242 0.08333333 10 0.7000000 0.3000000 13206.200 18866
34 0.30000000 36 0.8611111 0.1388889 16245.722 18866
123 0.67500000 81 0.5555556 0.4444444 10481.111 18866
211 0.13333333 16 0.7500000 0.2500000 14149.500 18866
29 0.10000000 12 0.5833333 0.4166667 11005.167 18866
绘图
# 第一个圈:绘制id
circle_size = unit(1, 'snpc')
circos.par(gap.degree = 0.5, start.degree = 90)
plot_data <- dat[c('id', 'gene_num.min', 'gene_num.max')]
ko_color <- c(rep('#F7CC13',10), rep('#954572',10), rep('#0796E0',9), rep('green', 6)) #各二级分类的颜色和数目
circos.genomicInitialize(plot_data, plotType = NULL, major.by = 1)
circos.track(
ylim = c(0, 1), track.height = 0.05, bg.border = NA, bg.col = ko_color,
panel.fun = function(x, y) {
ylim = get.cell.meta.data('ycenter')
xlim = get.cell.meta.data('xcenter')
sector.name = get.cell.meta.data('sector.index')
circos.axis(h = 'top', labels.cex = 0.4, labels.niceFacing = FALSE)
circos.text(xlim, ylim, sector.name, cex = 0.4, niceFacing = FALSE)
} )

# 第二圈,绘制富集的基因和富集p值
plot_data <- dat[c('id', 'gene_num.min', 'gene_num.rich', '-log10Pvalue')]
label_data <- dat['gene_num.rich']
p_max <- round(max(dat$'-log10Pvalue')) + 1
colorsChoice <- colorRampPalette(c('#FF906F', '#861D30'))
color_assign <- colorRamp2(breaks = 0:p_max, col = colorsChoice(p_max + 1))
circos.genomicTrackPlotRegion(
plot_data, track.height = 0.08, bg.border = NA, stack = TRUE,
panel.fun = function(region, value, ...) {
circos.genomicRect(region, value, col = color_assign(value[[1]]), border = NA, ...)
ylim = get.cell.meta.data('ycenter')
xlim = label_data[get.cell.meta.data('sector.index'),1] / 2
sector.name = label_data[get.cell.meta.data('sector.index'),1]
circos.text(xlim, ylim, sector.name, cex = 0.4, niceFacing = FALSE)
} )
# 第三圈,绘制上下调基因
dat$all.regulated <- dat$up.regulated + dat$down.regulated
dat$up.proportion <- dat$up.regulated / dat$all.regulated
dat$down.proportion <- dat$down.regulated / dat$all.regulated
dat$up <- dat$up.proportion * dat$gene_num.max
plot_data_up <- dat[c('id', 'gene_num.min', 'up')]
names(plot_data_up) <- c('id', 'start', 'end')
plot_data_up$type <- 1
dat$down <- dat$down.proportion * dat$gene_num.max + dat$up
plot_data_down <- dat[c('id', 'up', 'down')]
names(plot_data_down) <- c('id', 'start', 'end')
plot_data_down$type <- 2
plot_data <- rbind(plot_data_up, plot_data_down)
label_data <- dat[c('up', 'down', 'up.regulated', 'down.regulated')]
color_assign <- colorRamp2(breaks = c(1, 2), col = c('red', 'blue'))
circos.genomicTrackPlotRegion(
plot_data, track.height = 0.08, bg.border = NA, stack = TRUE,
panel.fun = function(region, value, ...) {
circos.genomicRect(region, value, col = color_assign(value[[1]]), border = NA, ...)
ylim = get.cell.meta.data('cell.bottom.radius') - 0.5
xlim = label_data[get.cell.meta.data('sector.index'),1] / 2
sector.name = label_data[get.cell.meta.data('sector.index'),3]
circos.text(xlim, ylim, sector.name, cex = 0.4, niceFacing = FALSE)
xlim = (label_data[get.cell.meta.data('sector.index'),2]+label_data[get.cell.meta.data('sector.index'),1]) / 2
sector.name = label_data[get.cell.meta.data('sector.index'),4]
circos.text(xlim, ylim, sector.name, cex = 0.4, niceFacing = FALSE)
} )

# 第四圈,绘制富集因子
plot_data <- dat[c('id', 'gene_num.min', 'gene_num.max', 'rich.factor')]
label_data <- dat['category']
color_assign <- c('BP' = '#F7CC13', 'CC' = '#954572', 'MF' = '#0796E0')#各二级分类的名称和颜色
circos.genomicTrack(
plot_data, ylim = c(0, 1), track.height = 0.3, bg.col = 'gray95', bg.border = NA,
panel.fun = function(region, value, ...) {
sector.name = get.cell.meta.data('sector.index')
circos.genomicRect(region, value, col = color_assign[label_data[sector.name,1]], border = NA, ytop.column = 1, ybottom = 0, ...)
circos.lines(c(0, max(region)), c(0.5, 0.5), col = 'gray', lwd = 0.3)
} )
category_legend <- Legend(
labels = c('BP', 'CC', 'MF'),#各二级分类的名称
type = 'points', pch = NA, background = c('#F7CC13', '#954572', '#0796E0'), #各二级分类的颜色
labels_gp = gpar(fontsize = 8), grid_height = unit(0.5, 'cm'), grid_width = unit(0.5, 'cm'))
updown_legend <- Legend(
labels = c('Up-regulated', 'Down-regulated'),
type = 'points', pch = NA, background = c('red', 'blue'),
labels_gp = gpar(fontsize = 8), grid_height = unit(0.5, 'cm'), grid_width = unit(0.5, 'cm'))
pvalue_legend <- Legend(
col_fun = colorRamp2(round(seq(0, p_max, length.out = 6), 0),
colorRampPalette(c('#FF906F', '#861D30'))(6)),
legend_height = unit(3, 'cm'), labels_gp = gpar(fontsize = 8),
title_gp = gpar(fontsize = 9), title_position = 'topleft', title = '-Log10(Pvalue)')
lgd_list_vertical <- packLegend(category_legend, updown_legend, pvalue_legend)
grid.draw(lgd_list_vertical)
circos.clear()

往期优秀图形目录













以上内容仅为群内部分内容,不代表全部。详细目录请看下方列表!
























 91
91

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








