本次教程的figure仍然是读者求助的图,算得上是kegg富集图的新流派。据我的调查,该图应该是基迪奥云平台首创(https://www.omicshare.com/tools/Home/Soft/enrich_circle),之后公众号小白鱼的生统笔记进行了复现(仿一个网图,使用circlize包绘制圈图可视化基因集富集分析结果)。
最开始也是跟着上述的帖子学习,之后自己对代码进行了改写,重新安排图形的布局,使之(在我看来)更有意义。另一个改动是增加了kegg pathway的注释信息,我在之前的帖子中也提到了如何获取这个信息,没有这个信息是画不了这张图的。最后,将代码模块化,「一行代码」就能出图。
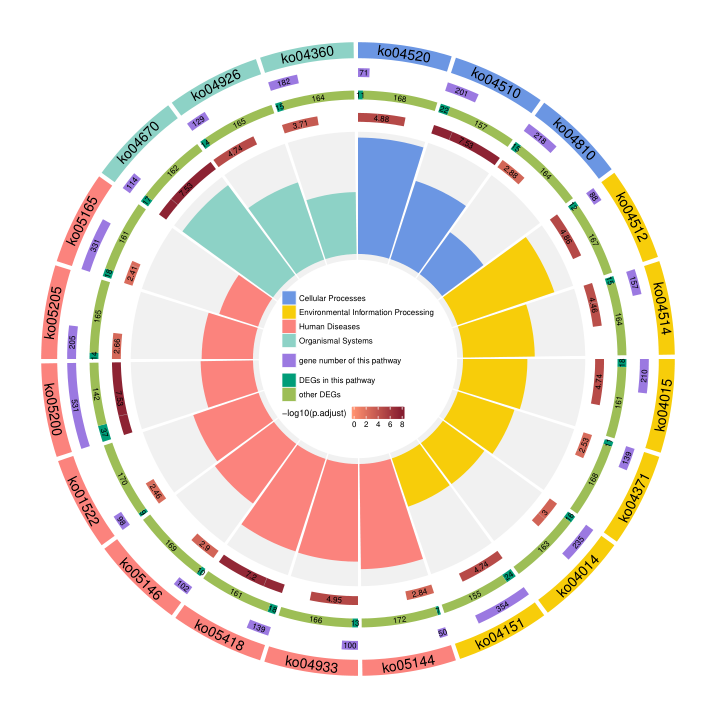
这张图如何解读:
-
不论是 组间比较还是cluster之间的比较,在得到某组/某cluster的差异基因后,都能进行KEGG富集分析,选取20-35个term画图 -
圆圈 从外向内看,第1圈是通路编号和分类,具体编号对应什么通路名称可以在代码输出的Excel文件中查询; -
第2圈表示这个通路有多少个基因; -
第3圈分为深绿和浅绿,二者加和始终是一样的,表示高表达基因的数目,深绿表示其中有多少基因属于这个通路,浅绿是不属于这个通路的基因数目; -
第4圈是富集分析的显著性,-log10(p.adjust),有一条灰色的竖线表示-log10(0.01); -
第5圈是富集因子,等于差异基因中落到这个通路的基因数除以这个通路的基因总数(第三圈深绿除以第二圈紫色)
代码演示
library(Seurat)
library(tidyverse)
library(xlsx)
testseu=readRDS("testseu.rds")
Idents(testseu)="anno_new"
### 找差异基因 #########################################################################
marker_celltype=FindAllMarkers(testseu,logfc.threshold = 0.8,only.pos = T)
# 过滤
marker_celltype=marker_celltype%>%filter(p_val_adj < 0.01)
marker_celltype$d=marker_celltype$pct.1-marker_celltype$pct.2
marker_celltype=marker_celltype%>%filter(d > 0.2)
marker_celltype=marker_celltype%>%arrange(cluster,desc(avg_log2FC))
marker_celltype=as.data.frame(marker_celltype)
write.xlsx(marker_celltype,file = "markers_log2fc0.8_padj0.01_pctd0.2.xlsx",row.names = F,col.names = T)
### 富集分析 ###########################################################################
library(clusterProfiler)
library(org.Hs.eg.db)
R.utils::setOption("clusterProfiler.download.method","auto") #https://github.com/YuLab-SMU/clusterProfiler/issues/256
source("syEnrich.R")
syEnrich(marker_celltype,outpath = "markers_log2fc0.8_padj0.01_pctd0.2")
### 挑一类细胞来作为演示 #######################################################
kegg.res=read.xlsx("markers_log2fc0.8_padj0.01_pctd0.2.KEGG.xls",sheetIndex = 1,as.data.frame = T,header = T)
kegg.res=kegg.res%>%filter(p.adjust < 0.05)
kegg.res=kegg.res%>%filter(cluster == "Endothelial")
导入《KEGG通路的从属/注释信息如何获取》这一讲的文件
# 导入《KEGG通路的从属/注释信息如何获取》这一讲的文件
kegg_info=read.xlsx("kegg_info.xlsx",sheetIndex = 1,startRow = 3)
kegg_info=kegg_info[,c("ID","Pathway","big.annotion")]
# 合并两个表格
kegg.res$ID=str_replace(kegg.res$ID,"hsa","")
kegg.res=kegg.res%>%inner_join(kegg_info,by = "ID")
# 画图展示的term控制在20到35个
kegg.res=kegg.res%>%arrange(p.adjust)
kegg.res=head(kegg.res,20)
write.table(kegg.res,file = "kegg.res.txt",quote = F,sep = "\t",row.names = F,col.names = T)
write.xlsx(kegg.res,file = "kegg.res.xlsx",col.names = T,row.names = F)
调用画图函数
### 调用画图函数 ###############################################################
source("kegg_loop.R")
kegg_loop(enrich.res = kegg.res,filename = "Endothelial_kegg")
然后就能得到推文开头那张图了。
获取代码
本文代码「限时公开」,24小时内是免费的。超过这个时间如何获取,后台回复2022A可知。
代码和测试数据的网盘链接如下: 链接:https://pan.baidu.com/s/1O1QJN-wvTdYSUhFEmt85vg 提取码:szcj
说点别的
这个系列到今天一共更新了10篇帖子(9篇2022A,1篇2022B),前后4个月的跨度,算是更新很慢的了。一方面,学校的事情越来越多,写博客的时间越来越少;另一方面,这些原创度很高的绘图帖子需要我花很多精力整理总结。
创作不易,觉得代码有用的话,可以给个三/二/一连(文末点赞、分享、点下小广告)。
























 2467
2467











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








