一 背景知识
1 基因表达量的熵值计算

利用直方图:1 确定每个基因的表达值范围 2 将基因的表达值分为n组
p(xi)代表在区间xi内,该基因出现在该区间的频率。当n区域无穷大时,就成为了该基因的概率密度函数
一个基因的熵值越大,代表它的分布越随机
2 两个基因之间的互信息


互信息为零意味着表达值的联合分布所包含的信息不超过单独考虑的基因
假设两个基因之间的互信息越高,它们之间的生物学关系就越有可能。
3 相关性网络的构建
利用酿酒酵母在79个不同条件下(有丝分裂阶段、孢子形成阶段等)2467个基因的表达谱,计算两两基因之间的互信息值,之后选择一个互信息阈值( threshold mutual information,TMI),只有高于这个阈值的基因对之间连接一条边。
二 结果
1 互信息计算值的分布

从图中可以看到,互信息主要分布在0.7左右,而排列RNA表达测定的互信息值没有超过1.3的。
2
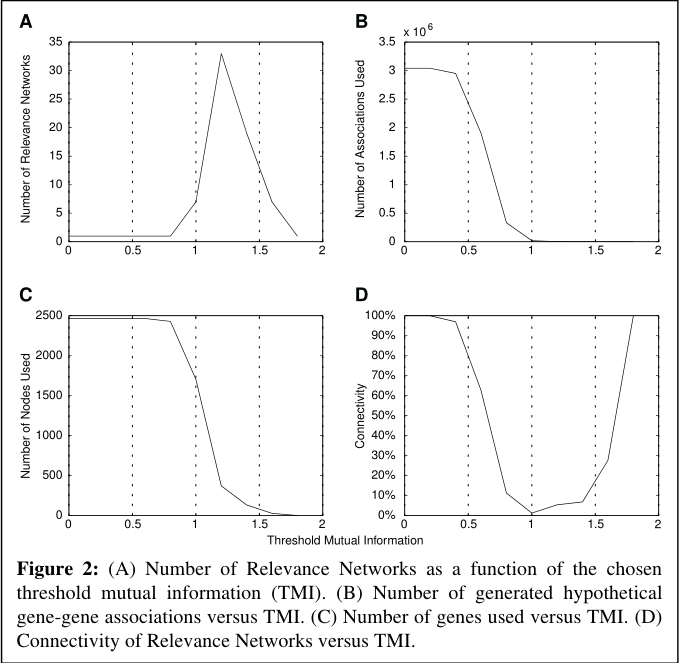
如图,当阈值从2下降到1.2时,相关性网络的数量渐渐增加,参与到网络中的基因也越来越多。
当阈值从1.2下降到0.8时,网络数量又呈现出减小趋势,这是因为新加入的节点连接了原本分开的两个网络
3
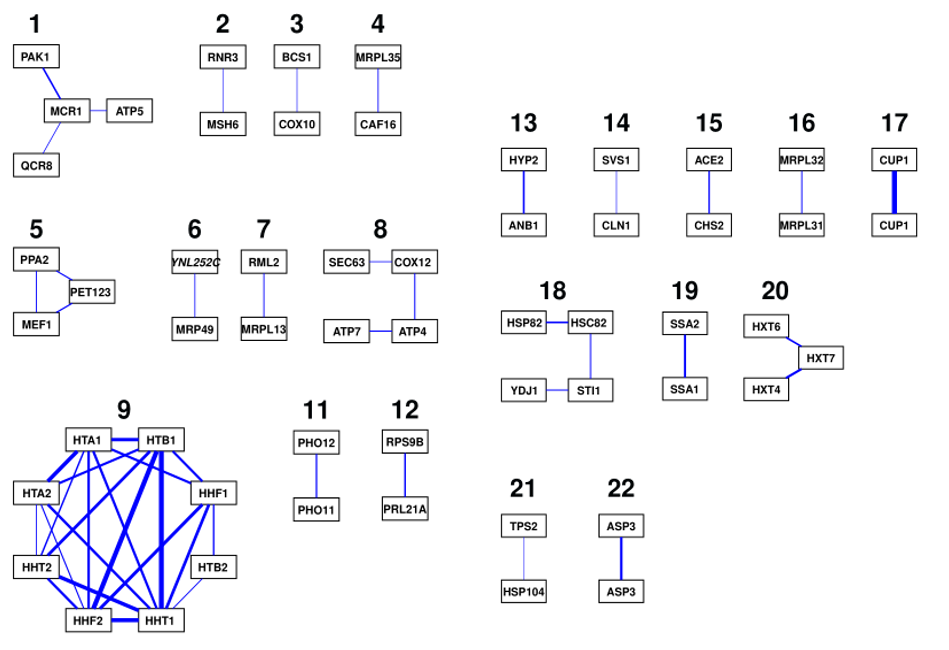
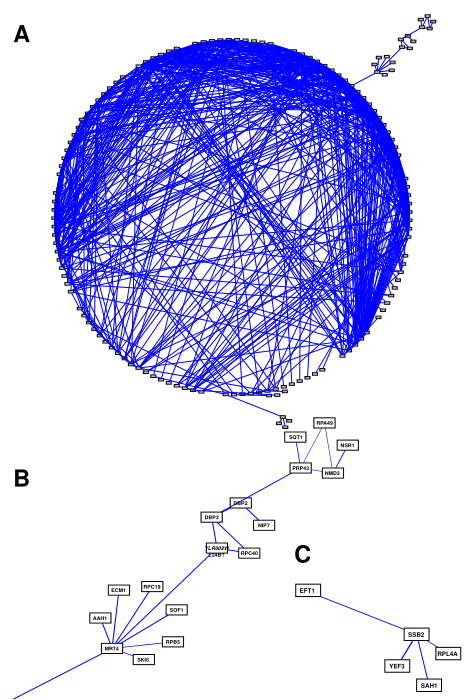
网络10
网络分析:(TMI为1.3)
①两个网络连着相同的两个基因(网络17和网络22)
②九个网络聚集了功能类似的基因 如网络9连接的8个基因都是编码组蛋白的,网络11连接的PHO12和PHO11,编码两种分泌的磷酸酶。(还包括网络12、13、19、20、6、7、16)
③五个网络中的基因参与到了相同的生物通路中,如网络2中的msh6,负责修复碱基对的错配;rnr3,诱发DNA损伤引起的反应。(还包括网络3,21,15,18)
三 讨论
相关性网络的优势:
①在描述基因之间的关系时,互信息比相关系数更普遍
因为相关性会受到离群点的影响,而互信息不会







 文章通过计算基因的熵值来评估其表达随机性,并通过互信息分析基因之间的关联程度。在酿酒酵母的数据集中,构建了基于互信息的相关性网络,发现网络数量和结构随阈值变化而变化。特定网络中发现了功能相关的基因聚类,例如编码组蛋白和参与生物通路的基因。互信息作为度量基因关系的方法,不受离群点影响,更具鲁棒性。
文章通过计算基因的熵值来评估其表达随机性,并通过互信息分析基因之间的关联程度。在酿酒酵母的数据集中,构建了基于互信息的相关性网络,发现网络数量和结构随阈值变化而变化。特定网络中发现了功能相关的基因聚类,例如编码组蛋白和参与生物通路的基因。互信息作为度量基因关系的方法,不受离群点影响,更具鲁棒性。
















 5967
5967

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








