一、 分子生态网络简介
分子生态网络分析是一个极具前景的群落生态分析方法,它能够较为轻松的探究出不同环境中的不同生物特征(物种或基因等)间的相互作用关系或共存模式。通过确定整个网络中的具有高连接度的微生物特征或该特征在模块内所处的位置,可以得到整个网络中的关键物种或基因(hub nodes)以及一些较为重要的物种或基因。这一类微生物特征可能对于微生物群落的结构和功能有着一定的决定作用。网络分析方法已经被广泛的应用于各个环境中的微生物群落的研究,在探寻微生物群落的特有属性、结构、功能以及群落的复杂性和稳定性等方面均有着重要意义,近来也被研究人员用于微生物生态理论的验证和群落应对环境扰动的响应研究。
分子生态网络分析包括两种类型:1)基于相关性的生态网络分析和2)以非随机共现C-scores分析为基础的网络。微生物生态基本都以相关性网络分析为主,通常用于生态系统组成要素矩阵中0不是特别多的数据,要求样品之间差距不能太大,一般用于多个重复样品数据构建网络。
相关性网络分析,需要确定阈值(一般是r和p值)以确定生物特征的相关性关系。确定的阈值可以是硬阈值或软阈值。
硬阈值:两个节点之间只存在有联系与无联系两种情况,得到二进制邻接矩阵,是unweighted networks。硬阈值可以是人为设定相关性系数r和p值。比如r的绝对值=0.6,p<0.05。但是人为设定阈值有一些太武断。有研究人员开发了很多算法,帮助进行阈值的筛选,比如基于 random matrix theory(随机矩阵理论,RMT)的方法构建网络。RMT理论假定了2种极端的最邻边空间分布的矩阵特征值的分布:Gaussian orthogonal ensemble (GOE)分布;Poisson分布。前者与随机复杂系统相对应,后者适用于某种特定的系统。通过改变阈值,得到不同阈值对应的邻接矩阵,计算其邻接矩阵的特征值,如果特征值分布近似于Poisson分布则认为这个阈值是合理的,近似于Gaussian orthogonal ensemble (GOE)分布则继续调整。最终得到的阈值便认为是合理的构建网络的阈值。MENAP网站(http://ieg4.rccc.ou.edu/mena/login.cgi )可以实现上述方法。还有其他的方法推荐阅读宏基因组的https://mp.weixin.qq.com/s/YPZar_aDmKE4W9s45NvhgQ文章。
软阈值:用一个权重值表示两个节点之间的相关程度,得到有权重的网络边,是weighted network。sigmoid函数和power函数可用于实现软阈值。
以非随机共现C-scores分析为基础的网络,用于分析生物特征存在/缺失矩阵,不考虑其具体的数值大小。如果在某一个样品中不存在,则用0表示,如果存在则用1表示。这样的分析适用于样品之间差距较大的数据。生物特征之间共现的关系常用C-scores值来衡量。通过构建零模型(null model),比较实验得到的观测值与随机值之间的差异,保留那些显著与随机不同的点与点之间的关系。
二、 数据准备
2.1 读入数据
使用的是原始otu和物种分类单元注释数据,需要合并具有相同注释结果的OTU。
# knitr::opts_knit$set(root.dir="D:\\network")# Rmarkdown工作目录设置方法
setwd("D:\\network")
options(stringsAsFactors=F)# R中环境变量设置,防止字符型变量转换为因子
library(ggraph)
library(igraph)
library(tidyverse)
library(wesanderson)
library(rstatix)
library(psych)
# 2.1 读入OTU丰度和分类单元数据
otutab <- read.csv("otu.csv",header = TRUE, row.names = 1)
otutax <- read.csv("taxonomy.csv",header = TRUE, row.names = 1)
head(otutab)
head(otutax)

图1|OTU丰度和样本分组数据,otutab。行为样本,列为变量。

图2|OTU分类单元注释数据,otutax。行为OTU ID,列为分类单元名。
2.2 合并注释为相同物种的otu
# 2.2 根据物种注释信息,合并注释为相同物种的otu
## 2.2.1 按各级分类单元合并otu数据表
#format2lefse()可以一次性执行界到属的物种数据合并,输出结果是相对丰度数据。
#修改原函数,也可以执行界到物种水平的数据合并,并输出绝对丰度。
library(amplicon) #
otu <- data.frame(t(otutab[3:ncol(otutab)]))
Group <- data.frame(name = rownames(otutab),otutab[1:2])
Group
### 按各级分类单元合并otu数据表-输出的各分类单元的绝对丰度数据。
#suppressWarnings(suppressMessages(library(amplicon)))
#format2lefse(otu, spetax, Group, thre, groupID,output = "LEfSe.txt")
source("format2lefse2.R")
format2lefse2(otutab = otu,
taxonomy = otutax,
metadata = Group,
Relative = FALSE, # 输出绝对丰度。
thre = 0, # 样本平均丰度筛选阈值,后续会对数据进行筛选,此处不进行筛选。
groupID = "name",#样本分组列名
output = "LEfSe.txt") # 输出LEfSe.txt文件到工作目录中。

图3|Group。
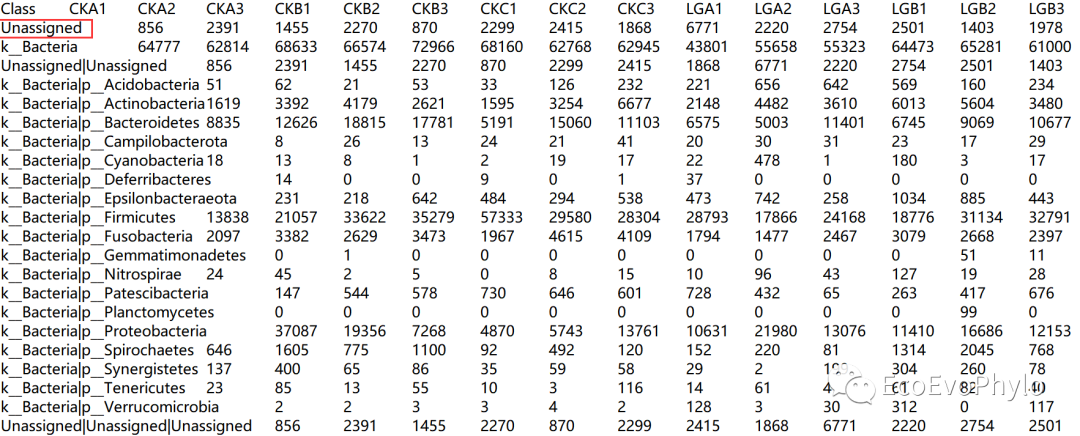
图4|OTU注释合并文件,LEfSe.txt。
## 2.2.2 重新读入LEfSe.txt,只保留其中包含所有分类单元信息的行
spetax <- read.table("LEfSe.txt",sep = "\t",header = TRUE)
spetax <- spetax[grep("s__",spetax$Class,fixed = TRUE),]
dim(spetax) # 477个物种
library(tidyr)
spetax <- tidyr::separate(spetax,col="Class",into=c(colnames(otutax)),sep = '\\|') # 特殊字符需要转义。
## 2.2.3 检查物种名是否具有重复值
#library(dplyr)
#duplicate_name <- spetax %>% group_by(Species) %>% summarise(freq = n()) %>% filter(freq > 1) %>% select(Species)
#duplicate_name # 使用duplicated()函数也可查看重复物种名
spetax$Species[duplicated(spetax$Species)] # 查看数据中是否还有重复物种名。
#spetax[duplicated(spetax$Species)==TRUE, ] # 查看数据中是否还有重复物种名,并返回最后一个重复值所在行
## 此处出现重复物种名的原因是,同一个科,却归属了2两个不同的目,需要手动核对。所以数据注释结果,也不是都可信的。
#spetax[spetax$Species == "s__uncultured_bacterium_f_Ruminococcaceae",]$Order <- c("o__Clostridiales","o__Clostridiales") # 结果显示因为没有明确的物种水平结果,科也相同,所以物种名重叠,为了不影响后续分析,对原始数据校正order名称。重新合并数据。
## 2.2.4 根据上述分析结果校正原始数据
otutax[otutax$Species == "s__uncultured_bacterium_f_Ruminococcaceae",]$Order <- "o__Clostridiales"
source("format2lefse2.R")
format2lefse2(otutab = otu,
taxonomy = otutax,
metadata = Group,
Relative = FALSE, # 输出绝对丰度。
thre = 0, # 样本平均丰度筛选阈值,后续会对数据进行筛选,此处不进行筛选。
groupID = "name",#样本分组列名
output = "LEfSe.txt")
spetax <- read.table("LEfSe.txt",sep = "\t",header = TRUE)
spetax <- spetax[grep("s__",spetax$Class,fixed = TRUE),]
dim(spetax) # 476个物种
library(tidyr)
spetax <- tidyr::separate(spetax,col="Class",into=c(colnames(otutax)),sep = '\\|') # 特殊字符需要转义
head(spetax)
#注释结果有部分序列未注释到细菌界,提取出来的数据相对丰度之和就不是100%。
colSums(spetax[-c(1:7)])
#如果想要提取全部,则可将“Unassigned”行也提取出来。
#spetax <- read.table("LEfSe.txt",sep = "\t",header = TRUE)
#spetax <- spetax[grep("(s__|^Unassigned$)",spetax$Class),]
## 2.2.5 对物种按照样本平均丰度进行降序排列
rownames(spetax) <- paste("Sp",seq(1,nrow(spetax),1),sep = "") # 如果要对数据进行重新排序,便于后续合并,提取数据,拥有行名就十分重要。
spetax <- arrange(spetax,desc(rowMeans(spetax[8:ncol(spetax)])))# 绝对丰度均值降序排列
head(spetax)

图5|OTU注释合并结果,spetax。
## 2.2.6 如果确定只使用某个分类水平的注释信息,比如门,可以进行单个合并
#otu <- data.frame(t(otutab[3:ncol(otutab)]))
otu2 <- merge(otu,otutax["Phylum"],by="row.names",sort = FALSE)
row.names(otu2) <- otu2$Row.names
otu2 <- otu2[-1]
otu2 %>% group_by(Phylum) %>% summarise_all(sum) -> otu_phylum
otu_phylum
2.3 过滤数据
# 2.3 过滤数据
## 2.3.1 计算物种相对丰度
data <- spetax
data[8:ncol(data)] <- sweep(data[8:ncol(data)],2,colSums(data[8:ncol(data)]),'/')*100
data <- arrange(data,desc(rowMeans(data[8:ncol(data)])))# 相对丰度均值降序排列
#colSums(data[8:ncol(data)])
## 2.3.2 对物种数据进行过滤
### 方法1:过滤掉平均丰度低于0.01%的物种
id <- rowMeans(data[-c(1:7)]) > 0.01
data1 <- data[id,] # 可以使用相对丰度进行后续分析,保留374个物种
#data1 <- spetax[id,] # 可以使用绝对丰度进行后续分析,保留374个物种
dim(data1)
summary(colSums(data1[8:ncol(data1)])) # 剩余物种在每个样本中的相对丰度在98.06%~99.89%之间。
## 如果保留物种的总体相对丰度较低,则表明此数据中低丰度物种的占据的总体比例较大,可能不适合使用此过滤方法进行过滤,可以更换过滤方法,或后续对过滤掉的物种进行进一步分析。此数据剩余物种的总相对丰度占比较大,可以使用此方法过滤。

图7|方法1过滤结果,summary(colSums(data1[8:ncol(data1)])) 。
### 方法2:保留累积相对丰度>99%的物种
tmp <- data[8:ncol(data)] %>%
map(~cumsum(.)) %>% # 必须使用降序排列后的相对丰度数据表。
data.frame()
rownames(tmp) <- rownames(data)
tmp[tmp>99] <- 1 # 每个样本累积丰度>99%的物种值赋1。
data2 <- data[1:which(rowSums(tmp) == 36)[1],] # 相对丰度
summary(colSums(data2[8:ncol(data2)])) # 剩余物种的相对丰度在99.08%~99.98%之间。
#data2 <- spetax[rownames(tmp[1:which(rowSums(tmp) == 36)[1],]),] # 绝对丰度
dim(data2)
head(data2)
## 物种数量不太多,想先看整体数据的分析结果,不提前过滤数据也可以,可过滤相关性分析产生NA的物种。
## 此处使用相对丰度data2进行后续数据分析。

图8|方法2过滤结果,summary(colSums(data2[8:ncol(data2)]))。
三 确定相关关系
3.1 进行相关性分析
# 3.1 进行相关性分析,pearson系数需要数据满足正态分布,如果数据不满足,建议使用spearman系数。
cor <- corr.test(t(data2[8:ncol(data2)]),
use = "pairwise",
method="spearman",
adjust="fdr",
alpha=.05,
ci=FALSE) # ci=FALSE,不进行置信区间计算,数据量较大时,可以加快计算速度。
cor.r <- data.frame(cor$r) # 提取R值
cor.p <- data.frame(cor$p) # 提取p值3.2 设置阈值,确定相关性关系
# 3.2 确定相关性关系:保留r绝对值>=0.6且p<=0.05的物种间相关关系
## 3.2.1 将数据转换为long, format,并将r和p值合并为一张表
#后面绘制网络图需要节点和链接数据,在这一步可以完成格式整理
cor.r$node1 = rownames(cor.r) # 需要增加一个ID列,不然转换时,列名将发生变化。推荐不要使用数字进行命名。
cor.p$node1 = rownames(cor.p)
p = cor.p %>%
gather(key = "node2", value = "p", -node1) %>%
data.frame()
cor.data = cor.r %>%
gather(key = "node2", value = "r", -node1) %>%
data.frame() %>%
left_join(p,by=c("node1","node2"))
dim(cor.data)
head(cor.data)
## 3.2.2 数据过滤,保留p<=0.05且abs(r)>=0.6的变量间相关关系,并添加网络属性。
cor.data <- cor.data %>%
filter(abs(r) >=0.6, p <= 0.05, node1 != node2) %>%
mutate(
linecolor = ifelse(r > 0,"positive","negative"),
linesize = abs(r)
)# 此输出仍有重复链接,后面需进一步去除。
head(cor.data)
dim(cor.data)
图9|未删除重复链接的链接属性表,cor.data。
## 3.2.3 删除中间文件,节约空间
rm(cor,cor.r,cor.p)
gc()四 构建graph网络结构数据
4.1 网络图节点属性整理
# 4.1 网络图节点属性整理
## 4.1.1 计算每个节点具有的链接数
c(as.character(cor.data$node1),as.character(cor.data$node2)) %>%
as_tibble() %>%
group_by(value) %>%
summarize(n=n()) -> vertices
colnames(vertices) <- c("node", "n")
head(vertices)
## 4.1.2 添加节点的分类单元属性
vertices %>%
select(-n) %>% # 因为此处的n不准确,所以先删除。
merge(data2[1:7],by.x = "node",by.y = "row.names") -> vertices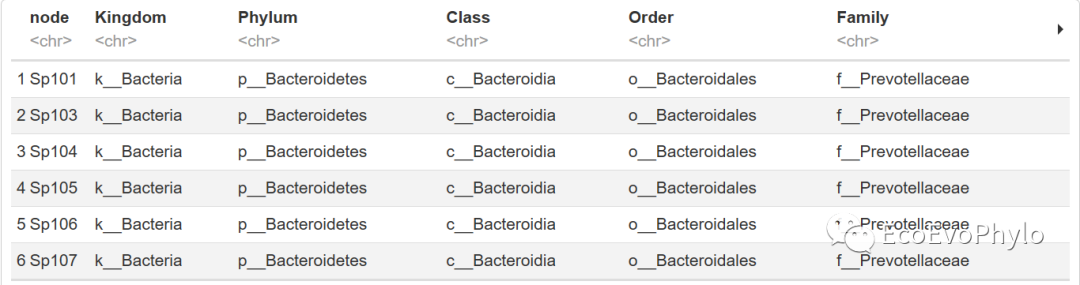
图10|包含分类单元信息的节点属性表,vertices 。
4.2 构建graph数据结构
# 4.2 构建graph数据结构
## 4.2.1 使用igraph包的graph_from_data_frame()形成graph结构数据
#?graph_from_data_frame # 查看函数帮助信息
graph = igraph::graph_from_data_frame(cor.data,directed = FALSE,vertices = vertices) # 数据框前两列要求是链接对应节点信息,directed = FALSE表示无向网络
graph # graph中包含的节点、链接及两者属性信息。v表示链接,n表示节点。

图11|非简单图数据结构,graph。
## 4.2.2 简单图:去除自身链接形成的loop和两两节点间的multiple edges
is.simple(graph)
graph <- igraph::simplify(graph,
remove.multiple = TRUE,
remove.loops = TRUE,
edge.attr.comb = "first")
#graph <- delete.vertices(graph,which(degree(g) == 0)) # 删除孤立点,适用于直接从r矩阵构建的graph。
is.simple(graph)
E(graph)$weight <- 1
is.weighted(graph)
get.edge.attribute(graph)
get.vertex.attribute(graph)
vcount(graph) # 279
ecount(graph) # 977
图12|简单图数据结构,graph。
4.3 网络拓扑属性计算
# 4.3 网络拓扑属性计算
## 4.3.1 节点属性计算
### degree
V(graph)$degree=degree(graph) # degree:每个节点具有的链接数量
### 节点中心性-使用连通图计算更准确
#### 邻近中心性(Closeness centrality)
V(graph)$closeness_centrality = closeness(graph,weights = NA)
#### 介数中心性(Betweenness centrality)
V(graph)$betweenness_centrality = betweenness(graph,weights = NA)
#### 特征向量中心性(Eigenvector centrality)
V(graph)$eigenvector_centrality = evcent(graph,weights = NA)$vector
#### clustering coefficient,聚类系数
V(graph)$transitivity = transitivity(graph,type="local",weights=NA)
### 网络内聚力-Network Cohesion
#### 子图
#table(sapply(cliques(graph), length)) # cliques是完整的子图,是完全内聚的顶点的子集
#clique.number(graph) #11个内聚子集
V(graph)$coreness=graph.coreness(graph)
### hub and authority score
V(graph)$hub.score=hub.score(graph)$vector
V(graph)$authority.score=authority.score(graph)$vector
## 4.3.2 链接属性
### Edge betweenness centrality:通过为每条边分配一个反映穿过该边的最短路径数的值
E(graph)$betweenness_centrality = edge.betweenness(graph)
E(graph)$betweenness_centrality %>%
head(n=10)
## 4.3.3 网络整体属性
### 网络整体属性指数
graph$name = "Network Graph" # 赋予网络图名称
graph$diameter=diameter(graph, weights=NA)
graph$diameter# 节点间geodesic distance的最长路径
graph$density=graph.density(graph)
graph$density # 实际的链接数相对于所有节点都相连的频率
graph$transitivity=transitivity(graph,weights=NA)
graph$transitivity# 节点的邻近节点是连通的比例,表明接近于形成三角形的连通三角形比例。
graph$average.path.length=average.path.length(graph,weights=NA)
graph$average.path.length# 从网络中的顶点到顶点的所有最短路径的长度的平均值。
## 网络中关键节点和链接:随机移除某些节点/边,剩余的子图是否连通?
vertex.connectivity(graph) # 移除1个节点或边,剩余子图就不再连通
edge.connectivity(graph)
## vertex-cut (edge-cut):移除图中的一组特定顶点(边)断开了该图的连接,移除的节点称为cut vertex或articulation point
graph$cut.vertices = articulation.points(graph) # cut vertex的名称
length(graph$cut.vertices)# cut vertex的数量:44
## assortativity coefficients:量化给定网络中分类混合程度的度量被称为分类系数,本质上是相关系数概念的变体。
###基于一些顶点标记或分配给顶点的值来衡量图形的同质性水平,该系数较高,则意味着连接的顶点往往具有相同的标签或相似的赋值。
####如果相似的顶点(基于某些外部属性)倾向于彼此连接assortativity coefficients为正,否则为负。
# type必须是整数,所以需要+1
assortativity.nominal(graph,type=(V(graph)$Phylum=="p__Bacteroidetes")+1, directed = FALSE) #0.0724908
### 使用degree作为节点属性计算assortativity:分类系数r的一个常见用法是在研究网络结构时总结相邻顶点的度-度相关性。
graph$assortativity.degree <- assortativity.degree(graph,directed = FALSE)
graph$assortativity.degree # 0.2558154
## 4.3.4 图分区(community detection)
#将一组元素分割成“自然”子集,显示出相对于底层关系模式的“内聚性(cohesiveness)”。
#igraph包含3种图分区算法:
#1)Hierarchical Clustering:cluster_fast_greedy();
#2)Walktrap community finding algorithm:cluster_walktrap()用与查找紧密链接的子图,短的随机步长(默认步长为4)往往停留在同一个群体中。寻找到的子图比cluster_fast_greedy多;
#3)spin-glass model and simulated annealing:cluster_spinglass()。
### cluster_walktrap()通过随机游动在图中找到密集链接的子图(群体),相当于网络的模块性分析。
#根据链接的紧密合作关系,分为不同群体,同一群体内的节点关系紧密,不同群体间的节点关系松散。
graph$community=cluster_fast_greedy(graph)
graph$community #层次聚类分为16组。
length(graph$community)
sizes(graph$community)
### 对每个群体中包含的节点进行统计,按节点数量进行排序,节点数量最多的群体序号为1,并添加到节点属性中。
graph$community$membership <- as.numeric(
factor(graph$community$membership,
levels=sort(summary(as.factor(graph$community$membership)),
index.return=TRUE,
decreasing = T)$ix,
order=TRUE))
length(graph$community)
sizes(graph$community)
V(graph)$modularity = membership(graph$community) # 将每个节点的community信息,添加到节点属性。
图13|重新排序后图分区序号及包含的节点数,sizes(graph$community)。
### 模块指数计算
graph$modularity <- modularity(graph$community) # 网络的模块指数:0.6982418
#modularity(graph, membership(graph$community)) # 计算结果一致
### 验证图分区-基于门分类单元信息
#vertex_attr_names(graph)
Phylum=data.frame(table(membership(graph$community), # 提取节点及其对应分区信息
vertex_attr(graph, "Phylum"),
useNA=c("no")))
Phylum %>% # 得到每个门分类单元在每个分区中存在的数量。
sample_n_by(Var1,size=5)

图14|每个门分类单元在每个图分区中存在的数量,Phylum。
4.4 保存graph到本地
# 4.4 graph保存到本地
## 4.4.1 保存整个graph数据结构
write.graph(graph,file = "graph.gml",format="gml") # gml能保存的graph信息最多,但会丢失计算的网络整体拓扑属性。
## 4.4.2 分别提取节点和链接信息保存
##后面修改节点或链接属性可以直接下面文件中修改,然后再重新生成graph数据结构。
net.data <- igraph::as_data_frame(graph, what = "both")$edges # 提取链接属性
write.csv(net.data,"net.data.csv",quote = FALSE,col.names = NA,row.names = FALSE) # 保存结果到本地。
head(net.data)
vertices <- igraph::as_data_frame(graph, what = "both")$vertices # 提取节点属性
write.csv(vertices,"vertices.csv",quote = FALSE,col.names = NA,row.names = FALSE)
head(vertices)
图15|最终链接属性表,net.data。

图16|最终节点属性表,vertices。
五、绘制网络图
网络数据是指不具备层次结构的关系数据,其不具备包含关系。网络数据通常用图(graph)结构表示,包含nodes(Vertices,节点或定点)和edges(边或链接)两个基础数据集合,还可以包含节点和链接的属性数据。根据链接方向有无和有无权重,网络关系可以分为4种类型:1)有向且有权重;2)有向无权重;3)无向且有权重;4)无向无权重。分子生态相关性网络中特征间的相关性系数是有大小的(有权重),但是相关性关系不是因果关系(无向),因此属于无向无权重网络关系。上面整理的节点和链接数据表已经可以用于Cytoscape或R绘制网络图。绘制网络图过程中,设置节点与链接在画布上的布局方式,还需要一个layout(布局)参数。
网络图的布局有很多种,可以多尝试几种布局方式,找到最适合自己数据的布局。R中有很多可用于graph分析绘图的R包,sna,network,igraph和ggraph等。下面使用ggraph进行网络图可视化。
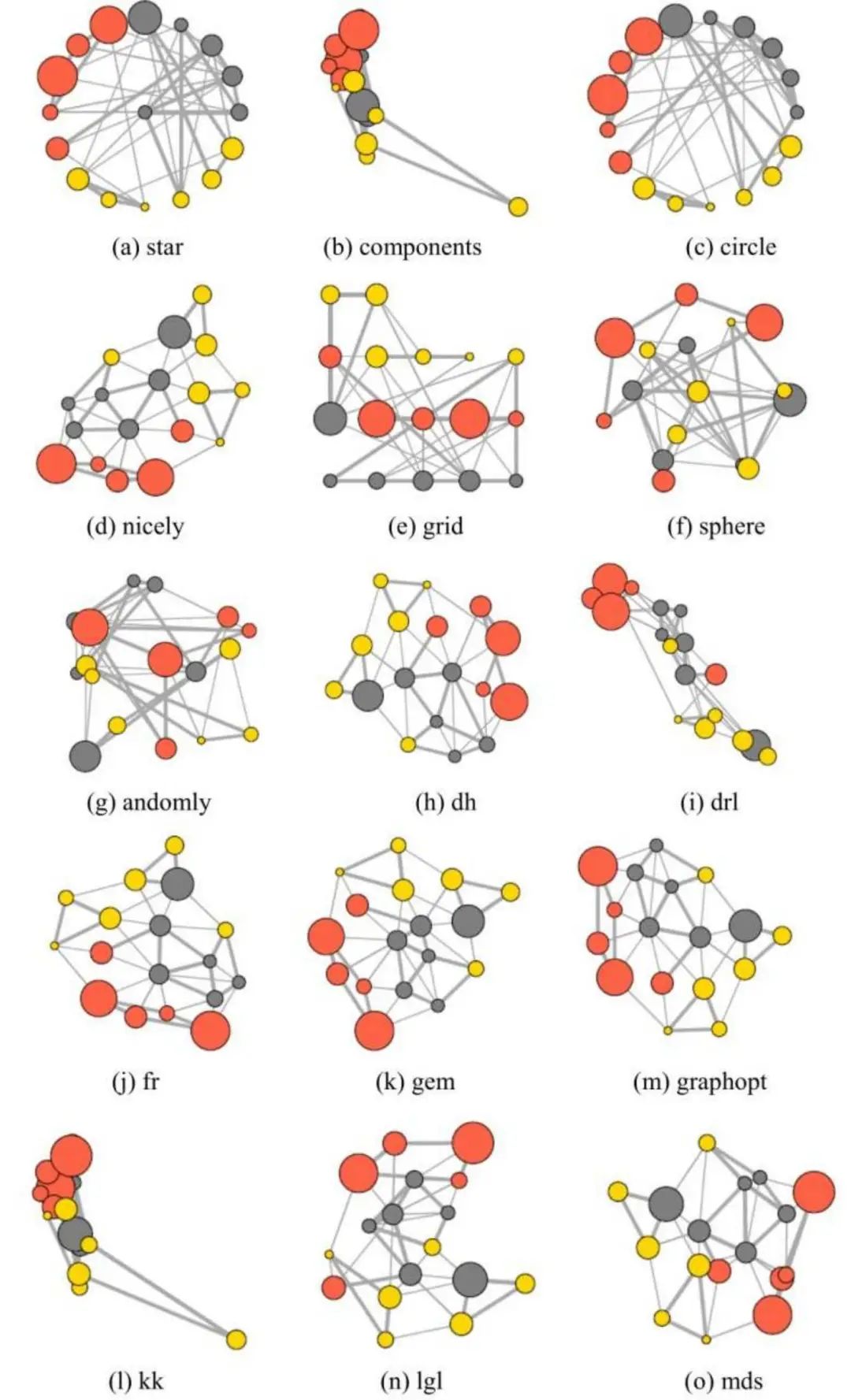
图17|网络图布局方式。来自《R语言数据可视化之美》。绘图时可以尝试多种布局,找寻适合自己数据的布局。
5.1 径向布局网络图
# 5.1.1 设置绘图颜色
#?wes_palette # 看函数帮助信息
#只对包含节点数≥10的分区设置颜色,包含节点数<10的分区,设置为灰色。
### 之前想着按某个分类单元设置颜色,所以挑选了很多颜色,颜色根据自己的需要自行设置即可。
#length(unique(V(graph)$Genus))
library(ggsci)
library(scales)
## 节点颜色
#mycolor <- wes_palette("Darjeeling1", local, type = "continuous")
#mycolor = pal_d3("category10")(10)
mycolor = pal_rickandmorty()(10)
mycolor
local <- max(which(table(V(graph)$modularity) >= 10)) # top6群体包含节点数≥10。
mycolor <- c(mycolor[1:local],
rep("grey50",max(V(graph)$modularity)-local)
)
mycolor
## 链接线颜色-根据相关性系数正负设置
color = c("positive" ="red", "negative" = rgb(0,147,0,maxColorValue = 255))
# 5.1.2 添加标签角度数据
## 将节点按照分区升序,degree降序排列
vertices <- vertices %>%
arrange(modularity, desc(degree))
## seq()生成一组1-数据表行数的数值。
number_of_bar <- nrow(vertices)
vertices$id <- seq(1, nrow(vertices))
angle= 360 * (vertices$id-0.5) /number_of_bar # 形成每个标签放置位置数据,字母中心排列,所以需要-0.5。
vertices$hjust<-ifelse(angle>180, 1, 0)
vertices$angle<-ifelse(angle>180, 90-angle+180, 90-angle)
## 重新构建graph数据结构
graph <- graph_from_data_frame(net.data, vertices = vertices, directed = FALSE )
graph # 重新生成的graph会丢失前面计算的graph整体拓扑属性。
图18|graph结构数据内容。增加了节点标签角度和水平对齐方式两个属性。
# 5.1.3 绘制径向布局网络图,ggraph()布局参数设置为layout = 'linear', circular = TRUE即可。
## geom_edge_arc()设置链接线为曲线, geom_edge_link()设置链接线为直线。按相关性系数正负对链接线进行着色。
## geom_node_point()设置节点,按链接数量设置节点大小,按群体设置填充颜色。
## scale_size_continuous()设置点的大小范围,根据节点链接数范围修改。
## scale_fill_manual()设置绘图颜色的选择范围。
## geom_node_text()设置节点标签,标签以物种名显示,字体颜色按群体着色。
## ggraph通过添加图层绘图,后添加的图形会出现在最上层,所以先绘制链接线,再绘制节点。
net.cir <- ggraph(graph,
layout = 'linear',
circular = TRUE) +
##设置链接属性##
geom_edge_arc(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.5 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(x = x*1.06,
y = y*1.06,
label=ifelse(degree/sum(degree)>0.01, as.character(Species), ""),
angle=angle,hjust=hjust,
color=as.factor(modularity)),size=3,
show.legend = FALSE) +
scale_color_manual(values = mycolor)+
expand_limits(x = c(-1.6, 1.6), y = c(-1.6, 1.6))+
coord_fixed()+
theme_minimal() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank(),
plot.margin=unit(c(0,0,0,0), "null"),
panel.spacing=unit(c(0,0,0,0), "null")
) # 只显示链接数占总链接1%以上的节点的物种名标签
# 5.1.4 输出pdf格式图片到本地
ggsave(net.cir,filename="net.cir.pdf",device="pdf",height=10,width=14)
net.cir # 径向布局不适合变量较多的数据。
图19|径向网络图。不适合变量较多的数据。
5.2 MDS(多尺度分析布局)网络图
# 5.2.1 MDS(多尺度分析布局),设置layout='mds'
net.MDS <- ggraph(graph,layout='mds') +
##设置链接属性##
geom_edge_arc(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_minimal() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank())
# 只显示链接数占总链接10%以上的节点的物种名标签,标签为黑色,没有则不显示。
# 5.2.2 输出pdf格式图片到本地
ggsave(net.MDS,filename="net.MDS.pdf",device="pdf",height=8,width=8)
net.MDS
图20|MDS(多尺度分析布局)网络图。
5.3 FR布局网络图
Fruchterman-Reingold 算法的力导向布局。
# 5.3.1 Fruchterman-Reingold 算法的力导向布局,设置layout='fr'
net.fr <- ggraph(graph,layout='fr') +
##设置链接属性##
geom_edge_link(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_bw() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
#plot.margin=unit(c(0,0,0,0), "null"),
#panel.spacing=unit(c(0,0,0,0), "null")
) # 只显示链接数占总链接10%以上的节点的物种名标签,标签字体为黑色
# 5.3.2 输出pdf格式图片到本地
ggsave(net.fr,filename="net.fr.pdf",device="pdf",height=10,width=10)
net.fr

图21|Fruchterman-Reingold 算法的力导向布局网络图。
5.4 nicely布局网络图
# 5.4.1 nicely(简单决策树simpe decision tree),设置layout='nicely'
## guide="none",不设置点大小图例。
set.seed(1234) # 设置随机种子,保证绘图布局的可重复性
net.nicely <- ggraph(graph,layout='nicely') +
##设置链接属性##
geom_edge_arc(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_bw() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
) +
guides(fill = guide_legend(title = "Community", title.position = "top",
title.theme = element_text(
size = 15,
colour = "black")))# 只显示链接数占总链接10%以上的节点的物种名标签,标签为红色
# 5.4.2 输出pdf格式图片到本地
ggsave(net.nicely,filename="net.nicely.pdf",device="pdf",height=10,width=10)
net.nicely

图22|nicely布局网络图。网络具有明显的模块。
5.5 drl布局网络图
# 5.5.1 drl布局,设置layout='drl'
## guide="none",不设置点大小图例。
set.seed(1234) # 设置随机种子,保证绘图布局的可重复性
net.drl <- ggraph(graph,layout='drl') +
##设置链接属性##
geom_edge_link(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_bw() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
) +
guides(fill = guide_legend(title = "Community", title.position = "top",
title.theme = element_text(
size = 15,
colour = "black")))# 只显示链接数占总链接10%以上的节点的物种名标签,标签为红色
# 5.5.2 输出pdf格式图片到本地
ggsave(net.drl,filename="net.drl.pdf",device="pdf",height=10,width=10)
net.drl

图23|drl布局网络图。
5.6 kk布局网络图
# 5.6.1 kk布局,设置layout='kk'
## guide="none",不设置点大小图例。
set.seed(1234) # 设置随机种子,保证绘图布局的可重复性
net.kk <- ggraph(graph,layout='kk') +
##设置链接属性##
geom_edge_link(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_bw() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
) +
guides(fill = guide_legend(title = "Community", title.position = "top",
title.theme = element_text(
size = 15,
colour = "black")))# 只显示链接数占总链接10%以上的节点的物种名标签,标签为红色
# 5.6.2 输出pdf格式图片到本地
ggsave(net.kk,filename="net.kk.pdf",device="pdf",height=10,width=10)
net.kk

图24|kk布局网络图。
5.7 graphopt布局网络图
# 5.7.1 graphopt布局,设置layout='graphopt'
## guide="none",不设置点大小图例。
set.seed(1234) # 设置随机种子,保证绘图布局的可重复性
net.graphopt <- ggraph(graph,layout='graphopt') +
##设置链接属性##
geom_edge_arc(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 16),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-1.2, 1.2))+
theme_bw() +
theme(
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
) +
guides(fill = guide_legend(title = "Community", title.position = "top",
title.theme = element_text(
size = 15,
colour = "black")))# 只显示链接数占总链接10%以上的节点的物种名标签,标签为红色
# 5.7.2 输出pdf格式图片到本地
ggsave(net.graphopt,filename="net.graphopt.pdf",device="pdf",height=10,width=10)
net.graphopt

图25|graphopt布局网络图
5.8 弧长链接图
绘图时设置layout = 'linear',circular = FALSE;再将链接线使用曲线链接函数geom_edge_arc(),实现弧线的连接。此数据不适合用弧长链接图,此处只用于举例。
# 5.8.1 绘制弧长链接图
pdf("弧长链接图.pdf",width = 12,height = 8,family="Times")
set.seed(1234)
ggraph(graph, layout="linear",circular=FALSE) +
##设置链接属性##
geom_edge_arc(aes(edge_colour = as.factor(linecolor),
edge_width = linesize
),
edge_alpha=0.7 ) +
###theme()中的legend参数不利于多个图例设置,这里在scale*函数中直接设置图例参数###
scale_edge_colour_manual(values=color,
breaks = c("positive","negative"),
guide = guide_legend(title="Correlation",
direction="vertical",
order=2,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
)) +
scale_edge_width(
breaks = seq(0.2,1,0.2),
label = seq(0.2,1,0.2),
range = c(0.2,1),
guide = guide_legend(title="ABS(r)",
direction="vertical",
order=1,# 图例排列序号
ncol=1,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
##设置节点属性##
geom_node_point(aes(size=degree, fill=as.factor(modularity)),
shape=21,color='black',alpha=1) +
scale_size(
breaks = seq(5,max(vertices$degree),5),
label = seq(5,max(vertices$degree),5),
range = c(2, 9),
guide = guide_legend(title="Degree",
direction="vertical",
order=3,# 图例排列序号
ncol=2, # 图例排两列
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black")
))+
scale_fill_manual(values=mycolor,
guide = guide_legend(title="Community",
#keywidth=grid::unit(2,"cm"),
#keyheight=grid::unit(2,"cm"),
direction="vertical",
order=4,# 图例排列序号
ncol=2,
byrow=FALSE,
title.theme = element_text(
size = 14,
face = "bold",
colour = "black"),
label.theme = element_text(
size = 12,colour = "black",face = "italic")
)) +
##设置节点标签属性##
geom_node_text(aes(label=ifelse(degree/sum(degree)>0.1, as.character(Genus), "")),
size=3, color="black") +
expand_limits(x = c(-1.2, 1.2), y = c(-5.6, 1.2))+
theme_classic() +
theme(
#legend.position="none",
panel.grid = element_blank(),
axis.line = element_blank(),
axis.ticks =element_blank(),
axis.text =element_blank(),
axis.title = element_blank()
)
dev.off()

图26|弧长链接图。此数据不适合用弧长链接图,此处只用于举例。图形也不美观。
其实网络分析的资料在网上有很多,实现的方法也有很多。最重要的还是要了解基础理论,让自己分析数据的时候有参与感,而不是只是按照别人的教程依次运行。经过积累,都会逐渐进步的。网络构建完成后,提取完网络拓扑参数后,还可以对子图的网络拓扑参数进行统计分析,这部分内容之后再写。
原始数据和代码下载:链接:https://pan.baidu.com/s/1SGKFXhHsYFYHIQnent2Vig?pwd=78b5
提取码:78b5
参考资料
Mehdi Layeghifard et al. Disentangling Interactions in the Microbiome: A Network Perspective. Trends in Microbiology. 10.1016/j.tim.2016.11.008
Statistical Analysis of Network Data with R,Kolaczyk and Csárdi , 2014.
沈烽,黄睿,曾巾,等.分子生态网络分析研究进展[J].环境科学与技术,2016,39(S1)
《R数据可视化之美:专业图表绘制指南(增强版)》
推荐阅读
R绘图-物种、环境因子相关性网络图(简单图、提取子图、修改图布局参数、物种-环境因子分别成环径向网络图)
R统计-PCA/PCoA/db-RDA/NMDS/CA/CCA/DCA等排序分析教程
R统计绘图-环境因子相关性+mantel检验组合图(linkET包介绍1)
机器学习-分类随机森林分析(randomForest模型构建、参数调优、特征变量筛选、模型评估和基础理论等)
R统计-单因素ANOVA/Kruskal-Wallis置换检验
R统计绘图-单、双、三因素重复测量方差分析[Translation]
R统计绘图-多变量相关性散点矩阵图(GGally::ggpairs())

EcoEvoPhylo :主要分享微生物生态和phylogenomics的数据分析教程。扫描上方二维码,关注EcoEvoPhylo。让我们大家一起学习,互相交流,共同进步。
知乎 | 生态媛
学术交流QQ群 | 438942456
学术交流微信群 | jingmorensheng412
加好友时,请备注姓名-单位-研究方向。




























 1258
1258

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










