记录
如果我在已经有了基因名的情况下,可以有几种方法
1.通过NCBI的GENE数据库
1)首先进入到https://www.ncbi.nlm.nih.gov/gds/中,然后选择你要的基因

2)例TP53,需要观察物种
3)选择并点击,进入,ctrf + F,输入 NCBI Reference Sequences (RefSeq) ,查询这个基因所具有的转录本 ,结果如下
总结:这种方法适用于基因个数比较少的情况,当基因个数比较多时,则需要利用R包biomart进行转换,或者数据库资源
2. 数据库资源
在NCBI的数据库中下载,你所需要的部分。你可以在这个网址中https://ftp.ncbi.nlm.nih.gov/gene/DATA/,下载这个文件。
解压后打开如下,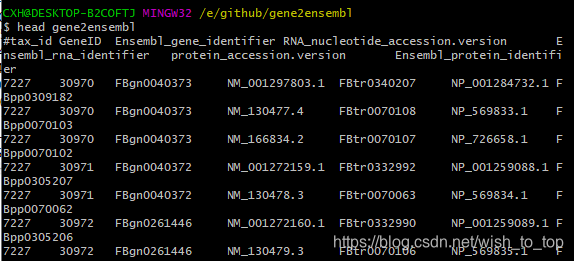
tax——id,为物种的ID,例如人类为9606,然后后面跟着的就算基因ID了,第四个为我们所需要的NM编号(RefSeq 的转录本号).当然了,如果你好需要基因ID所对应的基因symbol的,这边也可以下载。https://ftp.ncbi.nlm.nih.gov/gene/DATA/GENE_INFO/Mammalia/
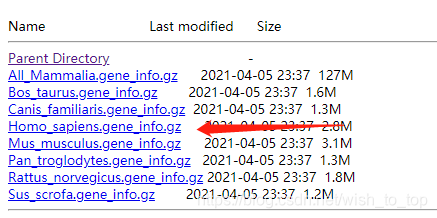
下了小鼠的,打开如下
第二列和第三列为基因ID和基因名。
有了这两张表,你基本上就能得到所有NM开头的所有基因转换关系了。你也可以合并这两份张表,通过R的merge或者excel来做vlookup都可以。
3.R包
R包可以参考这篇推文吧https://blog.csdn.net/weixin_40739969/article/details/89354167,既然都是基因转换,应该差不了多少。或者可以用biomart包
4.网站
DAVID吧。。。。
5.后续想到再说
6.在5月份处理小鼠的转录数据时碰到的问题
1.1 biomaRt
对于biomaRt这个包的转化函数为getbm,然后

看样子是要将dplyr降级了,但是实在是不想这么搞,因为不同的包可能到时候又要升级回去,所以算了算了。
1.2 clusterProfiler
gene.df <- bitr(gene, fromType = "ENSEMBL", #fromType是指你的数据ID类型是属于哪一类的
toType = "SYMBOL", #toType是指你要转换成哪种ID类型,可以写多种,也可以只写一种
OrgDb = org.Mmu.eg.db)#Orgdb是指对应的注释包是哪个
这个跟我一直报
找不到数据类型,oh,我的上帝呀。不想搞了,花了太多时间,从装包到比对,有解决方法的请指导下,谢谢。
1.3 AnnotationDbi
气急败坏的我又找了一篇,这次终于可以了
gene = data$Geneid# %>% as.data.frame()
df = data.frame(gene)
#BiocManager::install("AnnotationDbi")
library("AnnotationDbi")
df$symbol <- mapIds(org.Mm.eg.db,
keys=gene,
column="SYMBOL",
keytype="ENSEMBL",
multiVals="first")
df = na.omit(df) # 删除掉没有匹配掉的























 3174
3174

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








