AlphaFold是DeepMind公司下的一个预测蛋白质结构的AI,这个公司下的AI还有我们所熟悉的AlphaGo。它对大部分蛋白质结构的预测与真实结构只差一个原子的宽度,达到了人类利用冷冻电子显微镜等复杂仪器观察预测的水平。该程序利用Python写成。今天我们将利用它的云端Jupyter Notebook根据人的红细胞H抗原的氨基酸序列对人的红细胞H抗原的蛋白结构进行预测。
首先先打开NCBI找到相关氨基酸序列,把它copy下来
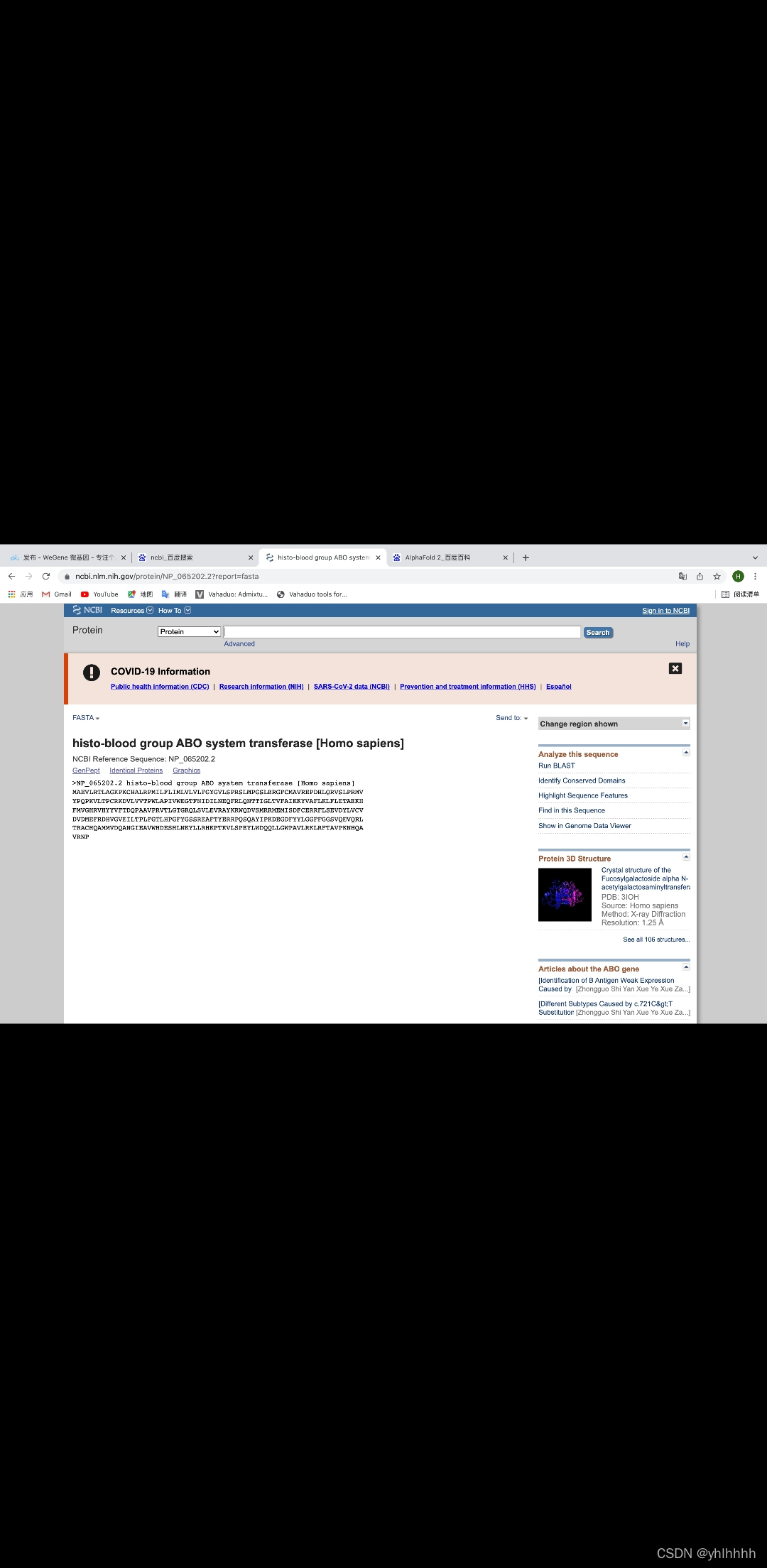
之后打开这个网址: https://colab.research.google.com/github/deepmind/alphafold/blob/main/notebooks/AlphaFold.ipynb
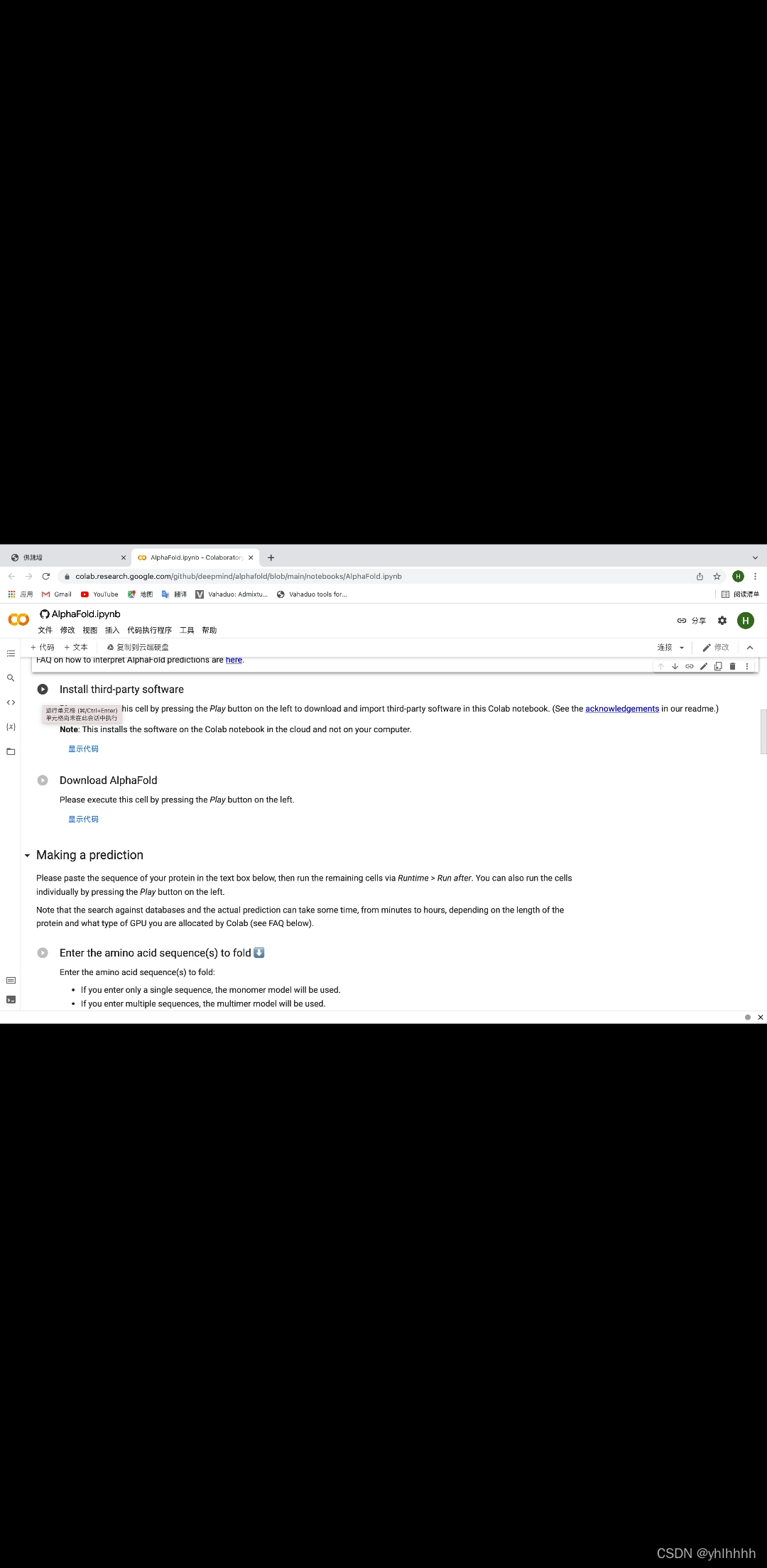
之后首先按顺序运行前两单元,安装运行环境(这个安装过程只会安装到云端,不会安装到本地,放心)
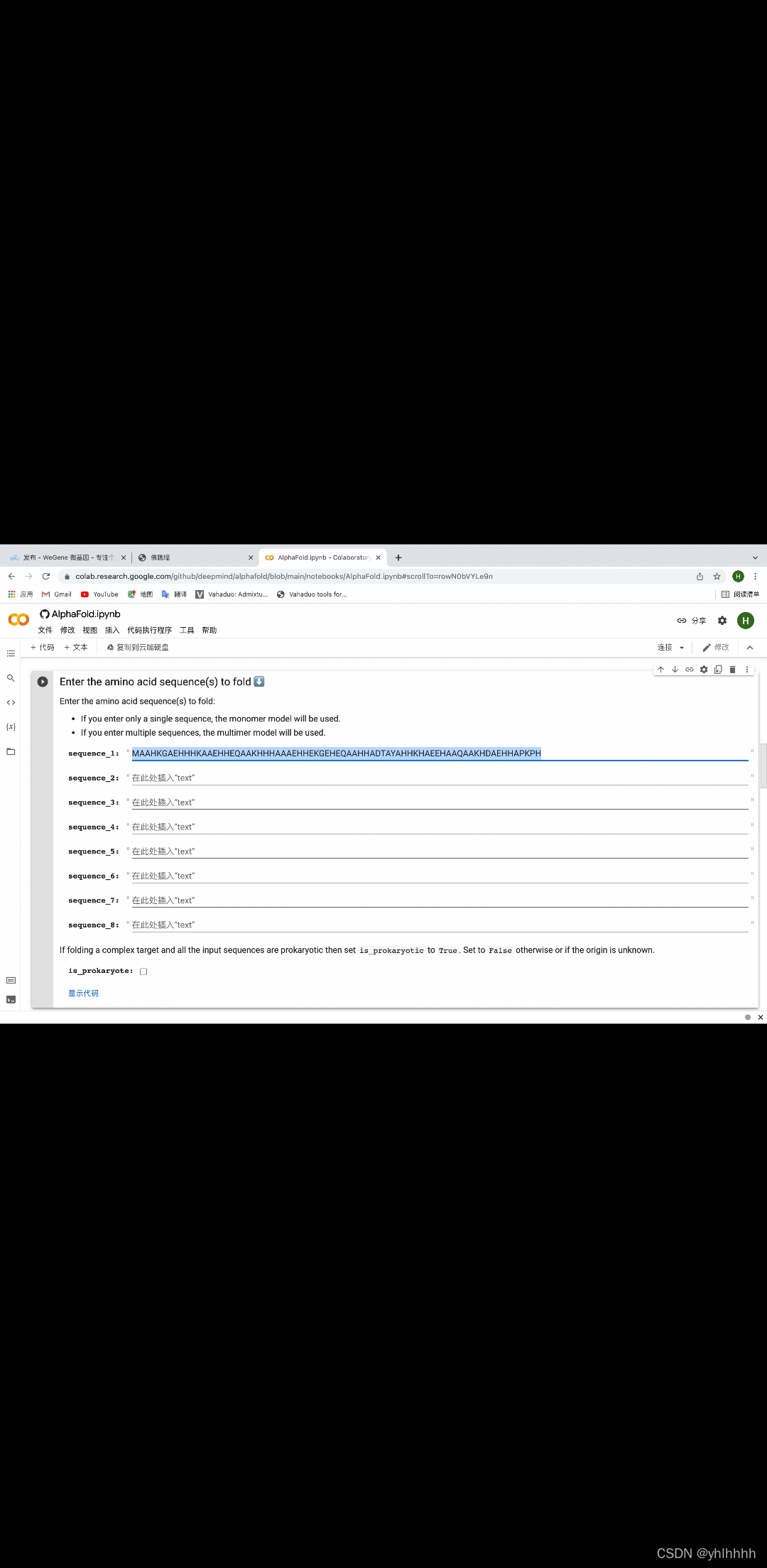
再将刚才copy的氨基酸序列放在图中选中的部分,点运行(注意如果预测氨基酸序列来自于原核生物记得勾选is_prokaryote)
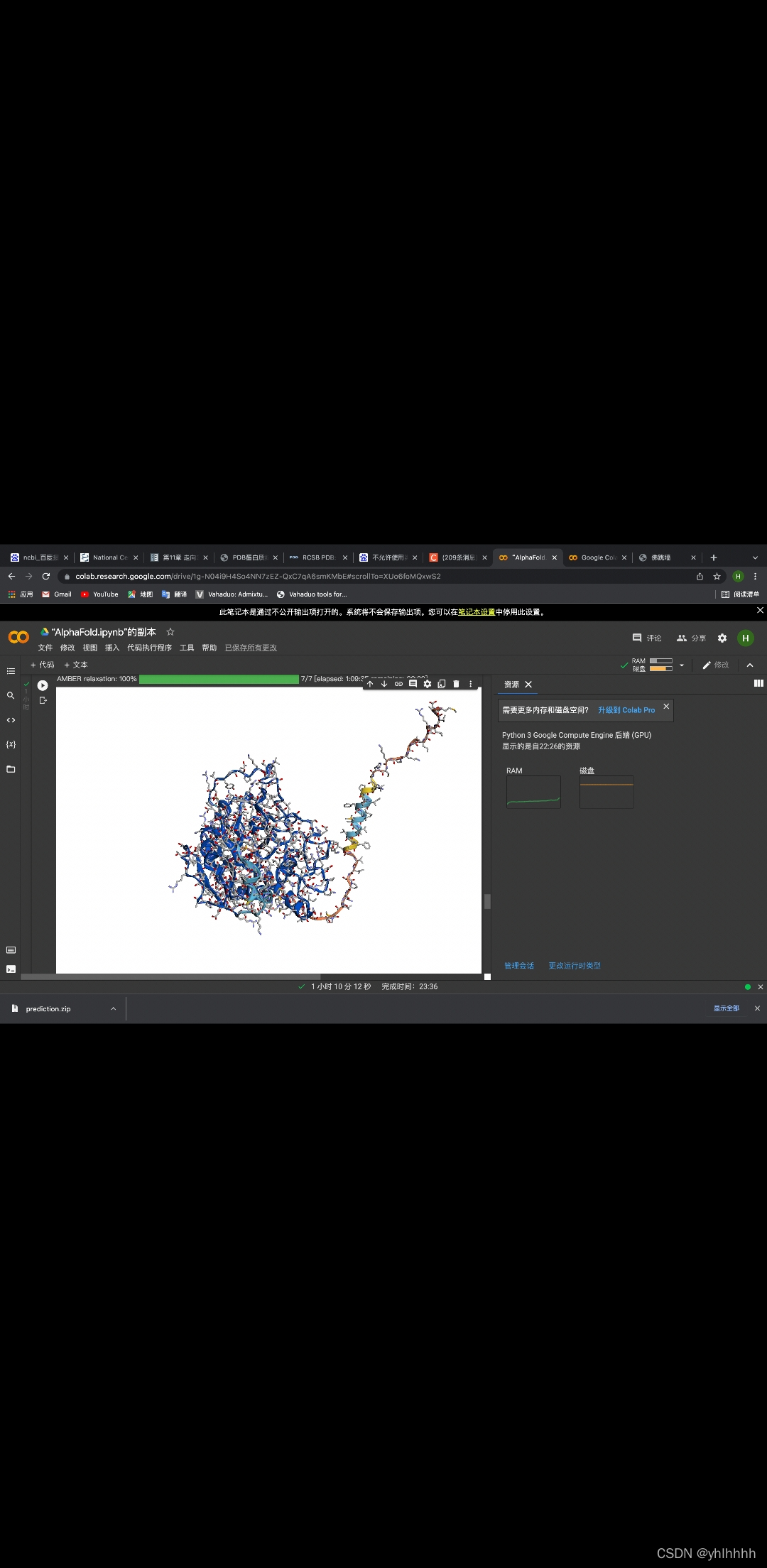
之后接着向下运行完所有单元(时间会比较长,因为会和数据库对比)
运行完会自动下载预测好的蛋白质结构的PDB文件

之后可以利用pyMOL软件在线下打开PDB文件,输出图片
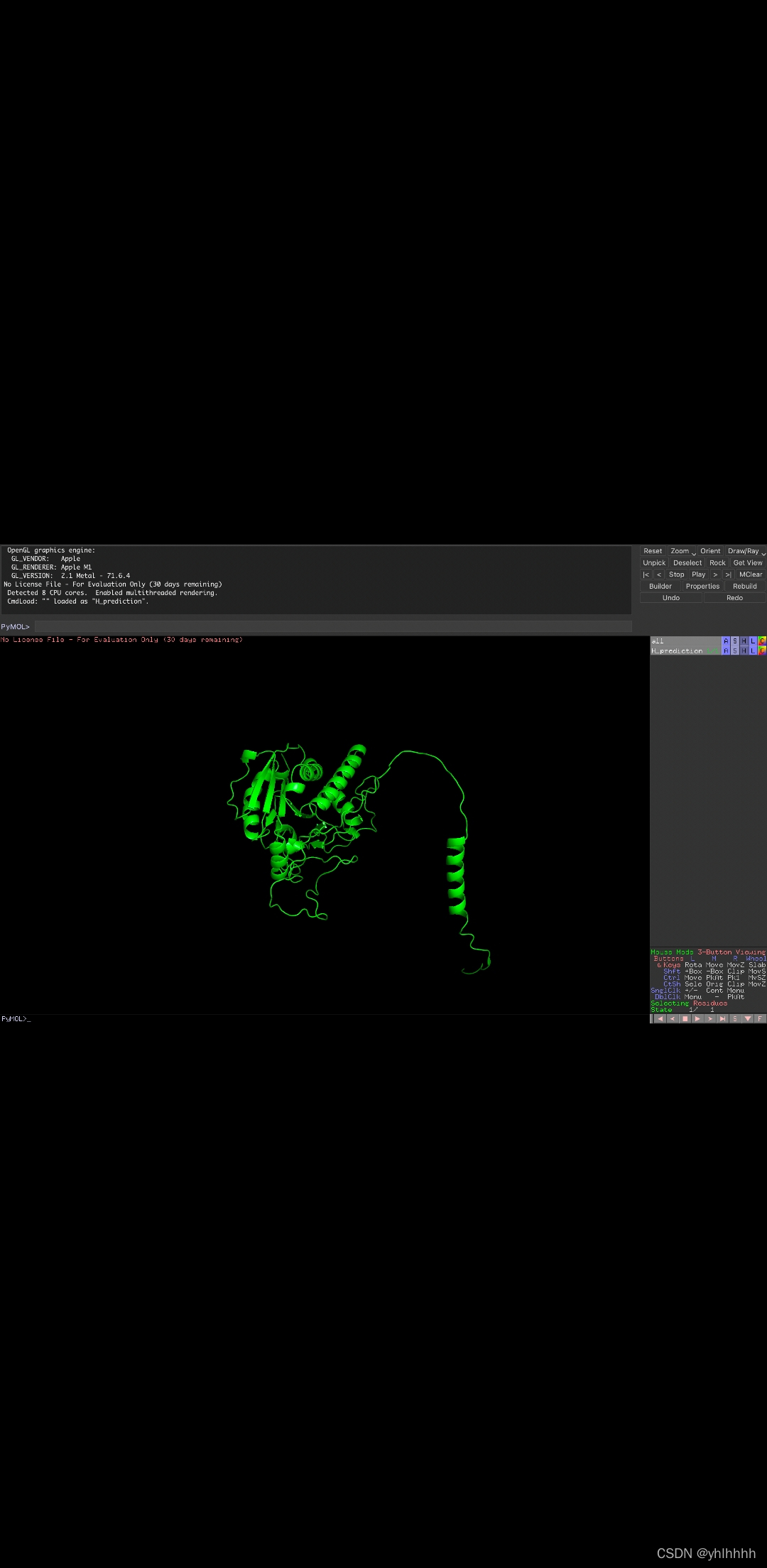
你还可以通过这个软件改变颜色
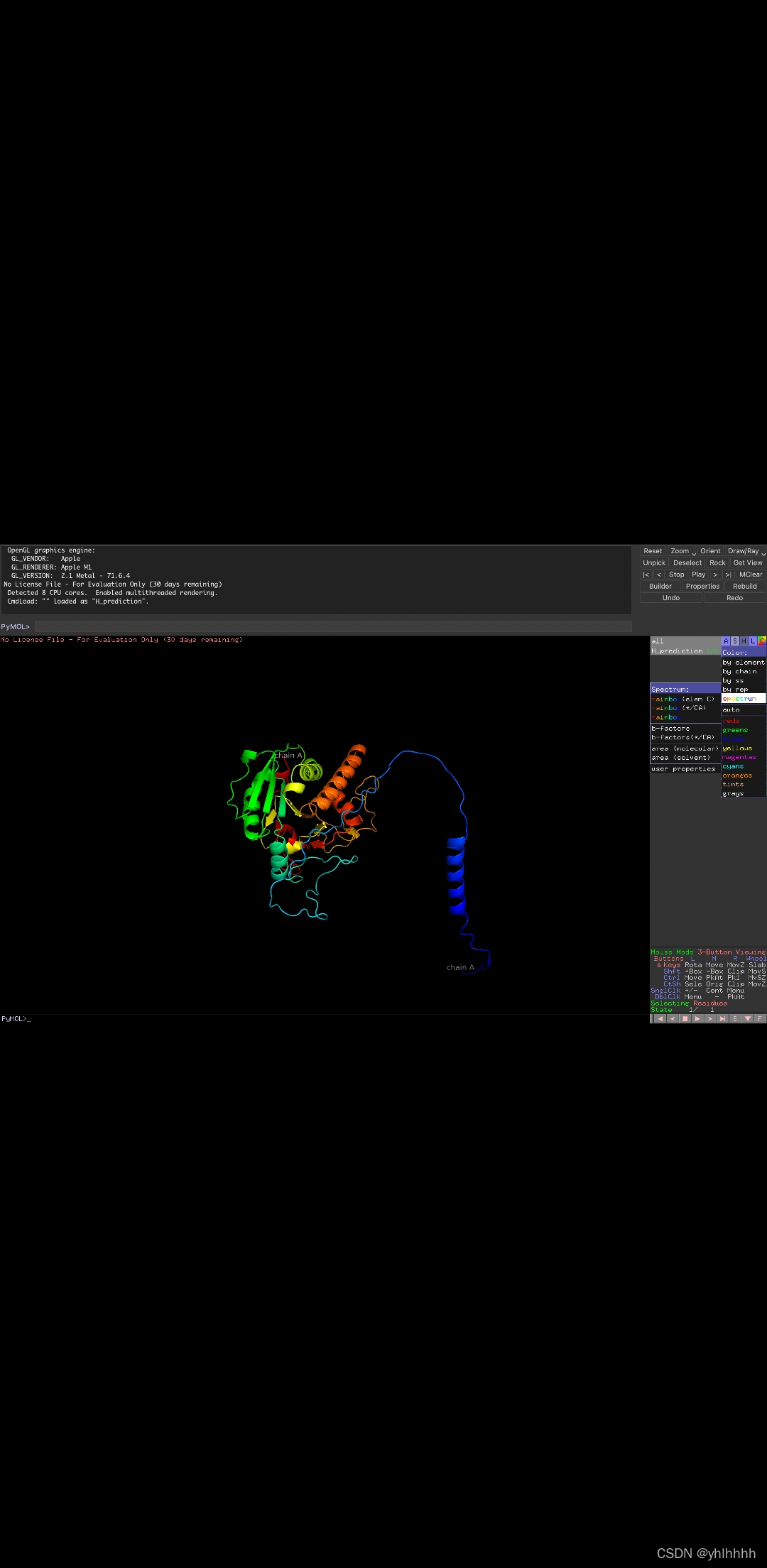
结果展示:
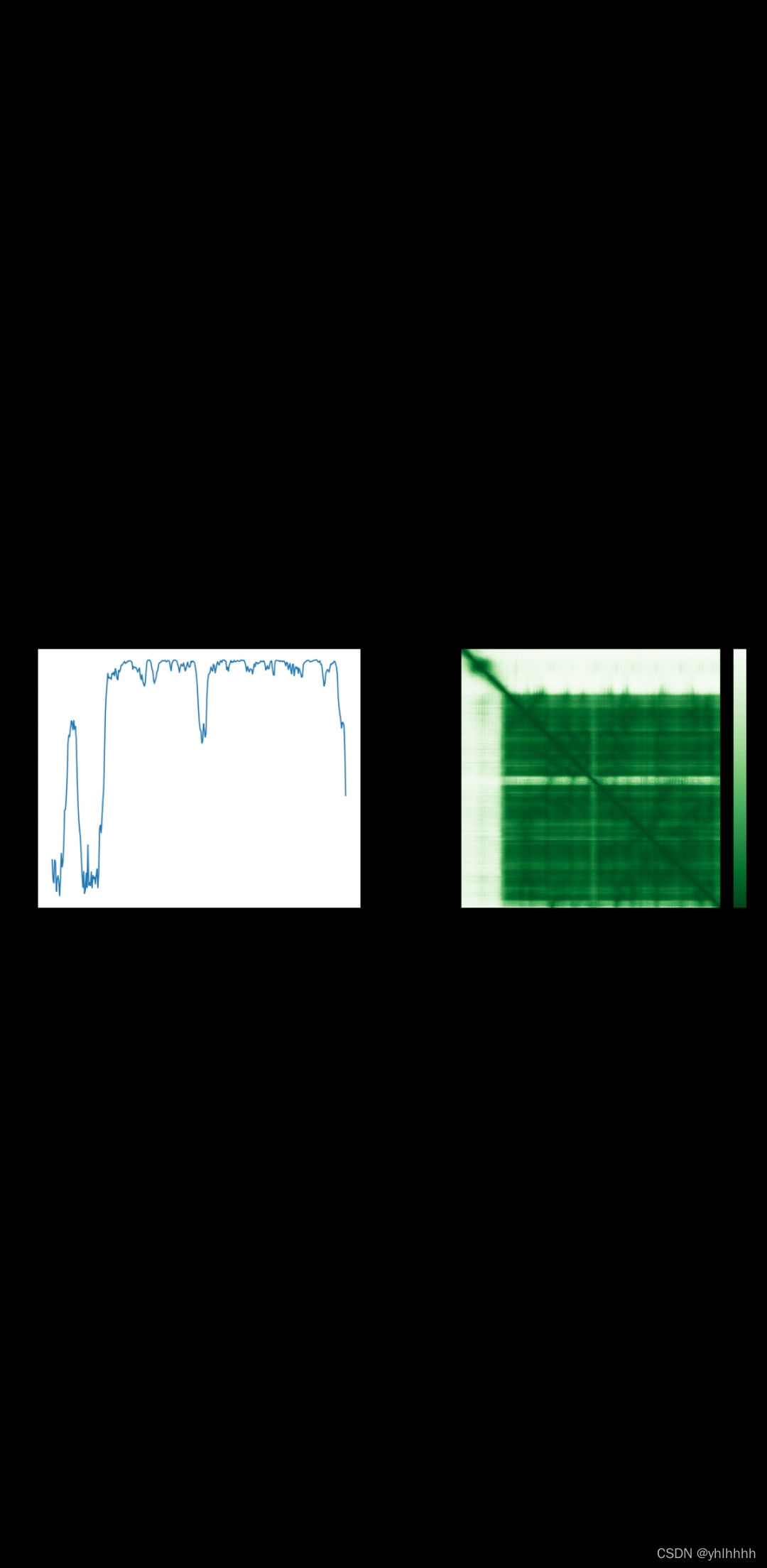
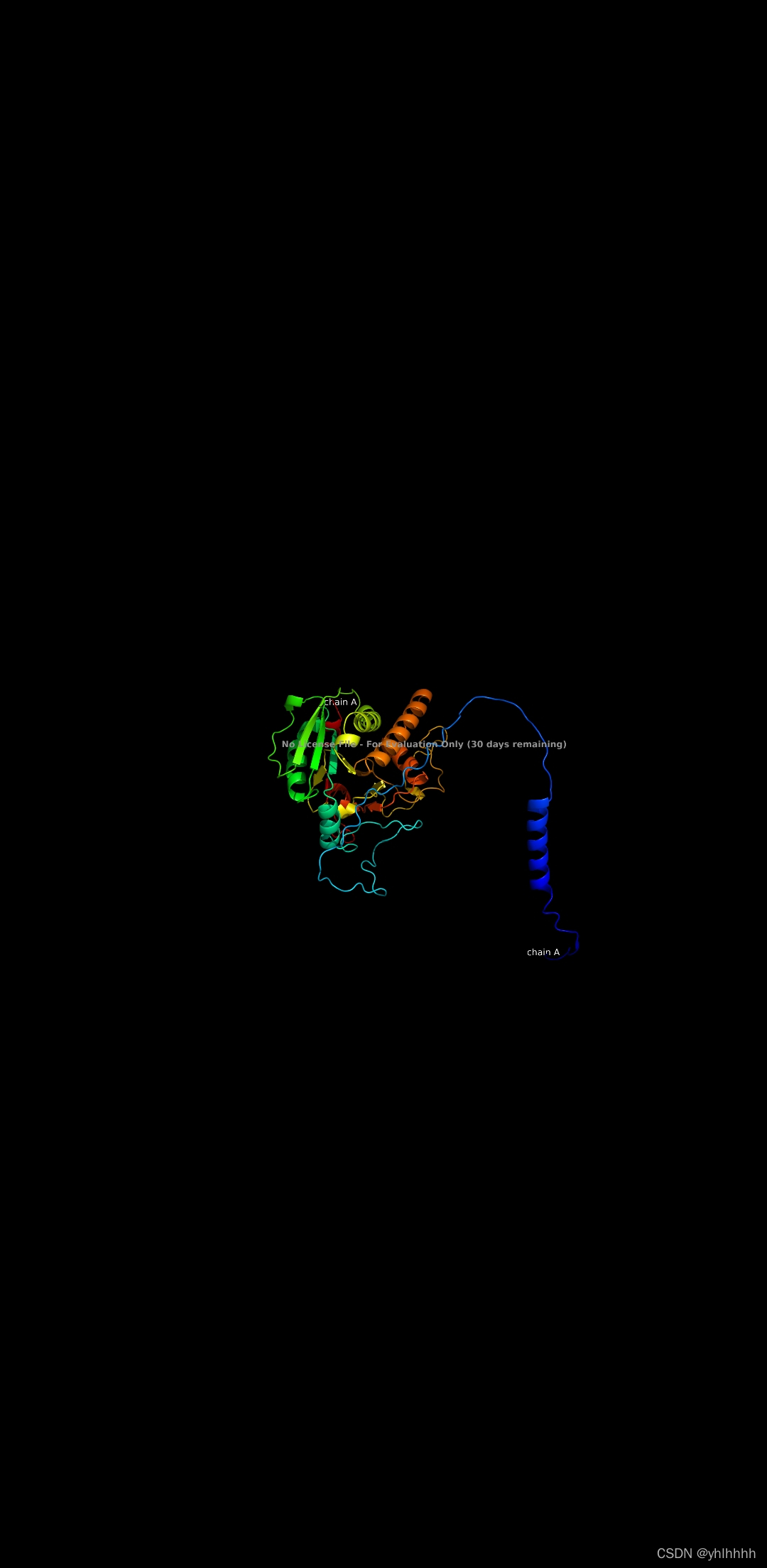
现在我正在研究如何把PDB文件转换为STL文件,之后或许能将预测结构用3D打印机打印出来,敬请期待~

























 1292
1292

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










