大家好,我是邓飞。
GWAS中LD衰减图,可以形象的查看群体LD衰减的情况。
LD衰减是由于连锁不平衡所致,LD衰减速度在不同物种或者不同亚种中差异不同,通常用LD衰减到一般的距离来作为群体的衰减距离(还有其它计算方法),如果LD衰减很快,则在进行GWAS分析时需要更多的位点才能达到一定的精度(https://blog.csdn.net/yijiaobani/article/details/122812786)。另外,LD衰减也可以反应群体受选择的情况,一般来说,野生群体比驯化改良群体LD衰减快,异花授粉比自花授粉植物LD衰减快。
LD图分为单个群体和多个群体的图,今天使用软件PopLDdecay来实现一下。这个软件需要Linux系统。
目标图:

1. 数据
基因型数据是vcf数据,plink格式的数据可以转化为vcf数据。
- plink数据
- vcf数据
$ ls hebing.ped hebing.map
hebing.map hebing.ped
上面示例中用的是plink数据,这里转化为vcf格式的数据:
plink --file hebing --recode vcf-iid --allow-extra-chr --chr-set 40 --out file
结果:
$ ls file.vcf
file.vcf
2. PopLDdecay软件安装
官网:https://github.com/BGI-shenzhen/PopLDdecay
安装方法:
git clone https://github.com/hewm2008/PopLDdecay.git
cd PopLDdecay; chmod 755 configure; ./configure;
make;
mv PopLDdecay bin/; # [rm *.o]
测试:
$ PopLDdecay
Usage: PopLDDecay -InVCF <in.vcf.gz> -OutStat <out.stat>
-InVCF <str> Input SNP VCF Format
-InGenotype <str> Input SNP Genotype Format
-OutStat <str> OutPut Stat Dist ~ r^2/D' File
-SubPop <str> SubGroup Sample File List[ALLsample]
-MaxDist <int> Max Distance (kb) between two SNP [300]
-MAF <float> Min minor allele frequency filter [0.005]
-Het <float> Max ratio of het allele filter [0.88]
-Miss <float> Max ratio of miss allele filter [0.25]
-EHH <str> To Run EHH Region decay set StartSite [NA]
-OutFilterSNP OutPut the final SNP to calculate
-OutType <int> 1: R^2 result 2: R^2 & D' result 3:PairWise LD Out[1]
See the Help for more OutType [1-8] details
-help Show more help [hewm2008 v3.40]
显示安装完成
3. 单个品种的绘制LD图
~/bin/PopLDdecay -InVCF file.vcf -OutStat LDdecay
~/bin/Plot_OnePop.pl -inFile LDdecay.stat.gz --output re1 -bin1 10 -bin2 100
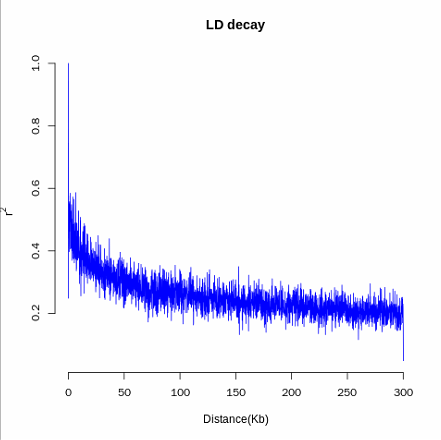
这里面,我用的是芯片数据,位点较少,所以曲线不平滑,可以修改bin2=500,在进行绘图:
~/bin/Plot_OnePop.pl -inFile LDdecay.stat.gz --output re2 -bin1 10 -bin2 500

4. 多品种绘制LD图
如果要运行A,B,C三个品种的LD图,先要把ID整理为三个文件:
- A.txt
- B.txt
- C.txt
文件:
$ ls *txt
A.txt B.txt C.txt total_name.txt
分别针对于每个品种,计算:
~/bin/PopLDdecay -InVCF file.vcf -OutStat A.stat.gz -SubPop A.txt
~/bin/PopLDdecay -InVCF file.vcf -OutStat B.stat.gz -SubPop B.txt
~/bin/PopLDdecay -InVCF file.vcf -OutStat C.stat.gz -SubPop C.txt
结果文件:
$ ls *stat.gz
A.stat.gz B.stat.gz C.stat.gz
生成multi.list,格式为两列,第一列为文件名称,第二列为品种名称,内容为:
$ cat multi.list
A.stat.gz A
B.stat.gz B
C.stat.gz C
作图:
perl ~/bin/Plot_MultiPop.pl -inList multi.list --output re3 -bin1 10 -bin2 500

























 5115
5115

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








