diamond/blast的nr或者nt数据库,在构建数据库时可以添加物种注释信息。如果建库时未添加,也可以后续通过taxonkit 进行注释。
这里以nt数据库的blast的XML格式结果为例,对注释的词条进行物种注释。
软件依赖
- taxonkit
教程:https://bioinf.shenwei.me/taxonkit/
- csvtk 可选
数据库文件
taxdump.tar.gz
wget -c ftp://ftp.ncbi.nih.gov/pub/taxonomy/taxdump.tar.gz
tar -zxvf taxdump.tar.gz
mkdir -p $HOME/.taxonkit
cp names.dmp nodes.dmp delnodes.dmp merged.dmp $HOME/.taxonkit
nt注释:
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/nucl_gb.accession2taxid.gz
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/nucl_gb.accession2taxid.gz.md5
nr注释
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz
https://ftp.ncbi.nlm.nih.gov/pub/taxonomy/accession2taxid/prot.accession2taxid.gz.md5
过程
获取比对到的accession。
这里使用shuf 随机抽取一部分作为演示。
$ cat test.xml | grep '<Hit_num>1</Hit_num>' -A 3 |grep 'Hit_accession' |sed -e 's/.*>\(\S\+\)<.*/\\b\1\\b/g' > all_accession.txt
$ shuf -n 100 all_accession.txt > some_all_accession.txt
$ head some_all_accession.txt
KF294186
XM_010369188
AC192591
DQ816305
FM252655
AC213341
M63543
JN685492
KT588645
AC191970
根据accession获取txid
some_all_accession.txt词条较少时还可以用grep抓取,词条过多的时候耗时会很多。- 个人想法是,这一步用R读取
nucl_gb.accession2taxid.gz,然后保存为Rdata。需要用时直接在R中加载,然后读入some_all_accession.txt的词条进行匹配过滤。
$ zcat nucl_gb.accession2taxid.gz |grep -F -f some_all_accession.txt > some_accession2taxid.txt
$ head some_accession2taxid.txt
AB031215 AB031215.1 86665 7007447
AB225911 AB225911.1 5062 84573614
AC006354 AC006354.2 9606 4309813
AC055716 AC055716.24 9606 14589354
AC103779 AC103779.5 9606 28173127
AC191970 AC191970.3 9598 123187573
AC192591 AC192591.1 9601 116292321
AC194763 AC194763.3 9598 158262523
AC198151 AC198151.2 61853 126352953
AC206703 AC206703.3 9601 168986621
获取taxid
$ cut -f 3 some_accession2taxid.txt |sort -n |uniq > some_ids.txt
$ head some_ids.txt
1270
1402
1423
1773
5061
5062
5068
7165
9544
taxonkit 转换
taxonkit lineage some_ids.txt -j 10 |taxonkit reformat -f "{k}\t{p}\t{c}\t{o}\t{f}\t{g}\t{s}" -F | cut -f 1,3- | sed '1i\Taxid\tKingdom\tPhylum\tClass\tOrder\tFamily\tGenu\tSpecies' > result.xls
sed -i '1i\Taxid\tKingdom\tPhylum\tClass\tOrder\tFamily\tGenu\tSpecies' result.xls
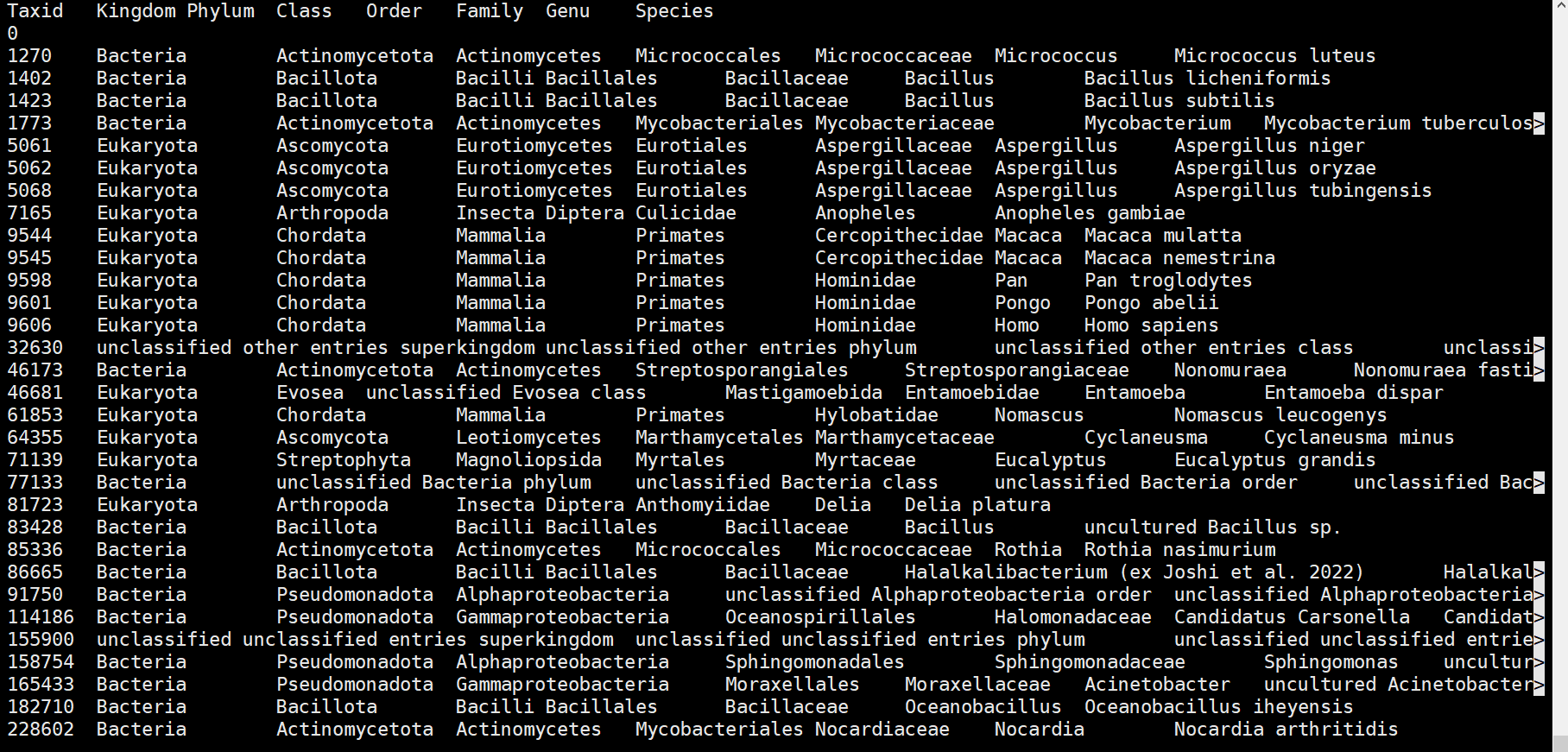
然后可以通过result.xls 和accession2taxid 对比对结果进行注释和统计。






















 505
505

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








