PennCNV官网:https://penncnv.openbioinformatics.org/en/latest/
PennCNV软件的文献: DOI: 10.1101/gr.6861907
本次基于这个2007年发布的文献进行学习,后续根据官网的说明进行进一步学习。(所以文中的部分言论是以2007年的视角进行论述的)
杂交芯片(array-CGH)检测CNV的技术缺陷是仅限于检测数十或数千bp的CNV,而SNP芯片能够更加准确的检测SNP(更灵敏,并且SNP芯片不需要进行PCR扩增,不会造成基因组偏倚,所以获得的SNP检测信息更加真实),由于SNP芯片中SNP位点的距离可达几至几十Kb(不同密度的SNP芯片其距离范围不同),所以SNP芯片相比于杂交芯片更有利于发现大范围的CNV。
PennCNV是专门用于Illumina平台芯片数据进行CNV分析的,对于Illumina平台自带的CNV分析软件------BeadStudio软件的LOH-plus模块,LOH-plus模块通过在沿着染色体的滑动窗口中计算SNP的B等位基因频率的模式来鉴定拷贝数变化。虽然实现简单,但滑动窗口方法对于检测到的CNV具有有限且相对粗糙的边界分辨率。而PennCNV通过使用隐马尔可夫模型(HMM),纳入了log R比率和B等位基因频率,再加上基于贝叶斯模型将系谱等信息纳入矫正,能够显著提高CNV检测的分辨率(更加清晰的CNV编辑的鉴定)。
常规的算法将CNV的类型分为了三类:“loss,”“normal,” and “gain”,而PennCNV将CNV分为了五种类型(由六种可能的情况造成)(下表)。
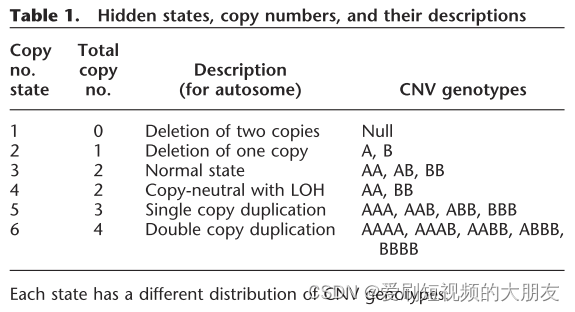
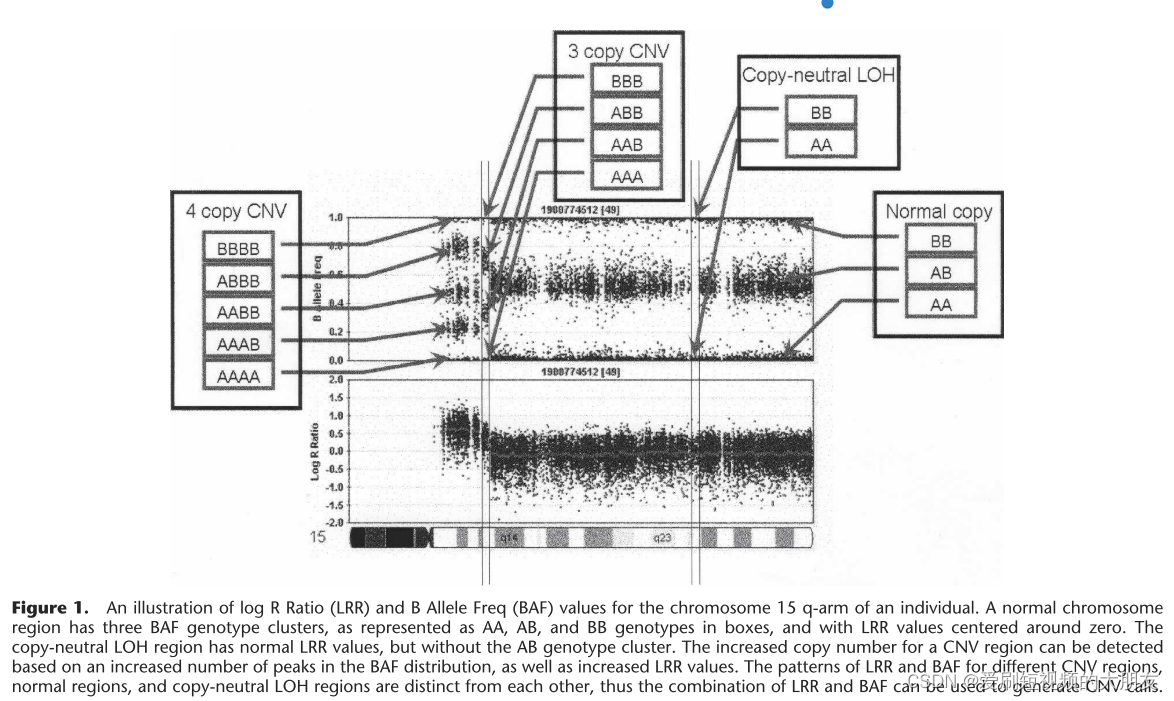
为了充分利用每个SNP的所有可用信息,PennCNV将几个组件一起合并到隐马尔可夫模型(HMM)中,包括LRR,BAF,相邻SNP之间的距离和B等位基因的群体频率。LRR和BAF值都可以从BeadStudio显示和导出,前提是存在具有每个SNP的规范聚类位置的适当聚类文件。相邻SNP之间的距离决定了它们之间具有拷贝数状态变化的概率。每个SNP具有被称为A和B等位基因的两个等位基因,因此我们使用术语“B等位基因的群体频率”将其与测量等位基因强度比的BAF术语区分开。所有SNPs的B等位基因的群体频率值是从具有混合种族背景和正常表型的大量个体汇编的;然后确定每个拷贝数状态的拷贝数基因型的可能性。
我们认为后代中的绝大多数CNV是从父母遗传的(Locke et al.2006),因此在后代中推断但在亲本中未检测到的CNV的分数(CNV-NDP)可用作假阳性和假阴性率的复合量度。因此,我们检查了由三个不同平台产生的CNV调用中HapMap CEU + YRI后代中CNV-NDP的分数。
CNV学习2(illumina芯片分析CNV的主流软件------PennCNV)
于 2023-05-05 12:47:49 首次发布






















 2338
2338











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








