桓峰基因公众号推出单细胞生信分析教程并配有视频在线教程,目前整理出来的相关教程目录如下:
SCS【4】单细胞转录组数据可视化分析 (Seurat 4.0)
SCS【6】单细胞转录组之细胞类型自动注释 (SingleR)
SCS【7】单细胞转录组之轨迹分析 (Monocle 3) 聚类、分类和计数细胞
SCS【8】单细胞转录组之筛选标记基因 (Monocle 3)
SCS【9】单细胞转录组之构建细胞轨迹 (Monocle 3)
SCS【10】单细胞转录组之差异表达分析 (Monocle 3)
SCS【11】单细胞ATAC-seq 可视化分析 (Cicero)
SCS【12】单细胞转录组之评估不同单细胞亚群的分化潜能 (Cytotrace)
SCS【13】单细胞转录组之识别细胞对“基因集”的响应 (AUCell)
SCS【15】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (CellPhoneDB)
SCS【16】从肿瘤单细胞RNA-Seq数据中推断拷贝数变化 (inferCNV)
SCS【17】从单细胞转录组推断肿瘤的CNV和亚克隆 (copyKAT)
SCS【18】细胞交互:受体-配体及其相互作用的细胞通讯数据库 (iTALK)
简介
如何使用Seurat (>3.2) 来分析空间分辨RNA-seq数据。虽然分析管道类似于单细胞RNA-seq分析的Seurat工作流程,但我们引入了更新的交互和可视化工具,特别强调空间和分子信息的集成。本教程将涵盖常见空间分析可视化内容:
归一化降维和聚类
检测空间变量特征
交互式可视化
与单细胞RNA-seq数据集成
处理多个切片
软件包安装
这几个软件包安装过程中难免会出现个别问题,可以参考下面几个debug文章,不一定全面,但是若相同还是有参考价值的:
软件安装系列:R中从github上安装 seurat-wrappers 解除API限制的问题
软件安装系列:SeuratData 中 Dataset无法下载
remotes::install_github("satijalab/seurat", "seurat5", quiet = TRUE)
remotes::install_github("satijalab/seurat-data", "seurat5", quiet = TRUE)
remotes::install_github("satijalab/azimuth", "seurat5", quiet = TRUE)
remotes::install_github("satijalab/seurat-wrappers", "seurat5", quiet = TRUE)
remotes::install_github("stuart-lab/signac", "seurat5", quiet = TRUE)
remotes::install_github("bnprks/BPCells", quiet = TRUE)数据读取
我们将使用最近发布的使用Visium v1化学生成的矢状小鼠大脑切片数据集。有两个连续的前部和两个(匹配的)连续的后部。使用LoadData()函数将其加载到Seurat中。
library(Seurat)
options(Seurat.object.assay.version = "v5")
library(ggplot2)
library(patchwork)
library(dplyr)
library(SeuratData)还可以使用我们的SeuratData包方便地访问数据:
InstallData("stxBrain")安装数据集后,?stxBrain以了解更多信息:
`?`(stxBrain)
Brain <- LoadData("stxBrain", type = "anterior1")实例操作
数据预处理
我们通过基因表达数据预处理步骤类似于典型的单细胞 scRNA-seq。首先需要对数据进行标准化,以解释跨数据点测序深度的差异。对于空间数据集,分子计数/点的差异可能是巨大的,特别是如果整个组织的细胞密度存在差异。我们在这里看到了大量的异质性,这需要有效的标准化。
绘制单细胞数据的小提琴图
VlnPlot(Brain, features = "nCount_Spatial", pt.size = 0.1) + NoLegend()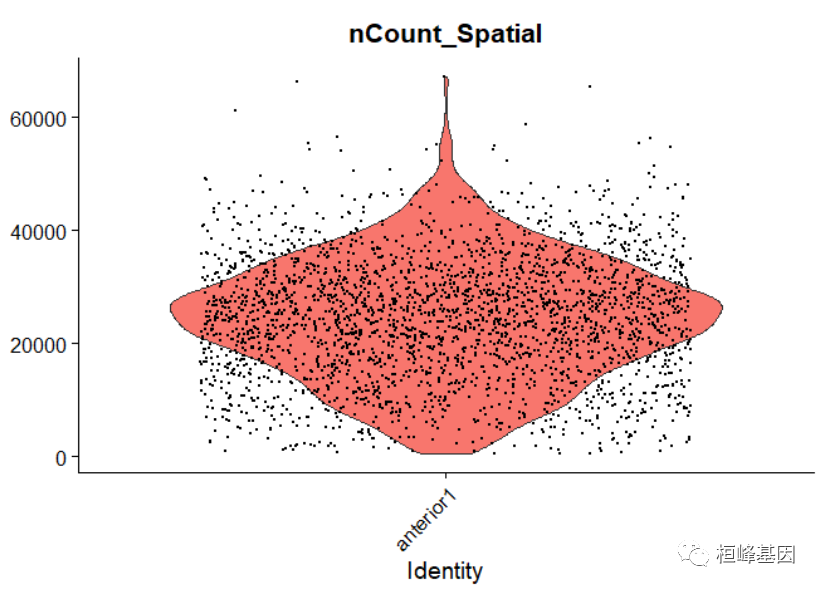
可视化空间聚类和表达式数据
SpatialFeaturePlot(Brain, features = "nCount_Spatial") + theme(legend.position = "right")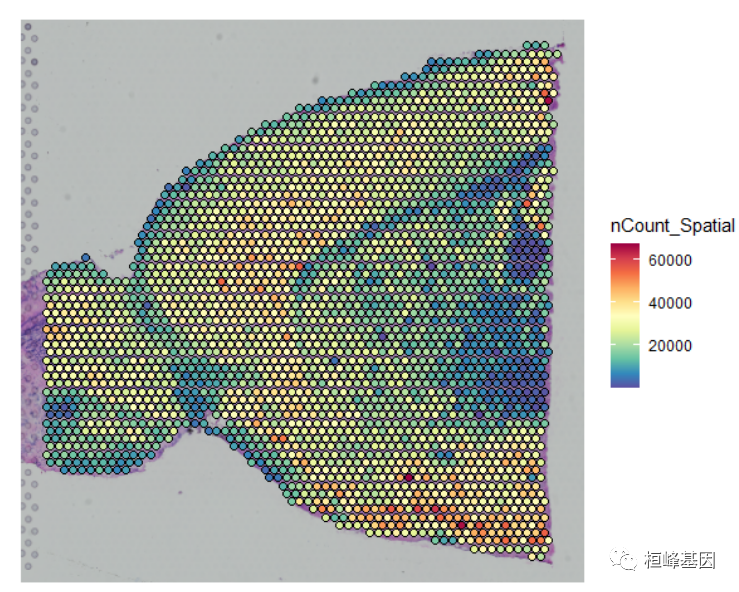
这些图表明,在分子计数的差异在各个点不仅仅是技术性的性质,但也依赖于组织解剖。例如,组织中神经元减少的区域(如皮层白质),重复地表现出较低的分子计数。因此,标准方法(如LogNormalize()函数)强制每个数据点在规范化后具有相同的底层“大小”,这可能是有问题的。
作为替代方案,建议使用 SCTransform(),构建了基因表达的正则化负二项式模型,以便在保留生物方差的同时解释技术伪影。SCTransform() 将数据规范化,检测高方差特征,并将数据存储在SCT分析中。
Brain <- SCTransform(Brain, assay = "Spatial", verbose = FALSE)基因表达可视化
在Seurat中,我们有功能来探索空间数据固有的视觉本质并与之交互。Seurat中的SpatialFeaturePlot()函数扩展了FeaturePlot(),并且可以在组织组织学之上覆盖分子数据。例如,在小鼠大脑的这个数据集中,基因Hpca是一个强海马标记物,Ttr是脉脉络丛标记物。
SpatialFeaturePlot(Brain, features = c("Hpca", "Ttr"))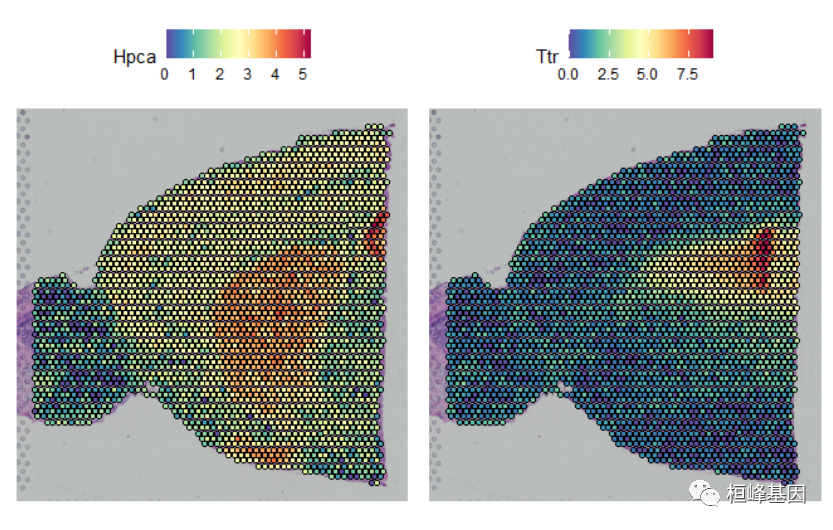
加上legend:
library(ggplot2)
SpatialFeaturePlot(Brain, features = c("Ttr")) + theme(legend.text = element_text(size = 0),
legend.title = element_text(size = 20), legend.key.size = unit(1, "cm"))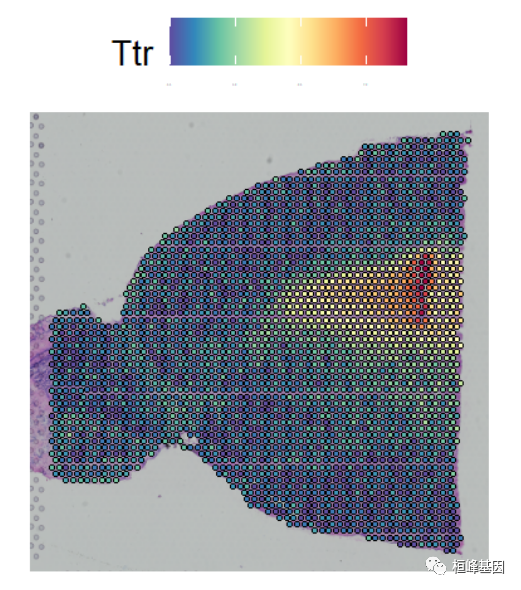
# jpeg(filename = 'spatial_vignette_ttr.jpg', height = 700, width = 1200,
# quality = 50) print(plot) dev.off()调整绘图参数
Seurat的默认参数强调分子数据的可视化。然而,你也可以调整斑点的大小(及其透明度),以提高组织图像的可视化,通过改变以下参数:因子-这将缩放斑点的大小。默认值是1.6 alpha -最小和最大透明度。默认值是c(1,1):
SpatialFeaturePlot(Brain, features = "Ttr", pt.size.factor = 1)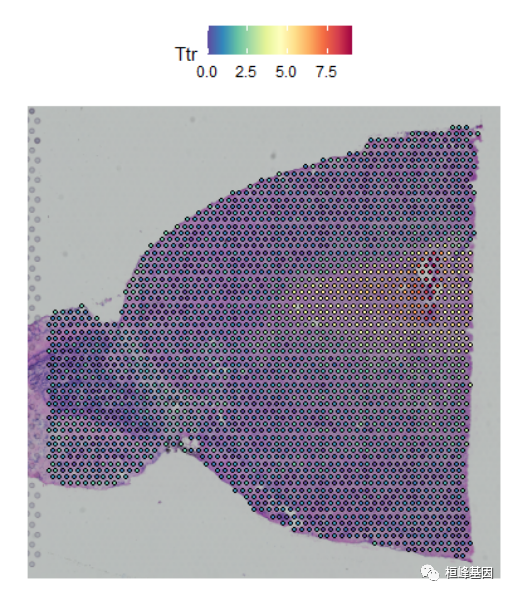
尝试设置为alpha c(0.1, 1),以降低低表达式点的透明度:
SpatialFeaturePlot(Brain, features = "Ttr", alpha = c(0.1, 1))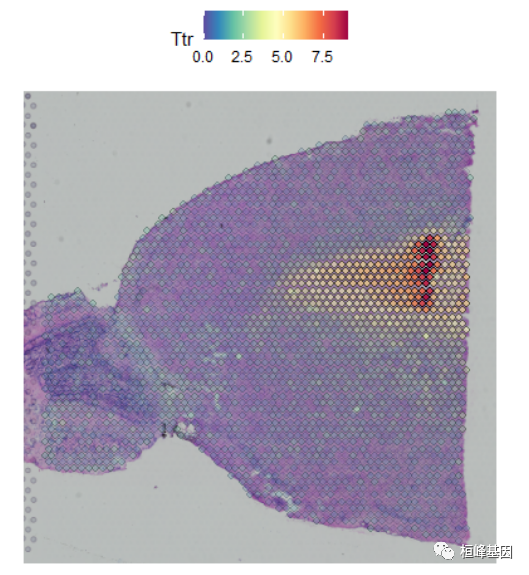
降维、聚类和可视化 然后,可以继续对RNA表达数据进行降维和聚类,使用与我们用于scRNA-seq分析相同的工作流程。
Brain <- RunPCA(Brain, assay = "SCT", verbose = FALSE)
Brain <- FindNeighbors(Brain, reduction = "pca", dims = 1:30)
Brain <- FindClusters(Brain, verbose = FALSE)
Brain <- RunUMAP(Brain, reduction = "pca", dims = 1:30)然后,我们可以在UMAP空间(使用DimPlot())中可视化聚类结果,或者使用SpatialDimPlot()在图像上叠加。
DimPlot(Brain, reduction = "umap", label = TRUE)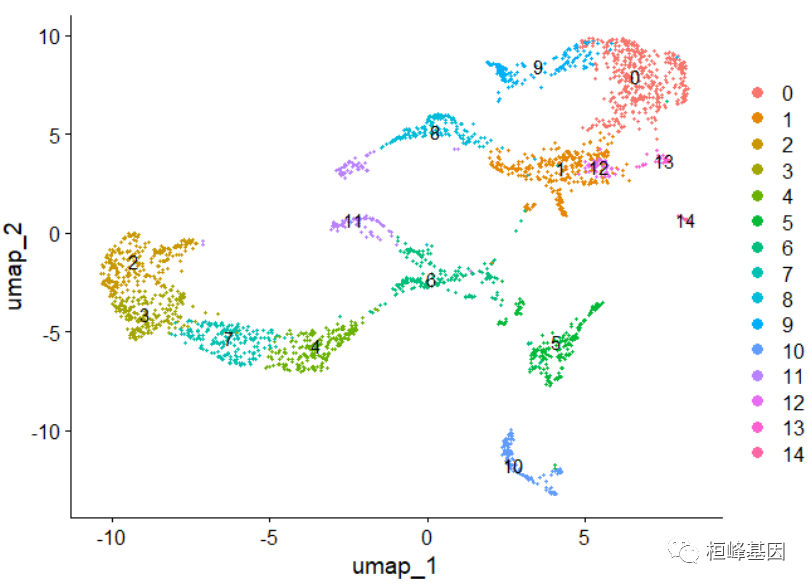
再次可视化空间聚类和表达式数据:
SpatialDimPlot(Brain, label = TRUE, label.size = 3)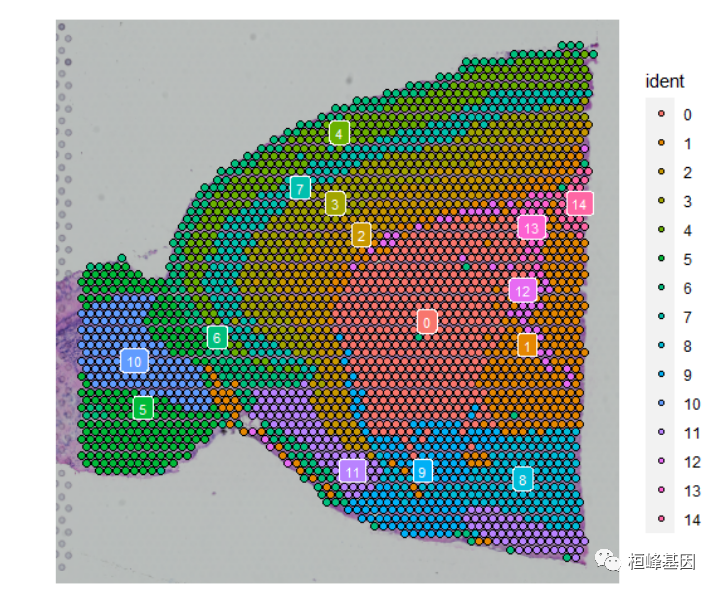
由于有许多颜色,可视化哪个体素属于哪个簇可能具有挑战性。我们有一些策略来帮助解决这个问题。设置label参数会在每个集群的中间位置放置一个彩色框(参见上面的图表)。参数以在SpatialDimPlot()上划分感兴趣的特定细胞。这对于区分单个cluster的空间定位非常有用,如下图所示:
SpatialDimPlot(Brain, cells.highlight = CellsByIdentities(object = Brain, idents = c(2,
1, 4, 3, 5, 8)), facet.highlight = TRUE, ncol = 3)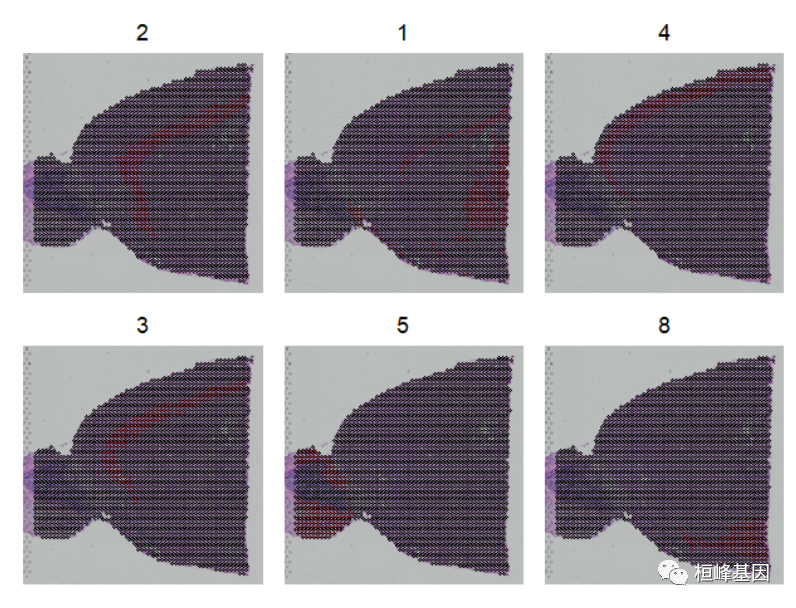
交互绘图
我们还内置了许多交互式绘图功能。SpatialDimPlot()和SpatialFeaturePlot()现在都有一个交互参数,当设置为TRUE时,将打开Rstudio查看器窗格,并显示一个交互式的Shiny图形。下面的示例演示了一个交互式的SpatialDimPlot(),您可以将鼠标悬停在点上并查看细胞的名称和当前细胞类型。
SpatialDimPlot(Brain, interactive = TRUE)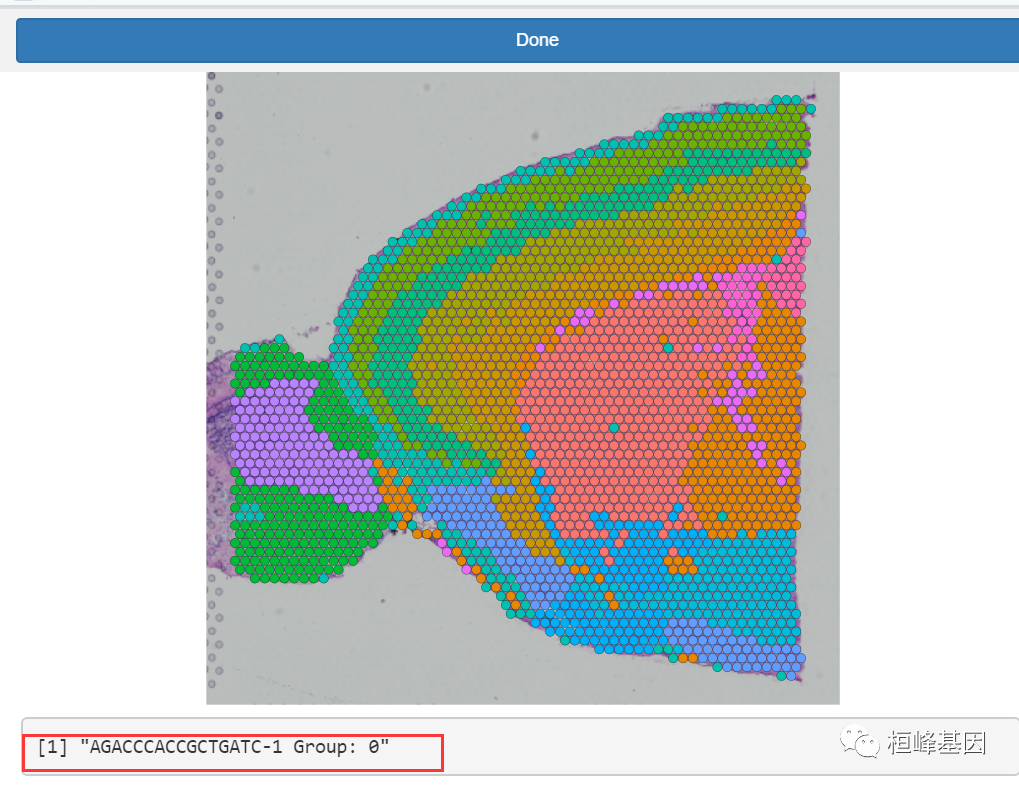
对于SpatialFeaturePlot(),将interactive设置为TRUE会弹出一个交互式窗格,在其中您可以调整斑点的透明度、点大小以及正在绘制的分析和特征。在浏览数据之后,选择done按钮将返回最后一个活动图形作为ggplot对象。
SpatialFeaturePlot(Brain, features = "Ttr", interactive = TRUE)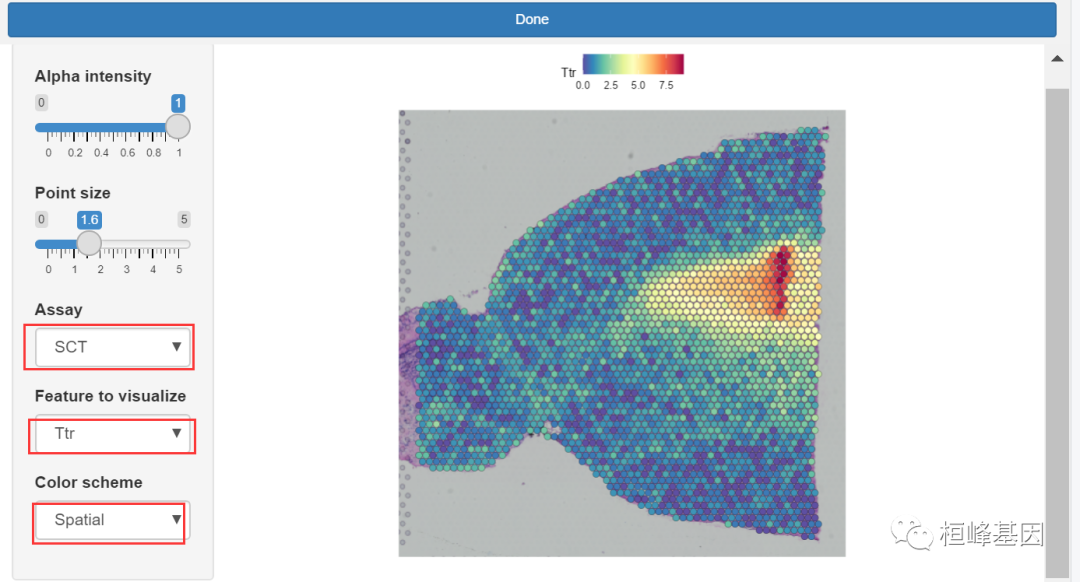
LinkedDimPlot()函数将UMAP表示与组织图像表示联系起来,并允许交互选择。例如,您可以在UMAP图中选择一个区域,图像表示中相应的点将被突出显示。
LinkedDimPlot(Brain)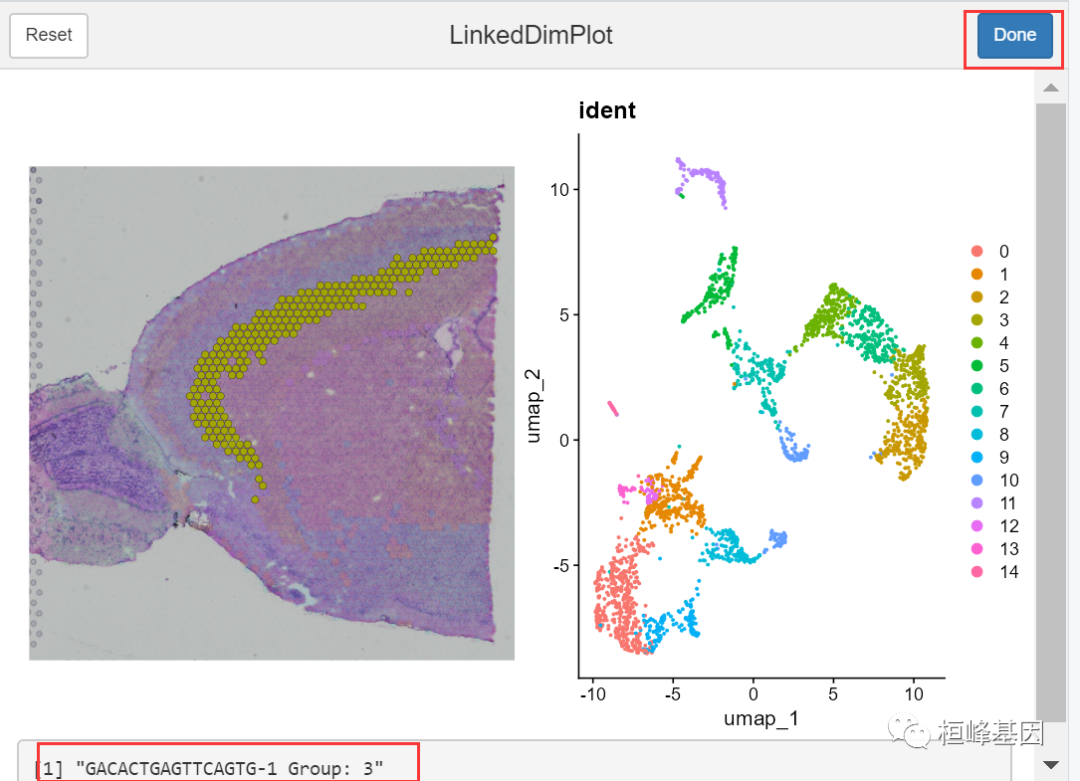
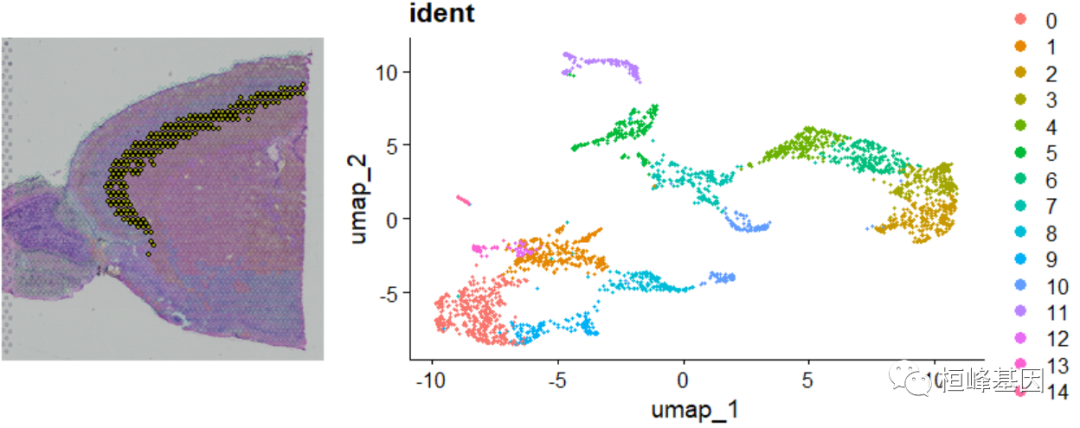
空间可变特征的识别
Seurat提供了两种工作流程来识别与组织内空间位置相关的分子特征。第一种是根据组织内预注释的解剖区域进行差异表达,这可能是由无监督聚类或先验知识决定的。这种策略在这种情况下是有效的,因为上面的集群表现出明显的空间限制。
de_markers <- FindMarkers(Brain, ident.1 = 5, ident.2 = 6)
SpatialFeaturePlot(object = Brain, features = rownames(de_markers)[1:3], alpha = c(0.1,
1), ncol = 3)
在FindSpatiallyVariables()中实现的另一种方法是在没有预先注释的情况下搜索显示空间模式的特征。默认的方法(method = 'markvariogram)受到trendscek的启发,该方法将空间转录组数据建模为标记点过程,并计算“变异函数”,以识别其表达水平依赖于其空间位置的基因。
Brain <- FindSpatiallyVariableFeatures(Brain, assay = "SCT", features = VariableFeatures(Brain)[1:1000],
selection.method = "moransi")现在,我们将通过该测量方法确定的前6个特征的表达式可视化。
top.features <- head(SpatiallyVariableFeatures(Brain, selection.method = "moransi"),
6)
SpatialFeaturePlot(Brain, features = top.features, ncol = 3, alpha = c(0.1, 1))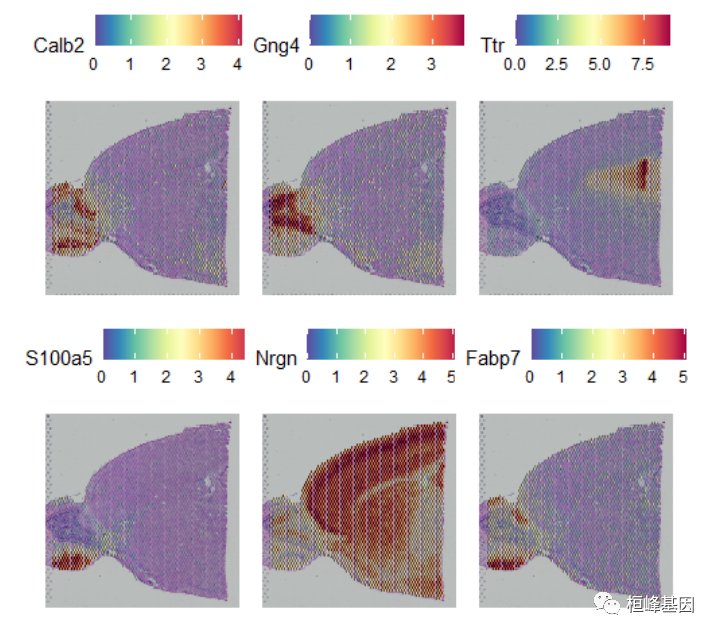
解剖区域的子集
与单细胞对象一样,可以对对象进行子集化,以集中于数据的子集。这里,我们大致划分了额叶皮层。这一过程还有助于将这些数据与下一节中的皮层scRNA-seq数据集集成。首先,我们取一个簇的子集,然后根据精确的位置进一步细分。在子集之后,我们可以在完整图像或裁剪图像上可视化皮质细胞。
cortex <- subset(Brain, idents = c(1, 2, 3, 4, 6, 7))现在删除额外的细胞,使用 SpatialDimPlots() 来可视化删除什么
cortex <- subset(cortex, anterior1_imagerow > 400 | anterior1_imagecol < 150, invert = TRUE)
cortex <- subset(cortex, anterior1_imagerow > 275 & anterior1_imagecol > 370, invert = TRUE)
cortex <- subset(cortex, anterior1_imagerow > 250 & anterior1_imagecol > 440, invert = TRUE)
SpatialDimPlot(cortex, crop = TRUE, label = TRUE)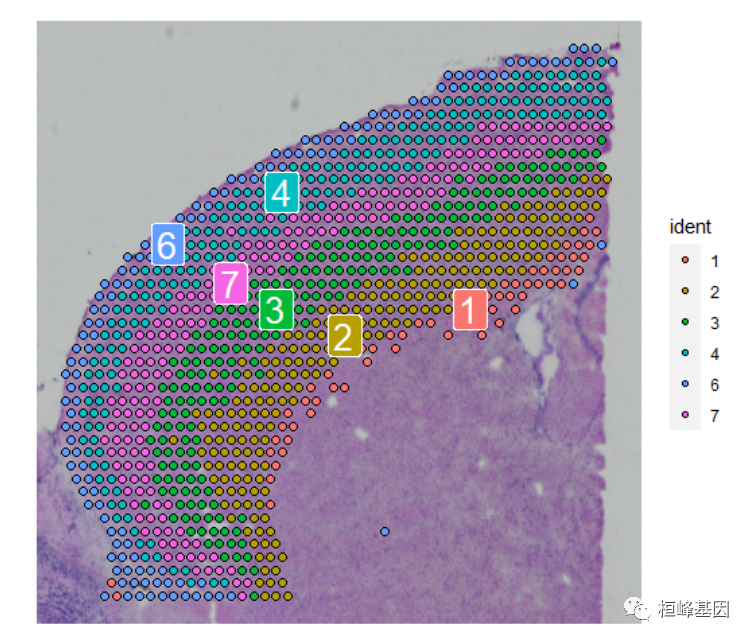
SpatialDimPlot(cortex, crop = FALSE, label = TRUE, pt.size.factor = 1, label.size = 3)
Reference
Hafemeister, C., Satija, R. Normalization and variance stabilization of single-cell RNA-seq data using regularized negative binomial regression. Genome Biol 20, 296 (2019).
这个软件包代码量还是很多的,需要具有一定 R 语言编程基础,并不是看起来那么简单,所有好多老师想直接自己学习教程来分析,但是实质上没有基础还是很难实现,每步报错都不知道该怎样处理,是最崩溃的,所以有需求的老师可以联系桓峰基因,提供最优质的服务!!!
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
有想进生信交流群的老师可以扫最后一个二维码加微信,备注“单位+姓名+目的”,有些想发广告的就免打扰吧,还得费力气把你踢出去!






















 6917
6917











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








