
简 介
对于Cox模型,响应最好是一个由生存包中的Surv()函数创建的Surv对象。对于右删减的数据,该对象的类型应该是“right”,对于(start, stop)数据,它的类型应该是“counting”。为了拟合分层Cox模型,在将响应传递给glmnet()之前,应通过stratifySurv()函数将地层添加到响应中。(为了向后兼容,右审查的数据也可以作为两列矩阵传递,列名为'time'和'status'。后者是一个二元变量,'1'表示死亡,'0'表示正确删除)

软件包安装
glmnet 软件包安装非常简单,直接install.packages()即可。
install.packages("dplyr")
install.packages("glmnet")数据读取
这里使用数据lung,比较经典,不足之处就是自变量少了些。
library(dplyr)
library(glmnet)
library(caret)
library(ggplot2)
library(survival)
library(survminer)
# Summary of dataset in package
head(lung)
## inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
## 1 3 306 2 74 1 1 90 100 1175 NA
## 2 3 455 2 68 1 0 90 90 1225 15
## 3 3 1010 1 56 1 0 90 90 NA 15
## 4 5 210 2 57 1 1 90 60 1150 11
## 5 1 883 2 60 1 0 100 90 NA 0
## 6 12 1022 1 74 1 1 50 80 513 0
table(lung$status)
##
## 1 2
## 63 165
lung$status <- ifelse(lung$status == 2, 1, 0)
lung <- na.omit(lung) # 去掉NA
head(lung)
## inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
## 2 3 455 1 68 1 0 90 90 1225 15
## 4 5 210 1 57 1 1 90 60 1150 11
## 6 12 1022 0 74 1 1 50 80 513 0
## 7 7 310 1 68 2 2 70 60 384 10
## 8 11 361 1 71 2 2 60 80 538 1
## 9 1 218 1 53 1 1 70 80 825 16数据分割
######## 数据分割
library(sampling)
set.seed(123)
# 每层抽取70%的数据
train_id <- strata(lung, "status", size = rev(round(table(lung$status) * 0.7)))$ID_unit
# 训练数据
trainData <- lung[train_id, ]
# 测试数据
testData <- lung[-train_id, ]输入:自变量X至少一项或以上的定量变量或二分类定类变量,因变量Y要求为定量变量(若为定类变量,请使用逻辑回归)。输出:模型检验优度的结果,自变量对因变量的线性关系等等。
# X and Y datasets
train.x<- as.matrix(trainData[, c(1,4:10)])
train.y <- Surv(trainData$time,trainData$status)实例操作
这个我们采用glmnet里面的函数和参数。在glmnet包中alpha数值不同表示的模型则不同.lambda是总的正则化程度,该值越大惩罚力度越大,最终保留的变量越少,模型复杂度越低;alpha是L1正则化的比例。
1)当 alpha=1, 则为Lasso模型;
2)当 alpha=0, 则为Ridge模型;
3)当 1>alpha>0,则为Elastic Net模型。
构建 Lasso + Cox 模型
参数说明:
family = "binomial"代表拟合二分类logistic模型
family = "gaussian"代表拟合线性回归模型
family = "cox"代表拟合cox比例风险回归模型
family = "poisson"代表拟合poisson回归模型
family = "multinomial"代表拟合多分类logistic回归模型
alpha = 1表示lasso回归,alpha = 0表示岭回归
set.seed(123)
fit <- glmnet(train.x, train.y, family = "cox", alpha = 1)
plot(fit, label = T, xvar = "lambda")
交叉验证
通过交叉验证找到最优的lambda值。
########### 交叉验证
fit_cv <- cv.glmnet(train.x, train.y, family = "cox", alpha = 1)
plot(fit_cv)
fit_cv$lambda.min
## [1] 0.03850337
fit_cv$glmnet.fit
##
## Call: glmnet(x = train.x, y = train.y, family = "cox", alpha = 1)
##
## Df %Dev Lambda
## 1 0 0.00 0.271600
## 2 2 0.74 0.247500
## 3 3 1.48 0.225500
## 4 3 2.14 0.205500
## 5 3 2.69 0.187200
## 6 3 3.15 0.170600
## 7 3 3.54 0.155400
## 8 3 3.86 0.141600
## 9 3 4.13 0.129000
## 10 3 4.36 0.117600
## 11 3 4.55 0.107100
## 12 3 4.71 0.097620
## 13 4 4.90 0.088950
## 14 4 5.08 0.081050
## 15 4 5.23 0.073850
## 16 4 5.36 0.067290
## 17 4 5.47 0.061310
## 18 4 5.56 0.055860
## 19 4 5.63 0.050900
## 20 4 5.69 0.046380
## 21 4 5.75 0.042260
## 22 4 5.79 0.038500
## 23 4 5.83 0.035080
## 24 4 5.86 0.031970
## 25 4 5.88 0.029130
## 26 4 5.90 0.026540
## 27 4 5.92 0.024180
## 28 5 5.94 0.022030
## 29 5 5.96 0.020080
## 30 5 5.98 0.018290
## 31 5 5.99 0.016670
## 32 5 6.01 0.015190
## 33 6 6.02 0.013840
## 34 7 6.03 0.012610
## 35 7 6.03 0.011490
## 36 7 6.04 0.010470
## 37 7 6.05 0.009538
## 38 7 6.05 0.008690
## 39 7 6.06 0.007918
## 40 7 6.06 0.007215
## 41 7 6.06 0.006574
## 42 7 6.06 0.005990
## 43 7 6.07 0.005458
## 44 7 6.07 0.004973
## 45 7 6.07 0.004531
## 46 7 6.07 0.004129
coef(fit_cv, s = "lambda.min")
## 8 x 1 sparse Matrix of class "dgCMatrix"
## 1
## inst .
## age .
## sex -0.891930379
## ph.ecog 0.538894763
## ph.karno -0.015540406
## pat.karno .
## meal.cal .
## wt.loss -0.008016507最优模型
glmnet_fit <- glmnet(train.x, train.y, family = "cox", nfolds = 10, keep = TRUE,
alpha = 1, lambda = fit_cv$lambda.min)与Cox回归模型比较
coxph_fit <- coxph(train.y ~ train.x)
plot(coef(glmnet_fit), coef(coxph_fit), col = "red", cex = 2)
abline(0, 1, lty = 2, lwd = 2)
生存分析
f = survfit(glmnet_fit, s = 0.05, x = train.x, y = train.y)
plot(f, col = "red", lwd = 2, ylab = "Time", xlab = "Survival Probability")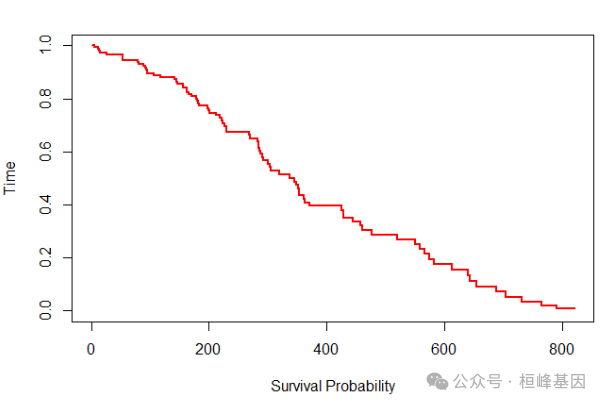
一致性C-index
pred <- predict(glmnet_fit, train.x)
apply(pred, 2, Cindex, y = train.y)
## s0
## 0.6953479
summary(coxph_fit)$concordance[1]
## C
## 0.6920954验证集验证
不同的family对应着不同的性能指标,可以通过glmnet.measures()查看每个family对应的性能指标:
glmnet.measures()
## $gaussian
## [1] "mse" "mae"
##
## $binomial
## [1] "deviance" "class" "auc" "mse" "mae"
##
## $poisson
## [1] "deviance" "mse" "mae"
##
## $cox
## [1] "deviance" "C"
##
## $multinomial
## [1] "deviance" "class" "mse" "mae"
##
## $mgaussian
## [1] "mse" "mae"
##
## $GLM
## [1] "deviance" "mse" "mae"test.x = as.matrix(testData[, c(1, 4:10)])
test.y <- Surv(testData$time, testData$status)
assess.glmnet(glmnet_fit, newx = test.x, newy = test.y)
## $deviance
## [1] 236.7396
## attr(,"measure")
## [1] "Partial Likelihood Deviance"
##
## $C
## [1] 0.5608384
## attr(,"measure")
## [1] "C-index"正则化的分层COX回归
coxph()支持strata()函数,因为它是使用公式形式的,但是glmnet不支持公式形式,只能使用x/y形式的输入,所以如果要实现分层,需要使用stratifySurv()。
继续使用上面的示例数据,我们把观测分成5层:
# 把观测分5层
strata <- rep(1:5, length.out = 117)
y2 <- stratifySurv(train.y, strata) # 对y进行分层
str(y2[1:6])
## 'stratifySurv' num [1:6, 1:2] 361 218 166 170 567 613
## - attr(*, "dimnames")=List of 2
## ..$ : NULL
## ..$ : chr [1:2] "time" "status"
## - attr(*, "type")= chr "right"
## - attr(*, "strata")= int [1:6] 1 2 3 4 5 1构建模型并利用assess.glmnet()函数查看模型性能。
fit_strata <- glmnet(train.x, y2, family = "cox", alpha = 1)
plot(fit_strata, label = T, xvar = "lambda")
cv.fit_strata <- cv.glmnet(train.x, y2, family = "cox", nfolds = 10)
plot(cv.fit_strata)
FinalModel <- glmnet(train.x, y2, family = "cox", alpha = 1, lambda = cv.fit_strata$lambda.min)
apply(predict(FinalModel, train.x), 2, Cindex, y = train.y)
## s0
## 0.6817465
assess.glmnet(FinalModel, newx = test.x, newy = test.y)
## $deviance
## [1] 228.0759
## attr(,"measure")
## [1] "Partial Likelihood Deviance"
##
## $C
## [1] 0.5802658
## attr(,"measure")
## [1] "C-index"Reference
Simon, N., Friedman, J., Hastie, T. and Tibshirani, R. (2011) Regularization Paths for Cox's Proportional Hazards Model via Coordinate Descent, Journal of Statistical Software, Vol. 39(5), 1-13, doi:10.18637/jss.v039.i05.
基于机器学习构建临床预测模型
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
MachineLearning 14. 机器学习之集成分类器(AdaBoost)
MachineLearning 15. 机器学习之集成分类器(LogitBoost)
MachineLearning 16. 机器学习之梯度提升机(GBM)
MachineLearning 17. 机器学习之围绕中心点划分算法(PAM)
MachineLearning 18. 机器学习之贝叶斯分析类器(Naive Bayes)
MachineLearning 19. 机器学习之神经网络分类器(NNET)
MachineLearning 20. 机器学习之袋装分类回归树(Bagged CART)
MachineLearning 21. 机器学习之临床医学上的生存分析(xgboost)
MachineLearning 22. 机器学习之有监督主成分分析筛选基因(SuperPC)
MachineLearning 23. 机器学习之岭回归预测基因型和表型(Ridge)
MachineLearning 24. 机器学习之似然增强Cox比例风险模型筛选变量及预后估计(CoxBoost)
MachineLearning 25. 机器学习之支持向量机应用于生存分析(survivalsvm)
MachineLearning 26. 机器学习之弹性网络算法应用于生存分析(Enet)
MachineLearning 27. 机器学习之逐步Cox回归筛选变量(StepCox)
MachineLearning 28. 机器学习之偏最小二乘回归应用于生存分析(plsRcox)
MachineLearning 29. 机器学习之嵌套交叉验证(Nested CV)
MachineLearning 30. 机器学习之特征选择森林之神(Boruta)
MachineLearning 31. 机器学习之基于RNA-seq的基因特征筛选 (GeneSelectR)
MachineLearning 32. 机器学习之支持向量机递归特征消除的特征筛选 (mSVM-RFE)
MachineLearning 33. 机器学习之时间-事件预测与神经网络和Cox回归
MachineLearning 34. 机器学习之竞争风险生存分析的深度学习方法(DeepHit)
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
























 267
267

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








