
简 介
GTF(基因转移格式)和GFF(通用特征格式)是生物信息学程序常用的文件格式,用于表示和交换有关各种基因组特征的信息,如基因和转录本的位置和结构。GffRead是开源程序,提供了广泛和有效的解决方案来操作GTF或GFF格式的文件。虽然GffRead可以转换、排序、过滤、转换或聚类基因组功能。
许多生物医学研究应用采用管道系统地分析基因组中的基因内容。由于转录组学数据的爆炸式增长,这些管道通常涉及处理大量数据,因此需要有效的生物信息学工具来处理多个注释和序列文件,以加快基因组分析。这些工具通常交换和使用有关基因、转录本或其他基因组特征的信息,以制表符分隔的文本文件格式,通常称为GFF(通用特征格式)。这种格式描述了基因、转录本和其他功能的精确坐标和属性,如开始和停止密码子、编码序列等。GFF有许多版本,包括其最新版本GFF3和较旧的GTF(基因转移格式),有时也被称为GTF2。虽然较旧的GTF格式仅限于表示基因和转录本位置及其结构,但较新的GFF3格式可以以分层方式表示更多的基因组特征和注释。一些转录数据或基因组注释只能以其中一种格式从源中获得,但应用程序可能需要另一种格式作为输入。

GffRead实用程序可以自动识别和无缝地使用这些文件格式,从丰富的GFF3注释文件中提取和选择功能,执行之间的转换,甚至可以将文件从BED或FASTA等其他格式转换为其他格式。来自不同来源的注释数据可能对染色体和组群使用不同的命名约定。GffRead可以帮助映射这样的基因组序列名称,从而将注释从一种参考命名约定转换为另一种。基因预测程序和转录本(RNA-Seq)组装程序通常以GTF或GFF3格式输出结果,在这种情况下,通常需要评估预测/组装转录本的准确性。
软件包安装
cd /some/build/dir
git clone https://github.com/gpertea/gffread
cd gffread
make release数据读取
GFF全称为general feature format,主要是用来注释基因组。
1 havana mRNA 169853074 169888888 . - . ID=ENST00000367770;Parent=ENSG00000000457
1 havana three_prime_UTR 169853074 169853712 . - . ID=utr_3_9648;Parent=ENST00000367770
1 havana CDS 169853713 169853772 . - 0 ID=CDS:ENSP00000356744;Parent=ENST00000367770
1 havana CDS 169854270 169854964 . - 2 ID=CDS:ENSP00000356744rep_1;Parent=ENST00000367770
1 havana CDS 169855796 169855957 . - 2 ID=CDS:ENSP00000356744rep_2;Parent=ENST00000367770
1 havana CDS 169859041 169859212 . - 0 ID=CDS:ENSP00000356744rep_3;Parent=ENST00000367770
1 havana CDS 169862613 169862797 . - 2 ID=CDS:ENSP00000356744rep_4;Parent=ENST00000367770
1 havana CDS 169864369 169864508 . - 1 ID=CDS:ENSP00000356744rep_5;Parent=ENST00000367770
1 havana CDS 169866896 169866973 . - 1 ID=CDS:ENSP00000356744rep_6;Parent=ENST00000367770
1 havana CDS 169868928 169869039 . - 2 ID=CDS:ENSP00000356744rep_7;Parent=ENST00000367770
1 havana CDS 169870255 169870357 . - 0 ID=CDS:ENSP00000356744rep_8;Parent=ENST00000367770
1 havana CDS 169873696 169873752 . - 0 ID=CDS:ENSP00000356744rep_9;Parent=ENST00000367770
1 havana CDS 169875978 169876091 . - 0 ID=CDS:ENSP00000356744rep_10;Parent=ENST00000367770
1 havana CDS 169878634 169878819 . - 0 ID=CDS:ENSP00000356744rep_11;Parent=ENST00000367770
1 havana CDS 169888676 169888840 . - 0 ID=CDS:ENSP00000356744rep_12;Parent=ENST00000367770
1 havana exon 169853074 169853772 . - . ID=exon_87833;Parent=ENST00000367770
1 havana exon 169854270 169854964 . - . ID=exon_87834;Parent=ENST00000367770
1 havana exon 169855796 169855957 . - . ID=exon_87835;Parent=ENST00000367770
1 havana exon 169859041 169859212 . - . ID=exon_87836;Parent=ENST00000367770
1 havana exon 169862613 169862797 . - . ID=exon_87837;Parent=ENST00000367770
1 havana exon 169864369 169864508 . - . ID=exon_87838;Parent=ENST00000367770
1 havana exon 169866896 169866973 . - . ID=exon_87839;Parent=ENST00000367770
1 havana exon 169868928 169869039 . - . ID=exon_87840;Parent=ENST00000367770
1 havana exon 169870255 169870357 . - . ID=exon_87841;Parent=ENST00000367770
1 havana exon 169873696 169873752 . - . ID=exon_87842;Parent=ENST00000367770
1 havana exon 169875978 169876091 . - . ID=exon_87843;Parent=ENST00000367770
1 havana exon 169878634 169878819 . - . ID=exon_87844;Parent=ENST00000367770
1 havana exon 169888676 169888888 . - . ID=exon_87845;Parent=ENST00000367770GTF全称为gene transfer format,主要是用来对基因进行注释。
1 havana transcript 169853074 169888888 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457"
1 havana exon 169853074 169853772 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169854270 169854964 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169855796 169855957 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169859041 169859212 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169862613 169862797 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169864369 169864508 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169866896 169866973 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169868928 169869039 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169870255 169870357 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169873696 169873752 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169875978 169876091 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169878634 169878819 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana exon 169888676 169888888 . - . transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169853713 169853772 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169854270 169854964 . - 2 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169855796 169855957 . - 2 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169859041 169859212 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169862613 169862797 . - 2 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169864369 169864508 . - 1 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169866896 169866973 . - 1 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169868928 169869039 . - 2 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169870255 169870357 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169873696 169873752 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169875978 169876091 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169878634 169878819 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
1 havana CDS 169888676 169888840 . - 0 transcript_id "ENST00000367770"; gene_id "ENSG00000000457";
如果根据GFF或者GTF提取CDS,蛋白质和外显子序列,需要准备基因组序列文件
实例操作
安装之后可以测试一下:
gffread -h
gffread v0.12.7. Usage:
gffread [-g <genomic_seqs_fasta> | <dir>] [-s <seq_info.fsize>]
[-o <outfile>] [-t <trackname>] [-r [<strand>]<chr>:<start>-<end> [-R]]
[--jmatch <chr>:<start>-<end>] [--no-pseudo]
[-CTVNJMKQAFPGUBHZWTOLE] [-w <exons.fa>] [-x <cds.fa>] [-y <tr_cds.fa>]
[-j ][--ids <IDs.lst> | --nids <IDs.lst>] [--attrs <attr-list>] [-i <maxintron>]
[--stream] [--bed | --gtf | --tlf] [--table <attrlist>] [--sort-by <ref.lst>]
[<input_gff>]参数说明:
GffRead输入选项,输入GFF3数据(转录)的过滤。

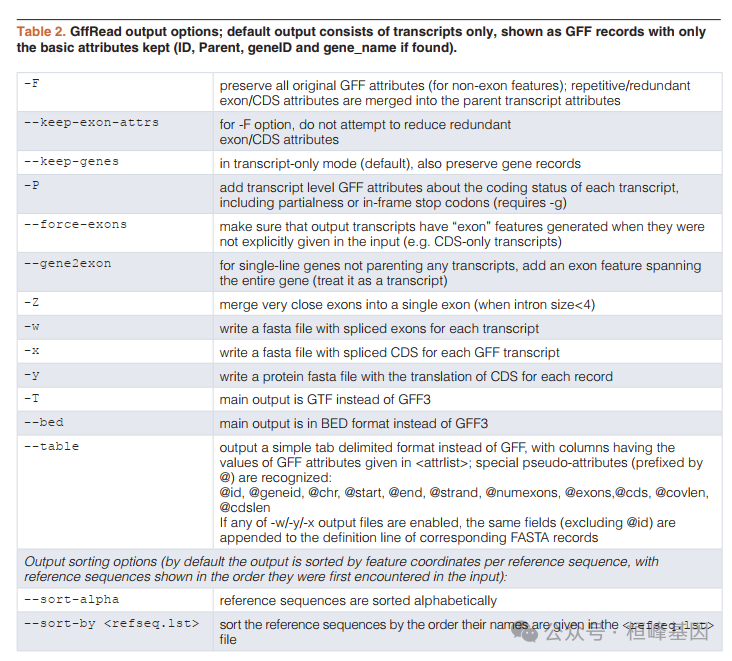
GffRead输出选项,默认输出仅由转录本组成,显示为GFF记录,仅保留基本属性(ID, Parent, geneID和gene_name)。
格式转换
快速读入过滤输出
这将显示输入文件 (注释)中找到的最简单的GFF3重新格式化的成绩单记录。GFF3或GTF2格式均可。-E选项 指示GffRead在解析输入文件时“暴露”(显示警告)遇到的任何潜在问题。
-E:显露(警告)重复的转录本ID和给定的GFF/GTF记录中的其他潜在问题。
gffread -E annotation.gff -o ann_simple.gffGFF转GTF3
为了获得相同转录本的GTF2版本,需要添加-T选项:
-T:主输出将是GTF,而不是默认的GFF3。
gffread annotation.gff -T -o annotation.gtf转录本的DNA序列生成FASTA文件
GffRead可以使用GFF文件中所有转录本的DNA序列生成FASTA文件。对于此 操作,还必须提供包含基因组序列的fasta文件。
-w 为每个转录本写一个包含剪接的外显子的fasta文件。
-g 全路径到具有基因组序列的多FASTA文件用于所有输入映射,或具有单FASTA文件的目录(每个基因组序列一个文件,文件名与序列名称匹配)。
gffread -w transcripts.fa -g genome.fa annotation.gffGFF3转GTF
gffread annotation.gff3 -T -o annotation.gtfGTF转GFF
gffread annotation.gtf -o annotation.gff3合并转录本
-M/--merge : 合并完全相同的或者存在包含关系的转录本
gffread -M annotation.gff3 -o annotation.gff3获取CDS序列
-x:输出CDS序列
gffread annotation.gff3 -g genome.fa -x cds.fa获取蛋白序列
-y:输出蛋白质序列
gffread annotation.gff3 -g genome.fa -y protein.fa获取转录本序列
-w:输出每个转录本的外显子序列
gffread annotation.gff3 -g genome.fa -w transcripts.faReference
Pertea G and Pertea M. GFF Utilities: GffRead and GffCompare [version 1; peer review: 3 approved]. F1000Research 2020, 9:304
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
























 2751
2751

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








