
简 介
生存分析(time-to-event analysis)广泛应用于经济金融、工程、医学等诸多领域。一个基本问题是理解协变量和生存时间(时间到事件)分布之间的关系。之前的大部分工作都是通过将生存时间视为随机过程的第一次命中时间来解决这个问题,假设潜在随机过程的特定形式,使用可用数据来学习协变量与模型参数之间的关系,然后推导协变量与第一次命中时间(风险)分布之间的关系。然而,以前的模型依赖于经常被违反的强参数假设。本文提出了一种非常不同的生存分析方法,DeepHit,它使用深度神经网络直接学习生存时间的分布。deepit没有对潜在的随机过程做任何假设,并允许协变量和风险之间的关系随时间变化的可能性。最重要的是,DeepHit 平稳地处理了相互竞争的风险;例如,存在多个可能的感兴趣事件的设置。与之前基于真实数据集和合成数据集的模型进行比较表明,与之前最先进的方法相比,DeepHit实现了巨大的、统计上显著的性能改进。

软件包安装
survivalmodels包使用reticulate从Python实现模型。为了使用这些模型,必须按照reticulate::py_install安装所需的Python包。Survivalmodels包含一个辅助函数,用于安装所需的pycox函数(如果还需要,则使用pytorch)。在运行此包中的任何模型之前,如果您尚未安装pycox,请运行。
install_pycox(pip = TRUE, install_torch = FALSE)然后再次安装survivalmodels:
remotes::install_github("RaphaelS1/survivalmodels")
install.packages("survivalmodels")数据读取
library(survival)
library(sampling)
dim(lung)
## [1] 228 10
lung = na.omit(lung)
table(lung$status)
##
## 1 2
## 47 120
set.seed(123)
# 每层抽取70%的数据
train_id <- strata(lung, "status", size = rev(round(table(lung$status) * 0.7)))$ID_unit
# 训练数据
trainData <- lung[train_id, ]
# 测试数据
testData <- lung[-train_id, ]实例操作
构建模型
DeepHit:一种竞争风险生存分析的深度学习方法用于计算的计算图的图示DeepHit的训练损失。

### 构建模型
library(survivalmodels)
set_seed(123)
fit = deephit(data = trainData, time_variable = "time", status_variable = "status")
# common parameters
fit = coxtime(data = trainData, frac = 0.3, activation = "relu", num_nodes = c(4L,
8L, 4L, 2L), dropout = 0.1, early_stopping = TRUE, epochs = 100L, batch_size = 32L)
fit
##
## Cox-Time Neural Network
##
## Call:
## coxtime(data = trainData, frac = 0.3, activation = "relu", num_nodes = c(4L, 8L, 4L, 2L), dropout = 0.1, early_stopping = TRUE, batch_size = 32L, epochs = 100L)
##
## Response:
## Surv(time, status)
## Features:
## {inst, age, sex, ph.ecog, ph.karno, pat.karno, meal.cal, wt.loss}验证
预测predict()参数中type有三种选择:"survival", "risk", "all", 可以获得预测生存矩阵和相对风险:
pred <- predict(fit, testData, type = "survival")
str(pred)
## num [1:50, 1:76] 0.977 0.977 0.976 0.976 0.977 ...
## - attr(*, "dimnames")=List of 2
## ..$ : chr [1:50] "0 " "1 " "2 " "3 " ...
## ..$ : chr [1:76] "11" "15" "26" "53" ...一致性
survivalmodels包含了一致性分析的函数cindex(),跟survival包里面的survival::concordance()使用非常相似。
# predict survival matrix and relative risks
pred <- predict(fit, testData, type = "all")
p <- predict(fit, type = "risk", newdata = testData)
cindex(risk = p, truth = testData[, "time"])
## [1] 0.5457516生存分析
根据风险值我们可以将患者分为高低风险组,然后绘制生存曲线:
library(survminer)
testData$risk = p
group = ifelse(testData$risk > median(testData$risk), "High", "Low")
f <- survfit(Surv(testData$time, testData$status) ~ group)
f
## Call: survfit(formula = Surv(testData$time, testData$status) ~ group)
##
## n events median 0.95LCL 0.95UCL
## group=High 25 17 285 245 NA
## group=Low 25 19 363 181 524
ggsurvplot(f, data = testData, surv.median.line = "hv", legend.title = "Risk Group",
legend.labs = c("Low Risk", "High Risk"), pval = TRUE, ggtheme = theme_bw())
绘制ROC曲线
由于我们所作的模型根时间密切相关因此我们选择timeROC,可以快速的技术出来不同时期的ROC,进一步作图:
library(timeROC)
res <- timeROC(T = testData$time, delta = testData$status, marker = testData$risk,
cause = 1, weighting = "marginal", times = c(1 * 365, 2 * 365), ROC = TRUE, iid = TRUE)
res$AUC_1
## t=365 t=730
## 0.7037037 0.6200000
options(digits = 2)
confint(res, level = 0.95)$CI_AUC
## NULL
plot(res, time = 1 * 365, col = "red", title = FALSE, lwd = 2)
plot(res, time = 2 * 365, add = TRUE, col = "blue", lwd = 2)
legend("bottomright", c(paste("1 Years ", round(res$AUC_1[1], 2), sep = ""), paste("2 Years ",
round(res$AUC_1[2], 2), sep = "")), col = c("red", "blue", "green"), lty = 1,
lwd = 2)
不同时间节点的AUC曲线及其置信区间
再分析不同时间节点的AUC曲线及其置信区间,由于数据量非常小,此图并不明显。
plotAUCcurve(res, conf.int = TRUE, col = "red")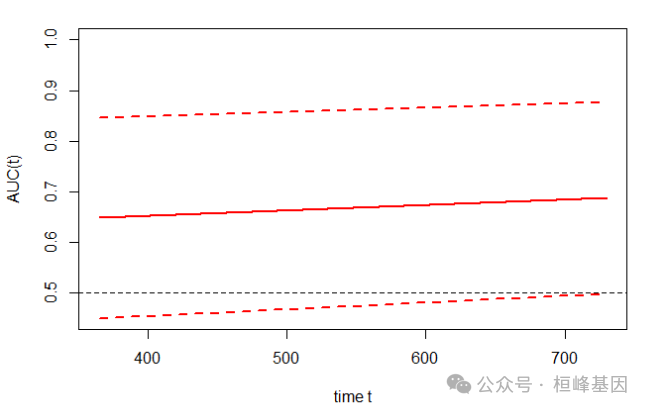
Reference
C. Lee, W. R. Zame, J. Yoon, M. van der Schaar, "DeepHit: A Deep Learning Approach to Survival Analysis with Competing Risks," AAAI Conference on Artificial Intelligence (AAAI), 2018
T.A. Gerds et al, "Estimating a Time-Dependent Concordance Index for Survival Prediction Models with Covariate Dependent Censoring," Stat Med., 2013
基于机器学习构建临床预测模型
MachineLearning 2. 因子分析(Factor Analysis)
MachineLearning 3. 聚类分析(Cluster Analysis)
MachineLearning 4. 癌症诊断方法之 K-邻近算法(KNN)
MachineLearning 5. 癌症诊断和分子分型方法之支持向量机(SVM)
MachineLearning 6. 癌症诊断机器学习之分类树(Classification Trees)
MachineLearning 7. 癌症诊断机器学习之回归树(Regression Trees)
MachineLearning 8. 癌症诊断机器学习之随机森林(Random Forest)
MachineLearning 9. 癌症诊断机器学习之梯度提升算法(Gradient Boosting)
MachineLearning 10. 癌症诊断机器学习之神经网络(Neural network)
MachineLearning 11. 机器学习之随机森林生存分析(randomForestSRC)
MachineLearning 12. 机器学习之降维方法t-SNE及可视化(Rtsne)
MachineLearning 13. 机器学习之降维方法UMAP及可视化 (umap)
MachineLearning 14. 机器学习之集成分类器(AdaBoost)
MachineLearning 15. 机器学习之集成分类器(LogitBoost)
MachineLearning 16. 机器学习之梯度提升机(GBM)
MachineLearning 17. 机器学习之围绕中心点划分算法(PAM)
MachineLearning 18. 机器学习之贝叶斯分析类器(Naive Bayes)
MachineLearning 19. 机器学习之神经网络分类器(NNET)
MachineLearning 20. 机器学习之袋装分类回归树(Bagged CART)
MachineLearning 21. 机器学习之临床医学上的生存分析(xgboost)
MachineLearning 22. 机器学习之有监督主成分分析筛选基因(SuperPC)
MachineLearning 23. 机器学习之岭回归预测基因型和表型(Ridge)
MachineLearning 24. 机器学习之似然增强Cox比例风险模型筛选变量及预后估计(CoxBoost)
MachineLearning 25. 机器学习之支持向量机应用于生存分析(survivalsvm)
MachineLearning 26. 机器学习之弹性网络算法应用于生存分析(Enet)
MachineLearning 27. 机器学习之逐步Cox回归筛选变量(StepCox)
MachineLearning 28. 机器学习之偏最小二乘回归应用于生存分析(plsRcox)
MachineLearning 29. 机器学习之嵌套交叉验证(Nested CV)
MachineLearning 30. 机器学习之特征选择森林之神(Boruta)
MachineLearning 31. 机器学习之基于RNA-seq的基因特征筛选 (GeneSelectR)
MachineLearning 32. 机器学习之支持向量机递归特征消除的特征筛选 (mSVM-RFE)
桓峰基因,铸造成功的您!
未来桓峰基因公众号将不间断的推出单细胞系列生信分析教程,
敬请期待!!
桓峰基因官网正式上线,请大家多多关注,还有很多不足之处,大家多多指正!http://www.kyohogene.com/
桓峰基因和投必得合作,文章润色优惠85折,需要文章润色的老师可以直接到网站输入领取桓峰基因专属优惠券码:KYOHOGENE,然后上传,付款时选择桓峰基因优惠券即可享受85折优惠哦!https://www.topeditsci.com/
























 6760
6760

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








