安装和更新
近期断断续续对linkET做了一些更新,主体是从方便用户使用,增强灵活性的角度出发的,至于我想在新版本中添加的功能,暂时还未完成,而且预计新功能完成可能得到端午节前后,所以这个版本(0.0.3)是未来一段时间比较稳定的版本,若是想尝新,不妨更新一下试试。
## install.packages("devtools")
devtools::install_github("Hy4m/linkET", force = TRUE)
library(linkET)correlate()函数
之前的版本(包括最早的ggcor),correlate()函数都仅仅是封装cor()和cor.test()函数,来作为默认的相关系数矩阵计算的函数,然后提供了统一的接口来把WGCNA等其他包计算的结果导入到linkET中。这一次更新,我尝试把其他包的函数(如psych::corr.test())作为计算引擎,完整的封装进correlate()函数,这样用户就不用去深究各种不同的包的函数差异。下面的例子给了一些示例,可以尝试运行查看结果。
## 默认计算方式,等价于上一个版本的correlate()
correlate(mtcars)
## 使用psych包作为计算引擎
correlate(mtcars, engine = "psych")
## 使用Hmisc包作为计算引擎
correlate(mtcars, engine = "Hmisc")
## 使用picante包作为计算引擎
correlate(mtcars, engine = "picante")
## 使用WGCNA包作为计算引擎
correlate(mtcars, engine = "WGCNA")当然,correlate()函数还增加了一个group参数,这个参数可以用来指定原始数据的行分组信息,例如比如著名的iris数据集中,若我们想根据物种la分别计算相关系数矩阵,我们就可以把group参数设置为Species这个向量。
# head(iris)
correlate(iris[-5], group = iris$Species)
直接输出分组计算的相关系数矩阵的结果,只能看到一点描述性信息,建议使用下面的代码把图画出来:
correlate(iris[-5], group = iris$Species) %>%
qcorrplot() + geom_square()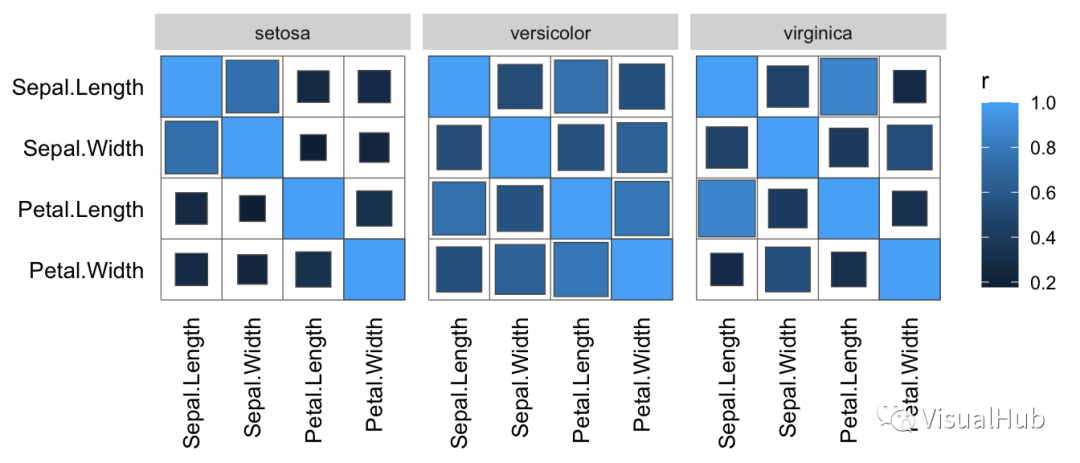
改变默认样式
在linkET的开发过程中,我一直保持克制,希望能最大程度保持原汁原味的ggplot2的感觉,但是经不住问,我还是提供了一个set_corrplot_style()函数,每次打开R会话框后运行下这个函数,就能保证之后的所有的qcorrplot()函数画的图都改成了你想要的样式。
set_corrplot_style()
qcorrplot(mtcars) + geom_square()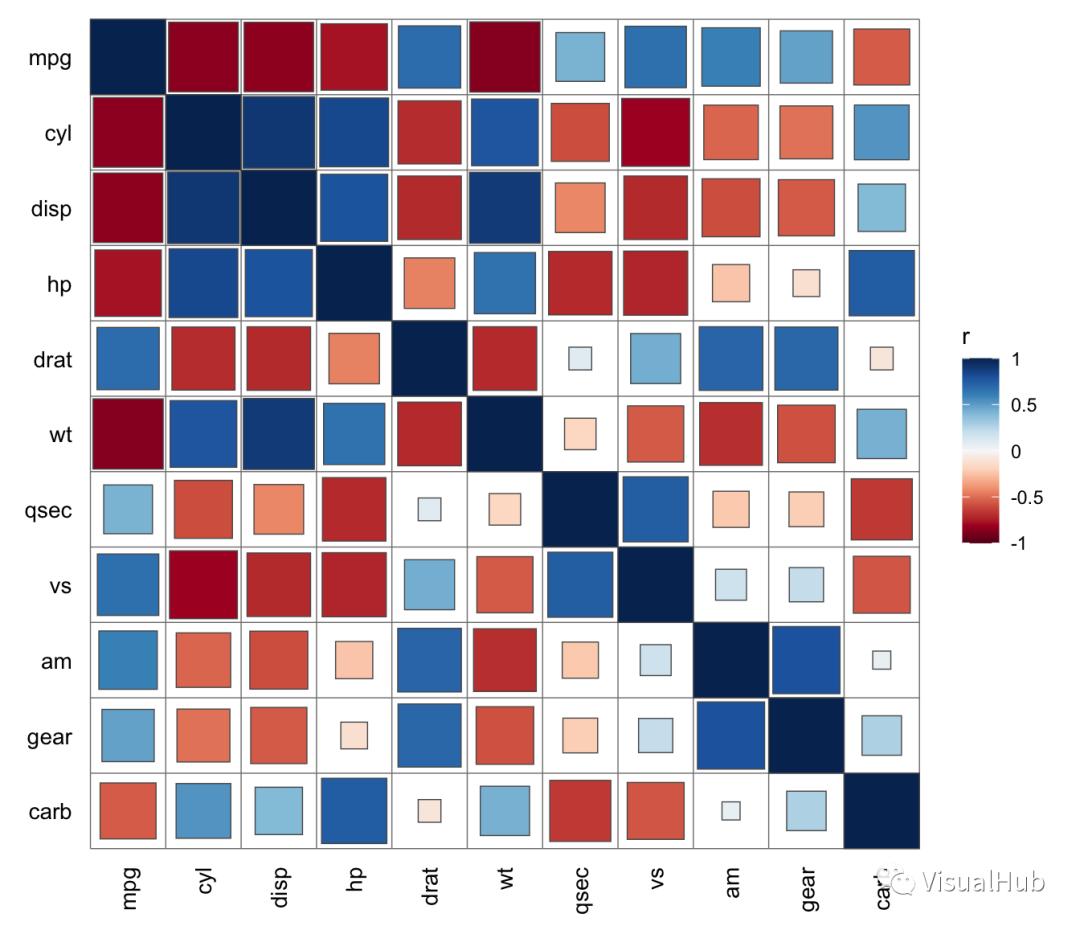
也可以使用你自己的调色板,比如改成红白蓝的配色:
set_corrplot_style(colours = c("red", "white", "blue"))
qcorrplot(mtcars) + geom_square()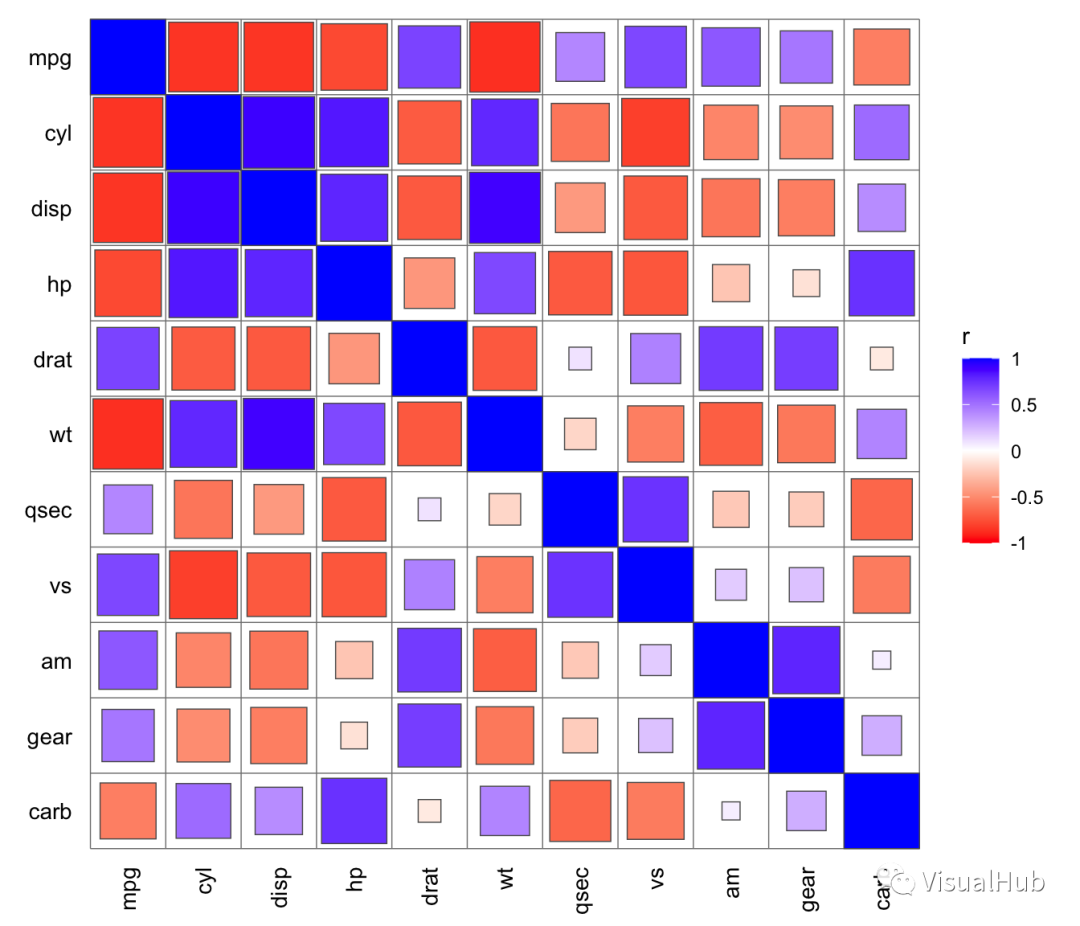
最后,要是你嫌弃默认使用的scale_fill_gradientn()函数不能满足你的需求,你完全可以提供的scale函数,比如:
set_corrplot_style(scale = ggplot2::scale_fill_viridis_c())
qcorrplot(mtcars) + geom_square()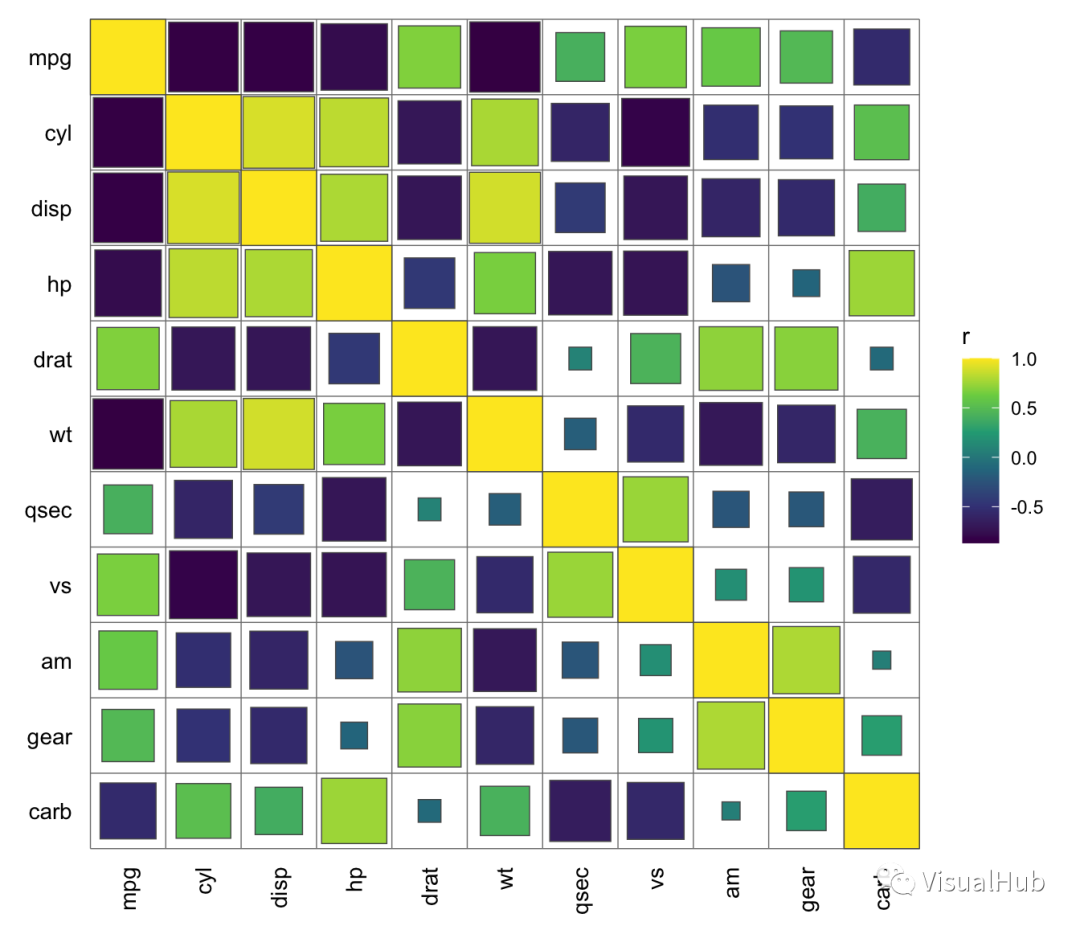
用完了记得还原一下,不然之后的图还会按照你设置的样式画图:
set_default_style()更丝滑的geom_couple()函数
我们先看看:
library(dplyr)
library(ggplot2)
data("varechem", package = "vegan")
data("varespec", package = "vegan")
mantel <- mantel_test(varespec, varechem,
spec_select = list(Spec01 = 1:7,
Spec02 = 8:18,
Spec03 = 19:37,
Spec04 = 38:44)) %>%
mutate(rd = cut(r, breaks = c(-Inf, 0.2, 0.4, Inf),
labels = c("< 0.2", "0.2 - 0.4", ">= 0.4")),
pd = cut(p, breaks = c(-Inf, 0.01, 0.05, Inf),
labels = c("< 0.01", "0.01 - 0.05", ">= 0.05")))
## 现学现用
set_corrplot_style()
qcorrplot(correlate(varechem), type = "lower", diag = FALSE) +
geom_square() +
geom_couple(aes(colour = pd, size = rd),
data = mantel,
curvature = nice_curvature()) +
scale_size_manual(values = c(0.5, 1, 2)) +
scale_colour_manual(values = color_pal(3)) +
guides(size = guide_legend(title = "Mantel's r",
override.aes = list(colour = "grey35"),
order = 2),
colour = guide_legend(title = "Mantel's p",
override.aes = list(size = 3),
order = 1),
fill = guide_colorbar(title = "Pearson's r", order = 3))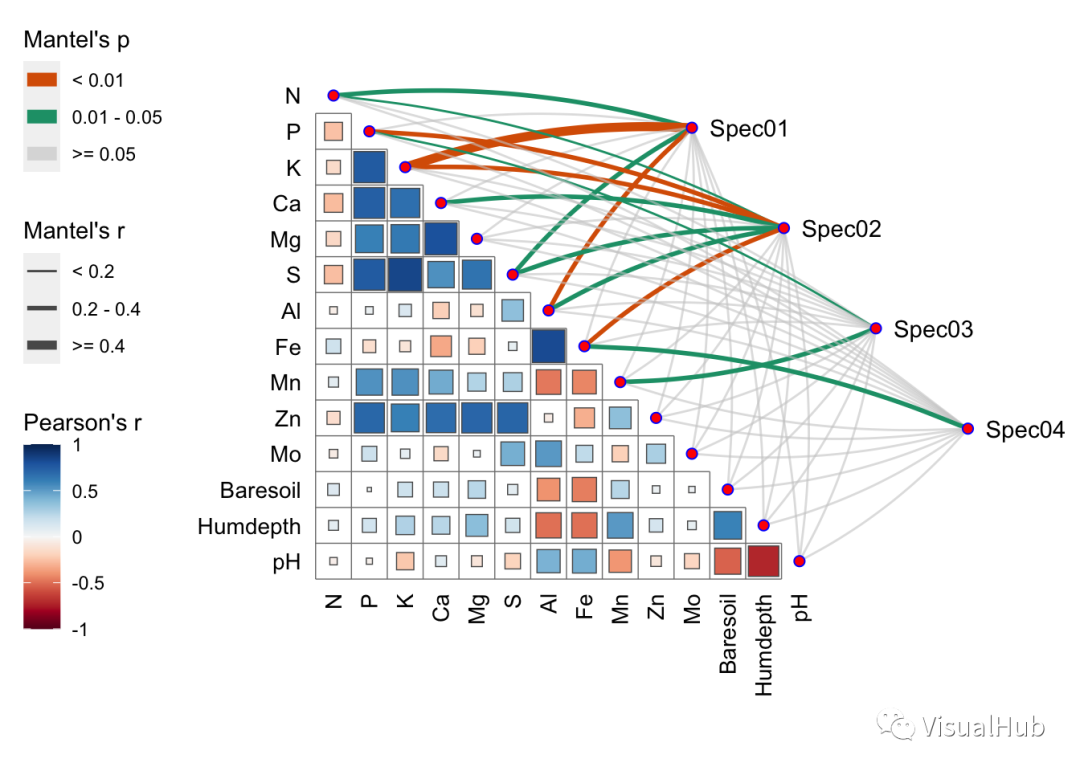
现在想把Spec的几个点的位置微调一下,在之前的三期文章中我提到了可以使用aes(x = .x + 1)之类的方式来调整,但是这种方法每次都是所有的点做相同的调整,在这个版本中我新增了offset_x和offset_y两个参数,这两个参数能对指定的点做调整,没有指定的保持在原位不动。
qcorrplot(correlate(varechem), type = "lower", diag = FALSE) +
geom_square() +
geom_couple(aes(colour = pd, size = rd),
data = mantel,
curvature = nice_curvature(),
offset_x = list(Spec02 = 3, Spec03 = 3),
offset_y = list(Spec01 = 0.5, Spec02 = 1)) +
scale_size_manual(values = c(0.5, 1, 2)) +
scale_colour_manual(values = color_pal(3)) +
guides(size = guide_legend(title = "Mantel's r",
override.aes = list(colour = "grey35"),
order = 2),
colour = guide_legend(title = "Mantel's p",
override.aes = list(size = 3),
order = 1),
fill = guide_colorbar(title = "Pearson's r", order = 3))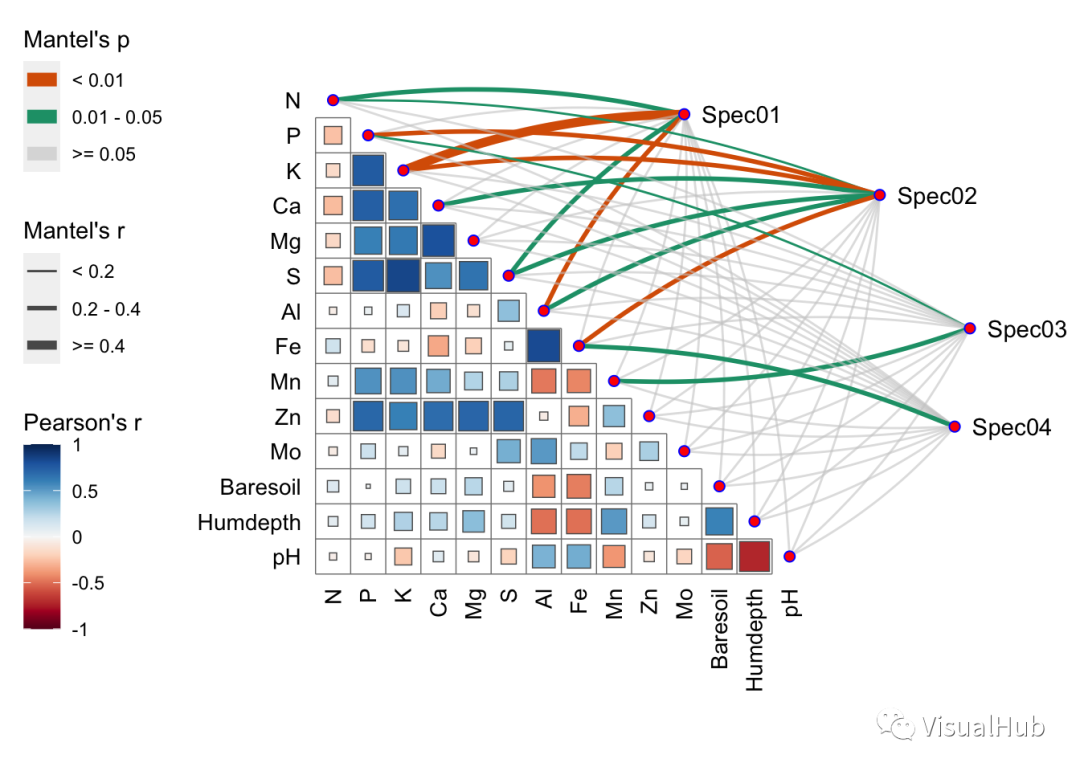
小结
linkET还会继续开发,功能也会越来越完整,希望所有人都能切换到linkET上来,忘了那个ggcor吧。
猜你喜欢
iMeta简介 高引文章 高颜值绘图imageGP 网络分析iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读





















 3万+
3万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








